Pcr secuenciacion
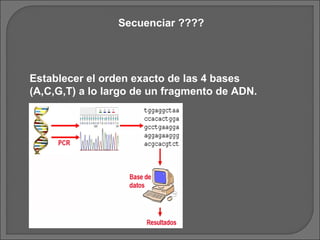
- 1. Secuenciar ???? Establecer el orden exacto de las 4 bases (A,C,G,T) a lo largo de un fragmento de ADN.
- 2. Métodos básicos de secuenciación Método químico de Maxam y Gilbert Maxam, A.M. and Gilbert,W. (1977) A new method for sequencing DNA. Proc. Natl. Acad. Sci. USA, 74, 560-564. Método enzimático de Sanger Sanger, F., Micklen, S.,and Coulson, A.R. (1977) DNA sequencing and chain terminating inhibitors. Proc. Natl. Acad. Sci. USA, 74, 5463-5467
- 3. Frederick Sanger y Walter Gilbert compartieron, en 1980, el premio Nobel de química por haber desarrollado independientemente métodos de secuenciación del ADN . Sanger ganó anteriormente el premio Nobel de 1959 por la secuenciación de la proteína insulina. F. Sanger W. Gilbert
- 4. Método químico de Maxam y Gilbert El fragmento de ADN se marca en sus extremos con Ɣ 32P ó Ɣ 32S dATP por acción de la Polinucleótido Quinasa. Las moléculas marcadas se rompen con reacciones químicas específicas para cada una de las cuatro bases. Los productos luego se resuelven en función de su tamaño en geles de poliacrilamida.
- 5. Extremo marcado radioactivamente 5' o 3‘. El tratamiento químico rompe la unión glicosídica entre la ribosa y el nucleótido. Reacciones químicas específicas –G : DMS, piperidina –A + G: DMS, ácido fórmico,piperidina – C+T: Hidracina, piperidina –C : Hidracina en 1.5M NaCl, piperidina(rompe la unión fosfodiester) Electroforesis en gel + Autoradiografía
- 8. USOS Secuenciar oligonucleótidos sintéticos Analizar modificaciones en la estructura del adn (ejemplo: metilación de bases) Estudiar la estructura secundaria del ADN o su interacción con proteínas (ejemplo: experimentos de protección química).
- 9. Método de secuenciación de Sanger Desarrollado en 1977 por Frederick Sanger. Método Bioquímico (usa una polimerasa). Basado en la incorporación de Dideoxinucleótidos (ddNTPs) que detienen el crecimiento de la cadena de ADN.
- 11. Los dideoxinucleótidos no tienen OH en carbono 2' ni en 3' de la desoxirribosa, por lo que no pueden formar enlaces fosfodiéster.
- 12. Durante la secuenciación se llevan a cabo cuatro reacciones de síntesis separadas incluyendo en cada una de ellas pequeñas cantidades de los dideoxinucleótidos (ddNTP: ddGTP; ddATP, ddCTP, ddTTP)
- 13. se requiere: + Primer Marcado + ADN polimerasa + 4 dNTPs + ddATP Al incorporarse un dideoxinucleótido finaliza la copia del templado y se generan fragmentos de distintos tamaños. Los ddNTP compiten con los dNTP para ser incorporados. produciendo una mezcla de cadenas de distintas longitudes.
- 14. La sustitución de uno de los dNTPs por el mismo nucleótido marcado radiactivamente permite la visualización de las bandas de distinta longitud por autoradiografía.
- 15. En el gel la separación de las distintas bandas se produce en función de su tamaño y la secuencia puede determinarse leyendo las bandas de los cuatro carriles.
- 16. Los protocolos de secuenciación automática: Son una alternativa al método de Sanger. Se utilizan compuestos fluorescentes para “marcar” al “primer” ó a los terminadores. Los productos de la reacción se detectan directamente durante la electroforesis. Los fluoróforos son excitados por la luz de un láser y luego el equipo detecta la fluorescencia emitida. La reacción se realiza en un ciclador
- 17. Las reacciones de secuenciación automática pueden emplear: Terminadores fluorescentes Primers fluorescentes
- 18. Secuenciación empleando terminadores fluorescentes Se realiza una única reacción de secuencia en presencia de los cuatro ddNTPs, cada uno de ellos marcados con una sonda fluorescente distinta.
- 19. Las sondas empleadas son: A ---> JOE. Emisión: verde. Derivado de fluoresceína C ---> FAM. Emisión: azul. Derivado de fluoresceína G --> TAMRA. Emisión: amarillo. Derivado de rodamina T --> ROX. Emisión: rojo. Derivado de rodamina
- 20. Secuenciación empleando primers fluorescentes Se realizan cuatro reacciones de secuencia distintas en cada una de las cuales se añade el “primer” marcado en 5´ con una sonda fluorescente distinta, junto con la polimerasa, los dNTPs y el ddNTP correspondiente. En la secuenciación cíclica solo se utiliza UNO de los dos primers a la vez. Si utilizáramos al mismo tiempo ambos “primers”, como en un PCR común, resultarían dos secuencias solapadas ( la del templado y la de su cadena complementaria).
- 26. Aplicaciones de la secuenciación de ADN Secuenciación genómica Evaluación del clonado en vectores Diagnóstico de enfermedades : bacterianas virales protozoos hongos cáncer genéticas Detección de polimorfismos (single nucleotide polymorphism) Forense ADNs fósiles