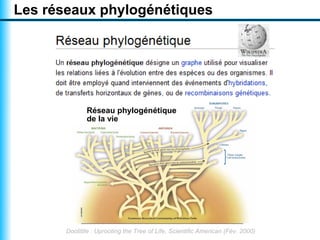
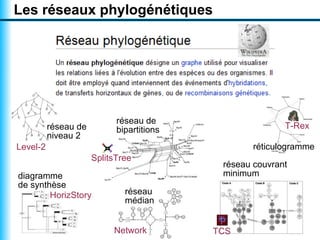
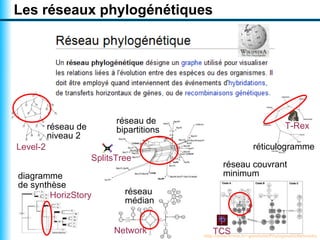
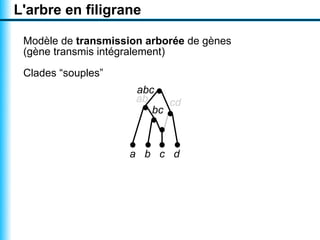
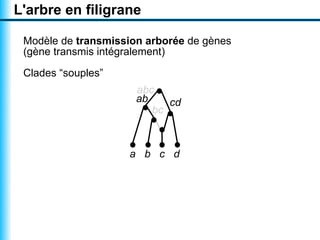
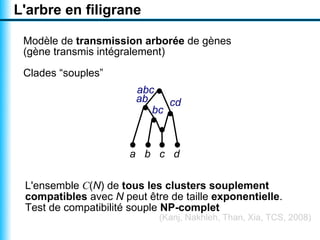
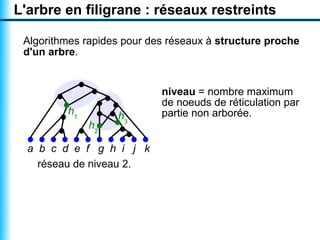
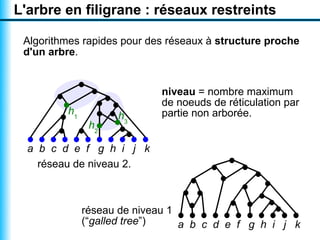
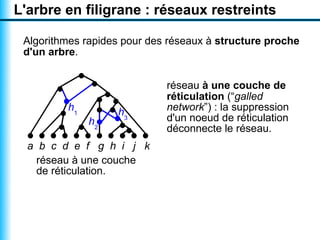
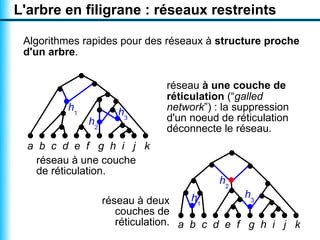
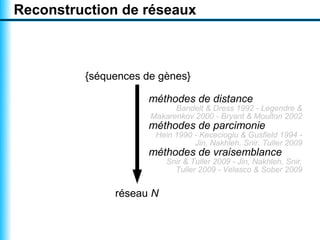
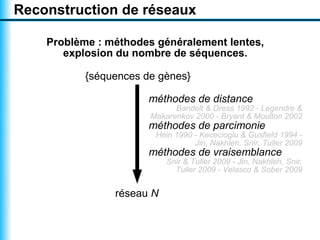
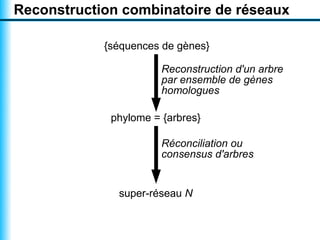
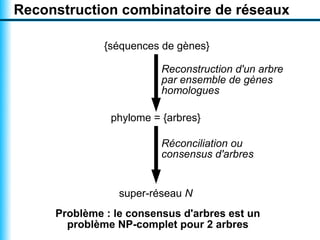
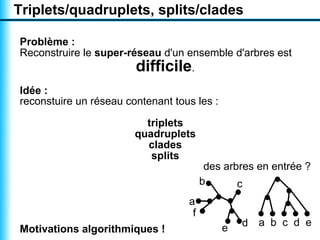
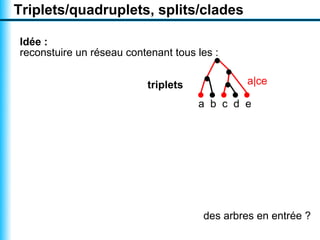
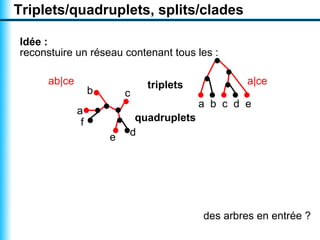
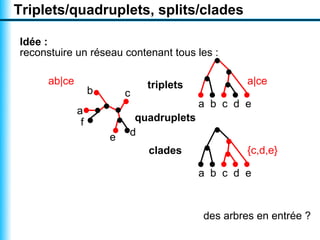
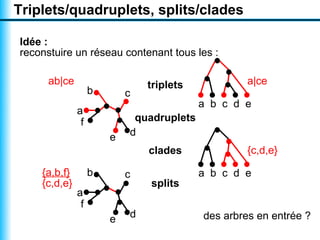
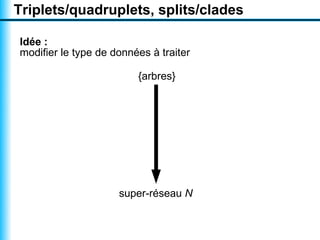
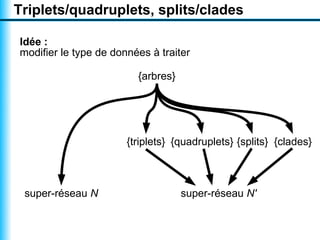
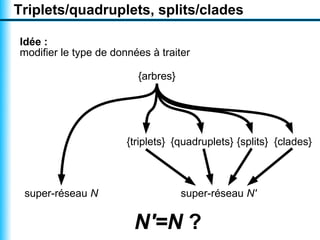
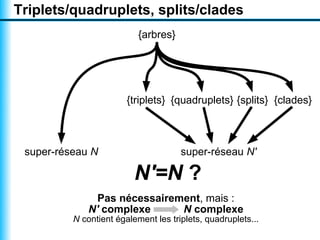
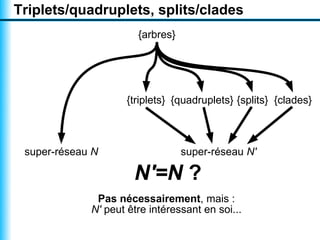
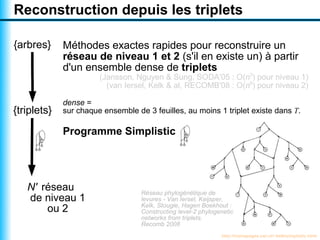
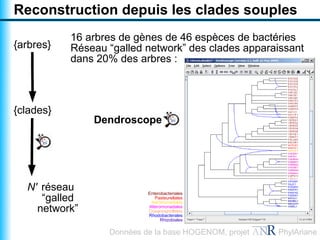
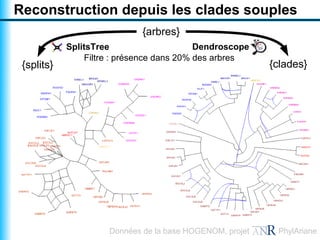
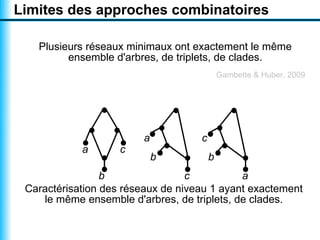
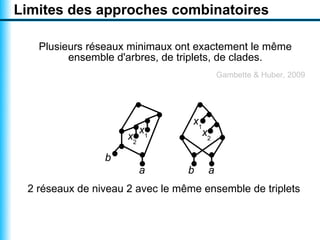
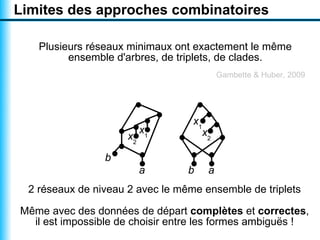
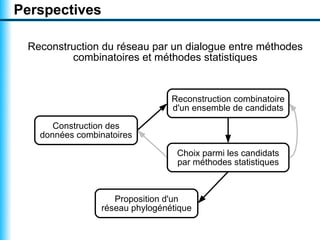
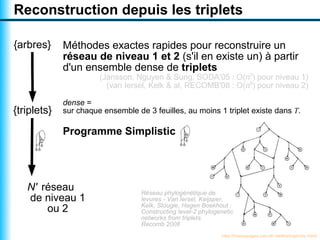
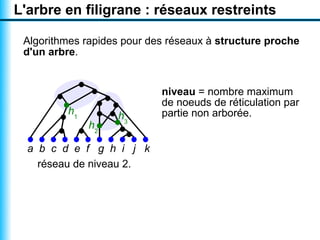
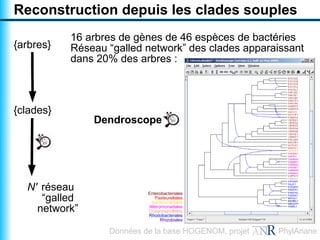
Le document traite de la reconstruction combinatoire de réseaux phylogénétiques, en abordant les concepts fondamentaux tels que les réseaux phylogénétiques, les arbres en filigrane et divers méthodes de reconstruction. Il souligne les limites des approches combinatoires et propose des perspectives pour l'intégration de méthodes combinatoires et statistiques. Des algorithmes adaptés et des recherches antérieures sont également mentionnés pour illustrer les défis posés par la reconstruction de réseaux à partir de données complexes.