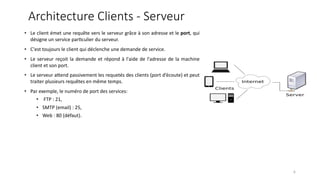
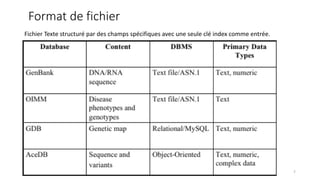
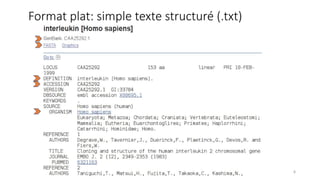
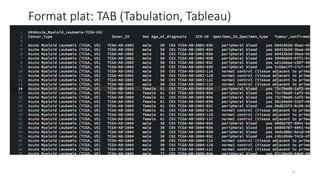
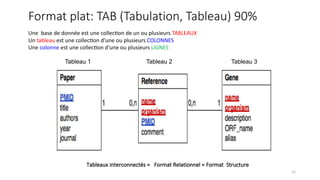
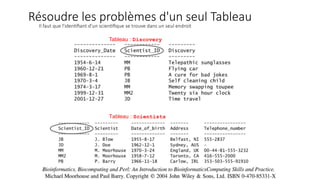
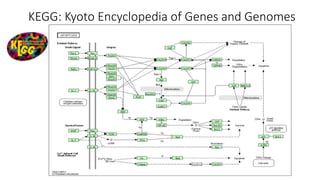
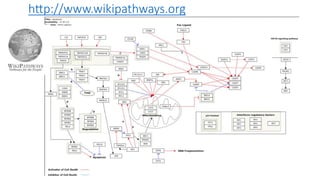
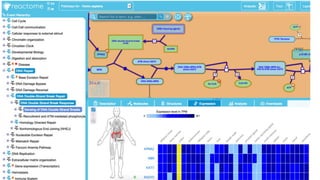
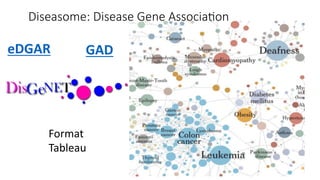
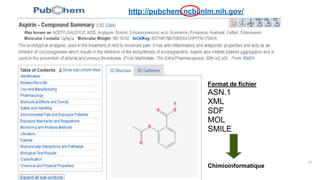
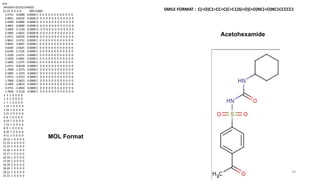
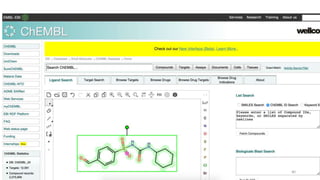
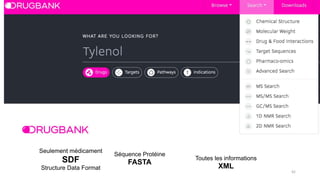
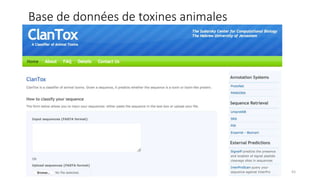
Le document présente l'évolution des bases de données en bioinformatique depuis les années 80, en soulignant leur rôle dans la gestion des données biologiques et médicales. Il décrit les défis de stockage et de consultation des données avant l'avènement d'Internet, et l'importance des architectures client-serveur pour faciliter l'accès à l'information. Enfin, il aborde les différents formats de fichiers et bases de données utilisés pour représenter les interactions biologiques et chimiques.