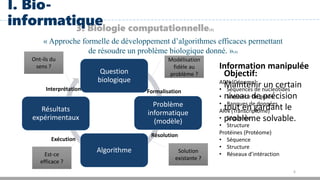
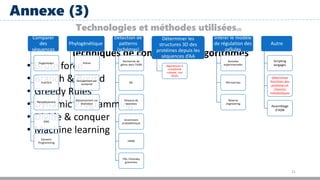
Le document présente une introduction à la bio-informatique, un domaine interdisciplinaire combinant biologie, informatique et mathématiques pour analyser les séquences biologiques. Il aborde l'historique, l'état des lieux, les défis de recherche et les méthodologies utilisées dans ce secteur en pleine expansion. Enfin, il souligne l'importance d'une approche transdisciplinaire pour faire face aux problématiques actuelles et futures de la bio-informatique.