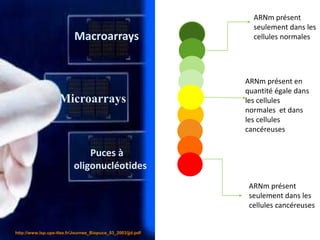
Les puces à ADN, technologie multidisciplinaire, permettent de mesurer l'expression de milliers de gènes simultanément, avec des applications croissantes dans divers domaines comme la pharmacologie et l'environnement. Le processus de fabrication consiste en plusieurs étapes, notamment l'hybridation et l'analyse des données, permettant de déterminer les niveaux d'expression génique à partir d'échantillons d'ADN. Bien que leur utilisation présente des avantages significatifs, comme l'étude simultanée de nombreux gènes, elle nécessite également un équipement coûteux et une analyse informatique complexe.