Cette thèse de doctorat présente une exploration des modules conservés de transformations chimiques dans le métabolisme, en utilisant des approches bioinformatiques. L'auteur, Maria Sorokina, exprime sa gratitude envers ceux qui ont contribué à son parcours académique et personnel durant ses trois années de recherche à l'Université d'Évry Val d'Essonne. Le document aborde notamment l'évolution du métabolisme, la modélisation des réseaux métaboliques et les méthodes de recherche de nouvelles activités enzymatiques.












![7
Introduction
Le métabolisme est un des aspects les plus basiques de la vie. Il s'agit d'un système complexe, qui
implique des enzymes, la régulation de leur expression et leurs interactions, ayant pour objectif de
produire, via la catalyse de réactions biochimiques, toutes les substances chimiques (métabolites)
nécessaires au maintien de la vie dans les cellules. L’avènement de la biochimie expérimentale
dans les années 1950 a permis de découvrir la grande partie des activités enzymatiques connues
actuellement. De nos jours, la découverte de nouvelles activités enzymatiques a beaucoup ralenti.
De plus, environ 30% des activités enzymatiques connues, au moment de la rédaction de cette
thèse, sont orphelines de séquence [1–8], c’est à dire que les enzymes qui les catalysent sont
inconnues. Aussi, l’expérimentation in vivo démontre que les organismes, selon les conditions,
peuvent adopter des comportements qui ne peuvent pas être expliqués par les connaissances
actuelles sur le métabolisme, ce qui suggère que beaucoup d’activités enzymatiques sont encore à
découvrir. Dans les années 2000, l’arrivée des nouvelles technologies de séquençage et le
séquençage des génomes complets ont permis d’obtenir un nombre colossal de séquences d’acide
désoxyribonucléique (ADN). Cependant, malgré cette quantité de données brutes, il est très
difficile de découvrir de nouvelles activités enzymatiques à partir des séquences seules, et
parallèlement, une très grande partie (plus d'un tiers chez Escherichia coli K-12 MG1655, un des
organismes les plus étudiés et les mieux connus [9, 10]) demeurent de fonction inconnue, sans
parler des nombreuses annotations erronées dans les banques de séquences [11]. Sans connaître
l’enzyme qui catalyse une réaction d’intérêt, il est compliqué de maîtriser et de reproduire cette
réaction au besoin, et, sans connaître la fonction d’une protéine, on peut passer à côté d’une
activité enzymatique nouvelle qui peut être intéressante. Les conséquences de cette double lacune
dans les connaissances fondamentales sur le fonctionnement du vivant sont nombreuses et
touchent, également, beaucoup de domaines appliqués dont l’ingénierie métabolique, la
pharmacologie, la médecine, l’industrie agro-alimentaire ou encore l’écologie.
Deux axes principaux de recherche pour résoudre ces lacunes sur la connaissance du
métabolisme peuvent être identifiés en observant la littérature. Le premier axe est sur le
développement des techniques autour de l'annotation fonctionnelle des protéines, c'est à dire la
prédiction de la fonction d’une protéine à partir de sa séquence et de données connexes. Le](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-13-2048.jpg)
![8
deuxième axe de recherche consiste à résoudre les "trous" dans le métabolisme qui
correspondent à des réactions catalysées dont les enzymes sont inconnues (enzymes orphelines
de séquence) ou à des réactions inconnues, à découvrir via l'exploration des réseaux
métaboliques, qui permettent de produire des métabolites d'intérêt.
L'étude des génomes a commencé dans les années 1990 avec en 1995 le premier séquençage d'un
organisme procaryote, Haemophilus influenzae Rd KW20. Vingt ans plus tard, près de cinquante
mille génomes complets (981 archées, 41001 bactériens et 6481 eucaryotes) sont disponibles dans
les bases de données (source Genomes Online, https://gold.jgi-psf.org), et le séquençage de
beaucoup de génomes et métagénomes est en cours de route. L'annotation fonctionnelle est le
processus d'assignation d'une fonctionnalité moléculaire et/ou biochimique à une séquence
d’ADN et/ou polypeptidique. D'après une étude [12], une fonction peut être potentiellement
associée par homologie pour environ 70% des gènes d'un organisme. Pour cela, les outils de
recherche de similarité entre séquences comme BLAST, FASTA et HMMER [13–17] sont
communément utilisés. Les 30% restants de gènes sont soit homologues à un gène de fonction
inconnue, soit ne ressemblent à aucune autre séquence précédemment élucidée. Ces pourcentages
sont très variables suivant les organismes étudiés et dépendent de leur proximité phylogénétique
avec des organismes expérimentalement étudiés. Dans la base de données UniProt [18], les
protéines de fonction inconnue sont référencées avec des termes comme "hypothetical",
"uncharacterized", "unknown" ou encore "putative" et représentent plus de 42% des 50 millions
de protéines publiées.
Plusieurs méthodes ont été développées pour essayer d'assigner une fonction aux nouvelles
séquences ou d'améliorer la qualité de l'annotation des séquences déjà connues. Parmi ces
méthodes, on trouve de la prédiction de fonction à partir du contenu en domaines structuraux et
fonctionnels d’une protéine [19], en s'aidant des informations sur la structure des protéines [20],
en créant des systèmes à bases de règles [21] ou encore en créant un réseau mondial
d’annotateurs experts [22]. La curation humaine a aussi une place importante dans les projets
d’annotation, notamment grâce aux efforts de SwissProt [23]. Ce genre d'études et de méthodes a
apporté énormément à l’amélioration de la qualité des annotations des gènes et des protéines
qu'ils encodent. Cependant, elles ne permettent pas de trouver la fonction d’un gène si aucune
caractérisation expérimentale directe ou indirecte n’est disponible (on parle alors de gènes
orphelins de fonction [24]).](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-14-2048.jpg)
![9
Parallèlement aux efforts liés à l'annotation fonctionnelle des gènes et des protéines, des
approches, plus orientées sur l’analyse de réseaux, sont développées pour en découvrir plus sur le
métabolisme du point de vue biochimique, notamment en résolvant le problème des trous
("gaps" en anglais) dans le métabolisme et celui d’activités enzymatiques inconnues. L’approche
utilisée pour appréhender ce problème est d’étudier la structure des réseaux métaboliques,
notamment en identifiant une logique dans les enchaînements de transformations chimiques de
métabolites, que l’on appelle communément "voies métaboliques".
En 2005, Lacroix et al. [25] mettent en place une méthode de recherche de motifs fonctionnels
dans les réseaux métaboliques et introduisent le terme de "motif réactionnel". Pour la première
fois, ce terme n’est pas basé uniquement sur les caractéristiques topologiques du réseau, mais
aussi sur la nature fonctionnelle des composantes de ce motif. Malgré des preuves exactes du bon
fonctionnement de la méthode, elle se limite à la recherche des motifs fréquents dans les réseaux
métaboliques organisme-centrés, et ne permet pas la découverte de modules qui permettront de
remplir les trous dans ces réseaux, ni d’associer des protéines enzymatiques à ces motifs.
En 2013, Barba et al. [26] ont identifié le fait que l’enchaînement des réactions constituant les
voies de dégradation des purines et pyrimidines présente la même biochimie, ainsi que le fait que
ces réactions sont catalysées par des enzymes homologues. Ceci a permis d’introduire la notion
de module réactionnel, comme étant une succession de transformations enzymatiques catalysées
par des protéines homologues. Ils ont aussi démontré, grâce à l’expérimentation biochimique, que
le module découvert a une capacité prédictive et renferme une voie de catabolisme des purines
encore inconnue. Cependant, cette étude ne permet pas de généraliser l’approche de découverte
de modules conservés du métabolisme et de l’appliquer d’une façon systématique et automatique
afin de découvrir de nouvelles voies métaboliques.
Toujours en 2013, Muto et al. [27] publient les résultats de leur recherche systématique de
modules réactionnels dans la base de données KEGG [28]. A partir de l’analyse des motifs de
transformation structurale des composés chimiques pour toutes les voies métaboliques présentes
dans cette base de données, ils ont mis en évidence l’architecture modulaire du métabolisme, ainsi
que le caractère conservé de ces modules au travers des voies métaboliques en les alignant.
Cependant, le lien entre ces modules réactionnels et les protéines permettant de catalyser les
réactions comprises dans ces modules n’est pas fait, la méthode ne peut s’appliquer à d’autres
donnés que celles présentes dans KEGG.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-15-2048.jpg)
![10
Ces études mettent en évidence la logique modulaire des réseaux métaboliques et on peut voir
que l’idée de prédire des nouvelles activités enzymatiques en explorant cette modularité
commence à apparaître. Cependant, l’étude de Barba et al. ne permet pas de généraliser
l’approche au métabolisme entier, et celles de Lacroix et al. et de Muto et al. ne permettent pas de
faire le lien entre les modules réactionnels et les familles de protéines qui catalysent ces réactions.
De plus, la méthode de Muto et al. ne permet pas de découvrir des modules réactionnels
chevauchant plusieurs voies métaboliques, point plutôt crucial pour découvrir des enchainements
nouveaux d’activités enzymatiques et nécessite une post-curation experte pour valider les
modules trouvés.
C’est dans ce contexte de double problématique de gènes de fonction inconnue et d’activités
enzymatiques inconnues que l'étude à l'origine de cette thèse a été développée. Le travail a
consisté à définir des modules de transformations chimiques dans le métabolisme, à identifier les
plus conservés d'entre eux et à les explorer en les associant à des modules génomiques (comme
les opérons, par exemple) de fonction pas ou peu connue.
Toutefois, avant de développer cette méthode, une étude étendue a été réalisée sur les activités
enzymatiques orphelines de séquences aussi appelées "enzymes orphelines". Il s'agit d'activités
enzymatiques démontrées expérimentalement comme étant présentes dans un organisme donné,
mais dont la séquence codant pour l'enzyme catalysant cette activité est inconnue. En effet,
depuis 2007 [5], il n'y a pas eu de mise à jour sur ce phénomène qui touche pourtant entre 20 et
30% [7, 8] des activités enzymatiques connues. Le concept d'enzyme orpheline locale a aussi été
introduit : une activité enzymatique non-orpheline dans un clade donné mais orpheline dans un
autre. Ce concept met à jour les difficultés rencontrées par l'annotation fonctionnelle
automatique et met en avant les "NISE" - "Non-Homologous Isofunctionnal Enzymes" : des
enzymes non-homologues mais ayant la même activité catalytique. Cette étude a fait l’objet d'une
publication [8] et est décrite dans le premier chapitre de ce manuscrit.
Un travail plus méthodologique a ensuite été réalisé et constitue l’objet principal de cette thèse.
La démarche a consisté en l'exploration du métabolisme au travers de modules conservés de
transformations chimiques via la construction d’un modèle compressé de tout le métabolisme
connu qui regroupe des réactions entre elles selon leur type de transformation chimique. Pour
cela, un réseau de réactions représentant un modèle global du métabolisme a été construit à partir](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-16-2048.jpg)
![11
des données sur les réactions et les voies métaboliques présentes dans les bases de données
publiques. Au préalable, une classification des réactions en fonction de leur type de
transformation chimique a été réalisée en utilisant les signatures moléculaires des réactions (RMS)
[29]. En regroupant les nœuds des réactions partageant le même type de transformation chimique
en un seul nœud, un réseau de RMS a été crée. Dans ce réseau, les nœuds représentent un type de
transformation chimique, regroupant ainsi toutes les réactions enzymatiques effectuant ce type de
transformation, et les arêtes reprennent tous les liens existants dans le réseau original de
réactions. Ce réseau de RMS contient l’information sur toutes les réactions connues à partir
desquelles il a été construit, mais aussi l’information sur les réactions encore inconnues, qu’il est
possible de déduire à partir de leur type de transformation chimique et de leur contexte dans ce
réseau. Ainsi, le réseau de RMS est une représentation globale et condensée des connaissances
actuelles sur le métabolisme et possède en plus un potentiel prédictif de nouveaux modules
réactionnels. Si on émet l’hypothèse de la modularité du métabolisme, c'est à dire que les
réactions forment des blocs conservés au cours de l'évolution, le modèle réduit de
transformations chimiques est aussi modulaire et contient des blocs conservés de transformations
chimiques. L’étape suivante consiste donc à identifier les différents types de conservation
d’enchaînements (ou chemins) de transformations chimiques dans ce réseau de RMS. Ensuite,
des métriques de conservation d'un chemin/module de RMS sont définies, basées sur la
conservation des motifs de transformations chimiques entre les voies métaboliques connues, la
conservation de ces motifs au travers de tout le métabolisme, leur conservation du point de vue
enzymatique dans la taxonomie ou encore du point de vue topologique du réseau. L’ensemble
des chemins possibles a été extrait à partir du réseau de RMS et un certain nombre s’est révélé
être très conservé. Cette méthode a fait l’objet d'une publication [30] et est décrite dans le
deuxième chapitre de cette thèse. Une partie de ces chemins conservés est identifiée, car ils
correspondent à des voies métaboliques connues, mais beaucoup de chemins ne correspondent à
rien de connu jusqu’ici, et nécessitent un effort d’identification.
Par conséquent, dans la troisième partie de ce manuscrit, est décrit le processus d’identification
de modules conservés dans le métabolisme de transformations chimiques pour l’annotation des
blocs génomiques fonctionnels tels que les opérons (unités génomiques fonctionnelles, présentes
essentiellement chez les bactéries et archées, contenant un ensemble de gènes co-transcrits et
contrôlés par un même promoteur) de fonction peu ou pas connue. Les gènes, qui encodent des
enzymes et qui sont retrouvés dans ce type de structures génomiques, sont souvent impliqués
dans les mêmes fonctions cellulaires, assimilables aux voies métaboliques. Un exemple classique](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-17-2048.jpg)





![17
I. Le métabolisme
La vie est un concept difficile à définir. Il y a plusieurs façons différentes de penser à la vie, et,
pour compliquer les choses encore plus, il y a de multiples définitions académiques. On peut
penser à la vie comme à « la chair et le sang », ou comme à une machine ou un automate. On
peut aussi penser aux briques élémentaires – les molécules de la vie, ou encore, à l’information
contenue dans celles-ci. Plusieurs définitions scientifiques plus ou moins précises existent. Leslie
Orgel [31] par exemple, a défini une entité vivante avec le terme « CITROENS » (Complex,
Information-Transforming Reproducing Object that Evolves by Natural Selection – des objets complexes
ayant la capacité de transformer l’information et de se reproduire tout en évoluant par sélection
naturelle). Norman Horowitz, un des premiers généticiens à travailler sur les théories de
l’évolution du métabolisme et après avoir travaillé sur la recherche de la vie dans le système
solaire, donne une définition de la vie basée sur la génétique. Selon lui, être en vie équivaut à
posséder des propriétés génétiques, qui sont notamment l’autoréplication, la catalyse et la
mutabilité [32]. De plus en plus de scientifiques, cependant, déclarent que l’on ne peut pas encore
définir ce qu’est la vie, car on n’en sait pas encore suffisamment sur sa nature, mais qu’on peut
toutefois prédire ce qu’est vivant ou non sans avoir une définition générale. La plupart des
définitions de ce que c’est qu’un organisme vivant, bien que différentes sur certains points, se
rejoignent sur le fait que transformer la matière par des réactions chimiques est nécessaire à la
création et au maintien de la vie. L’ensemble de ces réactions, souvent catalysées par des
protéines produites par l’organisme (ou par des protéines « empruntées » à d’autres organismes
comme c’est le cas des virus), ainsi que les petites molécules organiques qu’elles transforment,
s’appelle le métabolisme et est au cœur de cette thèse.
I.1 Qu’est-ce qu’est le métabolisme ?
Le métabolisme est l’ensemble de processus biochimiques à travers lesquels les organismes
vivants se maintiennent en vie, se développent, se reproduisent et interagissent avec
l’environnement. Par ailleurs, le terme « métabolisme », qui est retrouvé dans beaucoup de
langues différentes, vient du grec « µεταβολή » (metabôlé) et signifie changement ou
transformation. Les transformations chimiques opérées dans les organismes vivants concernent](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-23-2048.jpg)


![20
Le métabolome est l’ensemble des métabolites dans un organisme donné à un temps donné. Il
est donc constitué d’un grand nombre de molécules organiques appartenant à diverses classes
comme les acides aminés, les peptides, les lipides, les nucléotides ou les sucres. Le nombre total
de métabolites est estimé entre 200000 et 1000000 d’après [33].
La métabolomique est l’étude du métabolome dans des conditions biologiques données, et
s’emploie à identifier et quantifier les métabolites d’un organisme. Le métabolome d’un même
organisme peut être très différent selon l’environnement, de son état de stress, de l’âge, d’une
modification génétique, etc.. Deux techniques principales permettent de nos jours d’obtenir un
métabolome : la résonnance magnétique nucléaire et la spectrométrie de masse [34]. Les deux
doivent cependant être combinées pour obtenir un métabolome relativement complet, car aucune
n’est capable de d’identifier tous les types de métabolites. Le traitement automatique de ces
données est un des plus gros défis actuels en bio- et chemo-informatique [34].
Figure 2. Identifiants IUPAC de l’acide acétique, de la L-lysine et du Coenzyme A. Pour certaines molécules, plusieurs
identifiants officiels sont possibles. Lorsqu’il s’agit de grosses molécules ces identifiants deviennent compliqués à
utiliser pour un humain.
Un composé chimique possède une structure chimique unique et bien définie. La formule brute
d’un composé chimique n’indique que sa composition en atomes et ne reflète pas sa structure,
ainsi, deux composés chimiques distincts peuvent avoir la même formule brute (par exemple la](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-26-2048.jpg)
![21
formule brute C2H4O2 décrit l’acide acétique, le glycoaldehyde et le methyl formate, des composés
chimiques ayant une structure pourtant différente Figure 1). L’identification des molécules se fait
de plusieurs façons. Tout d’abord, il y a les numéros CAS (Chemical Abstracts Service Registry
Numbers [35]) qui sont des identifiants numériques uniques assignés à chaque molécule décrite
dans la littérature scientifique. Par exemple, l’identifiant CAS de l’acide acétique est 64-19-7.
Ensuite, il y a la nomenclature IUPAC (International Union of Pure and Applied Chemistry),
qui est une méthode systématique de nommage de composés chimiques organiques [36]. Dans
l’idéal selon cette nomenclature, chaque composé chimique devrait avoir un nom tel qu’une
structure 2D non-ambiguë puisse être crée. Par exemple, le nom IUPAC de l’acide acétique est
« acetic acid ». Cependant, les identifiants IUPAC sont rarement utilisés par la communauté de
biologistes car les noms pour les grandes molécules peuvent devenir très rapidement très
compliqués (Figure 2). Il en résulte des problèmes d’identification des composés chimiques,
notamment donner le même nom à des structures différentes ou des noms différents à la même
structure. Il existe donc plusieurs façons informatiques d’encoder la structure 2D des molécules
chimiques pour lever les ambiguïtés.
La première façon d’encode la structure 2D est celle des fichiers molfile (MDL molfile format).
C’est un format de fichier crée par la société MDL (maintenant devenu Symyx qui a fusionné
avec Accelrys : http://accelrys.com ; Accelrys ayant récemment été racheté par Dassault
Systèmes), et contient l’information sur les atomes, les liaisons entre les atomes, la connectivité et
les coordonnées spatiales pour une molécule (Figure 3). Les fichiers SDF (Structure-Data File)
Figure 3. Fichier MOLFILE de l’aldehydo-D-glucose-6-phosphate. Les fichiers MOLFILE décrivent les
coordonnées tridimensionnelles des atomes de la molécule.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-27-2048.jpg)
![22
utilisent le format molfile. Dans ces fichiers, il y a plusieurs composés chimiques au format
molfile séparés par des lignes de quatre caractères dollar ($$$$). Une des particularités du format
SDF est qu’on peut y inclure des données supplémentaires associées aux molécules, comme les
identifiants officiels des molécules, leurs identifiants dans différentes bases de données ou des
commentaires de l’utilisateur.
Figure 4. Descripteurs moléculaires de l’aldehydo-D-glucose-6-phosphate. (a) SMILES, (b) InChi, (c) InChi Key.
Une autre façon d’encoder la structure bidimensionnelle des composés chimiques est le format
SMILES (Simplified Molecular-Input Line-Entry System [37, 38]). C’est une notation linéaire
décrivant la structure de la molécule en utilisant des courtes chaines de caractères ASCII. Le
concept de génération d’une entrée SMILES est assez simple : il faut casser les éventuels cycles
pour ensuite décrire les branches à partir du squelette carboné de la molécule (Figure 4a).
Cependant, une même molécule peut être décrite par plusieurs signatures SMILES valables (par
exemple CCO, OCC et C(O)C spécifient correctement la structure de l’éthanol). Ainsi, des
algorithmes de canonisation de SMILES ont été créés pour assurer un code SMILES unique pour
une structure donnée indépendamment de l’ordre des atomes considéré dans la structure
dessinée. De ce fait, un SMILES officiel est unique pour chaque structure grâce à cette étape de
canonisation, c’est le SMILES canonique (Canonical SMILES). Pour une molécule donnée, il
peut aussi y avoir un SMILES isomérique, qui est une chaine de caractères contenant
l’information sur la conformation des doubles liaisons et la chiralité.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-28-2048.jpg)
![23
La dernière façon standard de représenter une structure chimique est le code InChI [39] (IUPAC
International Chemical Identifier - http://www.iupac.org/inchi). C’est un identifiant textuel pour
les composés chimiques basé sur plusieurs types d’information : les atomes, la connectivité
interatomique, l’information sur les tautomères, les isotopes, la stéréochimie et sur les charges
électroniques. C’est un identifiant unique à chaque molécule indépendamment de la façon dont
celle-ci est dessinée (contrairement, notamment, aux fichiers molfile et aux codes SMILES qui
varient en fonction de la façon dont la molécule est dessinée). Depuis 2009, est disponible un
logiciel générant des InChI standardisés, à partir desquels il est possible de générer des clés
uniques InChI Keys (Figure 4b et c). La standardisation des InChi simplifie leur comparaison du
point de vue informatique et permet une uniformisation des données à travers les ressources
publiques.
La conception et l’utilisation de descripteurs moléculaires (méthodes pour décrire toutes sortes
d’informations chimiques et topologiques d’une molécule chimique) est une branche à part
entière de la chemo-informatique (on pourra notamment consulter le livre [40] pour constater
l’étendue du domaine). Contrairement aux identifiants moléculaires présentés précédemment, les
descripteurs moléculaires sont utilisés pour calculer des propriétés chimiques (QSPR – quantitative
structure-property relationship – relation quantitative structure-propriété) ou d’activité chimique
(QSAR – quantitative structure-activity relationship – relation quantitative structure-activité). Les
descripteurs moléculaires peuvent être classifiés en cinq catégories, selon les dimensions qu’ils
couvrent : 0D (nombre de liens, poids moléculaire, nombre d’atomes), 1D (comptages de
fragments moléculaires, liens hydrogène, surface polaire, etc), 2D (rassemblant les descripteurs
Figure 5. Fullerène. Cette molécule sphérique est composée de cycles de carbone et est généralement complexe à décrire
d’une façon systématique avec des descripteurs moléculaires.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-29-2048.jpg)
![24
topologiques), 3D (contenant les descripteurs géométriques et les informations sur les propriétés
de surface) et 4D (contenant les coordonnées 3D ainsi que les informations de conformation).
Deux descripteurs moléculaires seront décrits ici : les descripteurs moléculaires de signatures
stéréo [41] calculés par le logiciel MolSig (http://molsig.sourceforge.net) et les descripteurs
KEGG Chemical Function and Substructure (KCF-S) [42].
L’algorithme MolSig [41], générateur des descripteurs moléculaires de signatures stéréo (MS),
tient compte de la conformation stéréochimique des molécules en plus de leur topologie. Il
permet de générer des MS pour des structures stéréochimiques complexes comme par exemple
les fullerènes (Figure 5) et est efficace du point de vue computationnel. Cette méthode considère
une molécule comme un graphe où les atomes sont des nœuds et les liens entre les atomes des
arêtes et calcule un sous-graphe d’un diamètre donné centré sur chacun des atomes de la
molécule. Le formalisme SMILES est utilisé pour décrire les sous-graphes pour chaque atome.
L’algorithme prend en entrée un fichier molfile. La signature moléculaire obtenue est une
représentation sur plusieurs lignes, avec une sous-structure par ligne et le nombre de fois où cette
sous-structure est rencontrée dans la molécule (un exemple de MS est présenté en Figure 6).
Figure 6. Signature moléculaire de hauteur 1 de l’aldehydo-D-glucose-6-phosphate calculée avec le logiciel MolSig.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-30-2048.jpg)
![25
Les KEGG Chemical Function and Substructure (KCF-S [42]) étend le format KCF en y
ajoutant sept attributs décrivant des sous-structures biochimiques. Le format KCF comporte
trois sections, « ENTRY », « BOND » et « ATOM ». ENTRY indique l’identifiant KEGG (base
de données métaboliques, cf. section II) de l’entrée ainsi que son type. Dans la section ATOM
sont présentés les numérotations des atomes, les « KEGG atom types » (les types d’atomes selon
le formalisme KEGG) pour les étiquettes sur les atomes, l’espèce chimique de chaque atome
(« C » pour carbone par exemple) ainsi que leurs coordonnées 2D. La section BOND décrit la
numérotation des liens, les numérotations des deux atomes impliqués dans le lien ainsi que la
configuration stérique du lien (Figure 7). Le descripteur moléculaire KCF-S étend cette
représentation de la molécule en y ajoutant les attributs suivants : TRIPLET, VICINITY, RING,
SKELETON, INORGANIC. La conversion en KCF et KCF-S se fait à partir d’un fichier
molfile.
Ces deux exemples de descripteurs moléculaires ajoutent des informations sur les sous-structures
moléculaires aux coordonnées spatiales de chaque atome, présentes dans un simple fichier
molfile. Ceci permet de réaliser des manipulations plus complexes sur les molécules, notamment
de suivre leurs implications dans les réactions ainsi que la façon dont les réactions les
transforment.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-31-2048.jpg)
![26
I.2.2 Réactions
Les métabolites sont transformés au cours des réactions biochimiques. Les molécules
transformées au cours d’une réaction sont appelées substrats et les molécules résultantes d’une
réaction sont des produits. Une réaction est souvent représentée par son équation bilan, dans
laquelle sont décrites les formules chimiques des produits et des substrats, leurs relations, la
direction de la réaction ainsi que sa stœchiométrie, c’est à dire la proportion de molécules
nécessaire au maintien du principe de conservation de la masse (« Rien ne se perd, rien ne se crée,
tout se transforme » d’après Antoine de Lavoisier, un des pères de la chimie moderne). Ainsi, au
cours d’une réaction les molécules échangent des atomes ou des groupes d’atomes. La
transformation chimique opérée pendant une réaction, c’est à dire la façon dont l’échange
d’atomes ou de groupes d’atomes se produit, peut être la même pour des réactions agissant sur
des molécules différentes. On dit alors que ces réactions réalisent le même type de
transformation chimique.
Figure 7. Descripteur moléculaire KEGG Chemical Function and Substructure (KCF-S) (image extraite de
Kotera et al. [42]).](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-32-2048.jpg)

![28
François Persoz [43], elle dégradait l’amidon et a été nommée « diastase », ce qui signifie
« séparation » en grec. Même si cette enzyme a par la suite été renommée en « amylase », la
tendance à donner aux enzymes des noms qui se terminent par le suffixe « ase » date de cette
époque. Le mot « enzyme » vient du grec ancien « zumê » qui signifie « levain », et a été introduit
en 1877 par Wilhelm Kühne qui travaillait sur le processus de fermentation.
Les enzymes sont généralement des protéines, elles sont donc encodées dans le génome et font
suite à l’expression des gènes par le processus de transcription et traduction amenant à la
synthèse de chaines polypeptides composés à partir d’acides aminés. Ces protéines peuvent être
constituées d’un seul polypeptide (protéine monomérique) ou de plusieurs chaines
polypeptidiques (protéine multimérique) encodées par un ou plusieurs gènes. D’autre part, les
protéines sont aussi constituées de domaines protéiques, qui sont des parties d’une ou plusieurs
chaines polypeptidiques ayant des propriétés particulières, par exemple, adopter une structure de
manière autonome ou quasi-autonome du reste de la molécule. Une des branches importantes de
la bioinformatique structurale consiste à effectuer une classification étendue des domaines
structuraux et des protéines en général. Un domaine peut être porteur, par exemple, de la
fonction de catalyse (c’est à dire qu’il contiendra le site catalytique de l’enzyme) et un autre peut
servir à lier le substrat. Les multiples aspects liés à l’assignation de fonctions enzymatiques aux
protéines et aux domaines protéiques sont présentés dans la section III de ce chapitre.
La catalyse est une action qui permet à la réaction de se dérouler dans un milieu dans lequel elle
ne pourrait pas se faire et/ou d’accélérer grandement cette réaction. Les enzymes agissent à faible
concentration (il en faut très peu dans le compartiment cellulaire donné pour que la catalyse
puisse avoir lieu) et ne sont généralement pas modifiées au cours de la réaction. Les enzymes
possèdent des poches catalytiques dans lesquelles les substrats sont stabilisés (différents
mécanismes sont utilisés pour cette stabilisation, comme le rapprochement forcé des substrats,
stabilisation par effet électrostatique ou par l’hydrophobicité, par exemple) afin que la réaction
puisse se produire. La taille et la forme de la poche catalytique de l’enzyme, ainsi que certains
acides aminés clés impliqués directement dans le mécanisme réactionnel, régissent la spécificité de
l’enzyme. En effet, certaines enzymes sont spécifiques d’un substrat donné, d’autres sont plus
généralistes et peuvent transformer plusieurs substrats possédant une même fonction chimique.
Une enzyme peut avoir plusieurs sites catalytiques, soit dans une même poche catalytique soit
dans deux poches catalytiques différentes (situées sur des domaines différents ou non), on parle](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-34-2048.jpg)
![29
alors d’enzyme multifonctionnelle. Une enzyme peut aussi changer de fonction catalytique et de
spécificité de substrat en fonction de l’environnement dans lequel elle est présente (température,
PH) ou en fonction de la présence de certains métabolites pouvant provoquer un changement de
conformation spatiale de l’enzyme. Les enzymes du premier cas se nomment les « moonlighting
proteins » et leur étude est assez complexe [44–46]. Les enzymes du deuxième cas appartiennent
à la catégorie des enzymes allostériques [47, 48]. Ces enzymes possèdent au moins un site de
fixation de métabolite distant de la poche catalytique, et la fixation d’un métabolite sur ce site
modifie la conformation structurale de l’enzyme. Ce changement de conformation peut avoir un
effet négatif (le métabolite est alors un inhibiteur) ou positif (métabolite activateur). En
ingénierie enzymatique, l’allostérie est de plus en plus utilisée pour contrôler les enzymes d’intérêt
[49].
I.2.4 Cofacteurs
Les derniers acteurs du métabolisme qui seront décrits ici sont les cofacteurs. Un cofacteur est
une molécule non-protéique qui se fixe sur une enzyme. Ces molécules sont souvent
indispensables à leur bon fonctionnement, ce sont des « molécules d’assistance ». Une enzyme
sans cofacteur et inactive est appelée apoenzyme. L’enzyme avec le cofacteur fixé est
l’holoenzyme. Les cofacteurs peuvent être classifiés en trois catégories : les ions métalliques, les
cofacteurs faiblement liés à l’enzyme et les cofacteurs fortement liés à l’enzyme.
Les ions métalliques permettent principalement le maintien de la structure de l’enzyme. Les ions
les plus fréquents sont les ions fer, cuivre, magnésium, nickel, zinc, manganèse et molybdenium.
Ils se lient d’une façon covalente à l’enzyme. Un ou plusieurs ions de même nature ou de natures
chimiques différentes peuvent être nécessaires à son bon fonctionnement. Les ions métalliques
ne sont pas transformés pendant la réaction enzymatique et n’apparaissent pas dans l’équation de
la réaction.
Les cofacteurs faiblement liés à l’enzyme sont des coenzymes et sont généralement libérés après
la réaction. La liaison à l’enzyme est généralement une liaison hydrogène ou ionique. Ils sont
transformés pendant la réaction enzymatique, sont souvent appelés co-substrats et apparaissent
dans l’équation de la réaction. Les coenzymes sont généralement en excès dans le milieu
cellulaire. Parmi les coenzymes les plus fréquents il y a le nucléotide adénosine monophosphate
(AMP), le nucléotide adénosine triphosphate (ATP), le coenzyme A (CoA), la nicotinamide](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-35-2048.jpg)
![30
adénine dinucléotide (NAD) et la nicotinamide adénine dinucléotide phosphate (NADP) et leur
formes réduites NADH et NADPH. Il est d’ailleurs intéressant de préciser que beaucoup de
cofacteurs possèdent dans leur structure l’AMP, ce qui peut refléter une origine évolutive
commune. Une hypothèse [50] suggère que la structure de l’AMP est considérée comme une
sorte de poignée dont les enzymes se servent pour basculer le coenzyme entre les différentes
poches catalytiques. Par ailleurs, la géométrie de la liaison de l’AMP mime d’une façon presque
exacte la géométrie de l’appariement des bases dans l’ADN et l’ARN.
Les cofacteurs fortement liés à l’enzyme, c’est à dire par une liaison covalente, sont appelés
groupements prosthétiques. Ce sont des molécules organiques au centre desquelles sont
souvent trouvés un ou plusieurs atomes métalliques. Les exemples les plus fréquents de
groupements prosthétiques sont l’hème (intervenant dans la plupart des réactions avec de
l’oxygène) et un certain nombre de vitamines.
Tous les acteurs du métabolisme ont pour but de satisfaire des objectifs de la cellule. Ces
objectifs peuvent concerner la production d’énergie, la communication, la défense ou la
construction ou le remplacement d’éléments constituant la structure même de la cellule. Afin
d’atteindre ces objectifs, il est souvent nécessaire d’effectuer plusieurs transformations chimiques
consécutives sur les métabolites. Ces enchainements sont aussi appelés voies métaboliques et
sont présentés dans la section suivante.
I.2.5 Voies métaboliques
Classiquement, on définit une voie métabolique comme un enchainement d’étapes de
transformations de métabolites, ces étapes de transformations étant catalysées la plupart du
temps par des enzymes. Une voie métabolique est caractérisée par un métabolite de départ
(substrat initial) et un métabolite cible (produit final de la voie). Il peut y avoir plusieurs
enchainements de réactions différents qui ont le même substrat initial et le même produit final.
Dans ce cas on dit que la voie métabolique possède plusieurs variants.
En 1999 Harold Morowitz [51] décrit l’ensemble des voies métaboliques connues comme « une
vaste généralisation empirique basée sur un siècle et demi de travail d’une armée de biochimistes
qui se sont efforcés de caractériser toutes les réactions chimiques se déroulant dans les cellules
vivantes ». Ainsi, lorsque l’on veut définir la notion de voie métabolique, il faut garder à l’esprit](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-36-2048.jpg)
![31
que celle-ci est une vision humaine pour diviser le réseau métabolique en sous-parties plus faciles
à comprendre, à étudier et à reproduire. C’est avant tout un concept créé pour appréhender une
fonction biologique donnée, car les enzymes et les métabolites sont la plupart du temps en état
libre dans le compartiment cellulaire où ils se trouvent, et la rencontre d’un métabolite et d’une
poche catalytique d’une enzyme peut âtre considérée comme « accidentelle/fortuite ». La
nécessité des organismes d’avoir l’ensemble des enzymes qui catalysent les réactions servant à
obtenir un métabolite essentiel à un moment donné, les « pousse » à co-réguler l’expression des
gènes codant pour ces enzymes. En effet, chez les procaryotes et certains eucaryotes, il existe
une relation entre l’ordre et la co-localisation des gènes sur les chromosomes qui favorise leur co-
expression et, ainsi, l’enchainement en voie métabolique des réactions catalysées par les enzymes
correspondantes [52]. De plus, des similitudes dans la structure des voies métaboliques dans un
organisme et entre les organismes, même éloignés du point de vue taxonomique et intra-
organismes, sont observées [25, 26]. Ainsi, il existe bien une logique conservée au cours de
l’évolution de l’agencement des réactions en voies métaboliques.
Les voies métaboliques peuvent être séparées en deux grands groupes selon qu’elles sont
essentielles ou non à la survie de l’organisme. Les voies essentielles à la survie de l’organisme
composent le métabolisme primaire, comme par exemple, les voies de biosynthèse des acides
aminés ou des nucléotides. Il est généralement très conservé au travers de l’arbre du vivant (un
ensemble de 124 réactions « super-essentielles » communes à tous les organismes a d’ailleurs été
défini [53]). Les voies métaboliques qui ne sont pas indispensables à la survie de l’organisme
composent le métabolisme secondaire. Le métabolisme secondaire varie beaucoup entre
différentes branches taxonomiques, mais aussi en fonction de l’environnement des organismes.
Ce sont notamment les voies du métabolisme secondaires qui permettent la production de
molécules de défense comme les toxines ou les antibiotiques, ou encore des molécules de
communication comme les hormones (Figure 9).](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-37-2048.jpg)

![33
I.3 Evolution du métabolisme
L’évolution (du latin « evolutio » - action de dérouler) est le passage progressif d’un état à un autre.
L’évolution biologique se définit comme le changement dans les traits héréditaires des
populations au fil des générations successives [54]. Les processus évolutifs ont des implications à
tous les niveaux de l’organisation biologique, que ce soit au niveau des espèces, des individus, des
cellules ou des molécules. L’évolution du métabolisme peut se définir comme l’acquisition de
nouvelles capacités métaboliques, c’est à dire la capacité de synthétiser et de dégrader de
nouvelles molécules, ou de réaliser ces transformations d’une manière plus efficace. La perte de
certaines parties du métabolisme fait aussi partie de son évolution. Dans cette section nous allons
nous intéresser à deux aspects complémentaires de l’évolution du métabolisme, l’évolution des
enzymes dans un premier temps et l’évolution des voies métaboliques ensuite.
I.3.1 Evolution des enzymes
Les protéines en général, et les protéines enzymatiques en particulier, ont différentes
formes/structures et tailles. Pour réaliser certaines fonctions, les protéines n’ont besoin que d’un
seul domaine, une unité de structure protéique stable. Il existe même des protéines qui n’ont pas
besoin d’être repliées en une structure particulière pour avoir une fonction catalytique, on parle
alors de protéines intrinsèquement non-structurées [55]. D’autres protéines, pour être
fonctionnelles, sont composées de plusieurs domaines reliés entre eux ou même de plusieurs
polypeptides formant un complexe protéique. L’apparition de nouvelles fonctions enzymatiques
dans les organismes se fait principalement via duplication de gènes suivie d’une divergence des
copies par acquisition de mutations qui sont sélectionnées pour être plus viables et/ou favoriser
l’adaptation de l’organisme à un milieu donné en augmentant son efficacité métabolique.
Divergence des fonctions enzymatiques - enzymes promiscuitaires
Les enzymes sont connues pour être des catalyseurs extrêmement spécifiques. Pourtant, l’idée
que beaucoup d’enzymes sont capables de catalyser d’autres réactions et/ou de transformer](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-39-2048.jpg)
![34
d’autres substrats en plus de ceux pour lesquels elles ont se sont spécialisées au cours de
l’évolution n’est pas nouvelle [56]. Ces enzymes, qui ne font pas que ce qu’on attend d’elles, sont
appelées enzymes promiscuitaires. Une des premières publications sur une enzyme
promiscuitaire date de 1921 et décrit la pyruvate décarboxylase pour sa capacité à former des
liaisons carbone-carbone entre de nombreuses molécules [57]. Une des grandes hypothèses
actuelles propose que les activités enzymatiques promiscuitaires servent de point de départ pour
l’évolution des organismes et de leur métabolisme. Il existe trois types de promiscuité :
• la promiscuité de substrat, où l’enzyme est capable de catalyser la même transformation
sur d’autres substrats que ceux pour lesquels elle est spécialisée, avec une plus ou moins
bonne efficacité
• la promiscuité de réaction, où l’enzyme a la capacité de catalyser plusieurs
transformations différentes
• la promiscuité de condition, remarquée chez des protéines dont la fonction peut varier
considérablement suivant les conditions physico-chimiques (variation de température,
pH, salinité, ou présence/absence de certaines molécules dans le milieu). Les enzymes
promiscuitaires de condition sont souvent appelées « moonlighting enzymes ».
Le potentiel promiscuitaire des enzymes entraine l’évolution de nouvelles fonctions enzymatiques
au sein de superfamilles structurales [58] et par conséquence, l’émergence de nouvelles familles
ou superfamilles d’enzymes [59, 60]. Chez les organismes procaryotes notamment, leur style de
vie influence les enzymes à être promiscuitaires [61], cette plasticité catalytique favorisant
grandement la survie en cas de changement brutal de l’environnement.
La promiscuité enzymatique, ainsi que le potentiel « d’évolvabilité » promiscuitaire des enzymes
peut être prédite avec des méthodes chémoinformatiques et statistiques [62].
Comme évoqué précédemment, la duplication de gènes est un des principaux facteurs favorisant
l’évolution de la fonction des protéines. La duplication d’un gène codant une enzyme entraine la
présence de deux versions de l’enzyme dans l’organisme. La pression évolutive pour garder la
fonction enzymatique présente initialement dans l’organisme ne s’exerçant que sur une seule des
deux copies, l’autre version peut évoluer en subissant un taux plus important de mutations [63].
Ce mécanisme permet à un organisme d’acquérir de nouvelles enzymes, soit ayant une activité
catalytique innovante et éventuellement bénéfique pour l’organisme [64], soit ayant la même
activité, mais la réalisant avec une efficacité plus ou moins grande. Ce dernier cas concerne les
isoenzymes.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-40-2048.jpg)
![35
Isoenzymes
Les isoenzymes (aussi appelées « isozymes ») sont des enzymes qui ont des séquences d’acides
aminés différentes mais qui catalysent la même réaction biochimique. La différence en séquence
peut être très importante, impliquant une origine évolutive différente des isoenzymes, ou
relativement faible, les isoenzymes étant homologues. Dans le premier cas, la même activité
enzymatique est acquise par convergence évolutive et le cas de ces enzymes isofonctionnelles sera
abordé dans la section suivante.
La présence de deux isoenzymes homologues dans un organisme a pour origine un événement de
duplication de gènes suivi de la différenciation des deux copies. Ces enzymes ont généralement
des modes de fonctionnement différents et/ou des propriétés de régulation différentes. Souvent,
les deux enzymes ont des vitesses d’évolution différentes, la pression de sélection ne s’exerçant
pas de la même manière sur les deux copies. La présence de deux isoenzymes dans un organisme
permet une meilleure adaptation de son métabolisme pour répondre à des besoins différents
suivant des conditions extérieures variables.
Un exemple très étudié d’isoenzymes porte sur l’activité pyruvate kinase chez Escherichia coli. Cette
bactérie, comme beaucoup d’autres, possède deux protéines ayant cette activité catalytique : PykA
et PykF. Ces protéines sont homologues (37% d’identité de séquence en acides aminés), mais
présentent des propriétés physico-chimiques différentes, sont sous un contrôle génétique
différent [65] et ne sont pas interchangeables.
Convergence évolutive de fonctions enzymatiques
Les NISE (Non-homologous Isofunctional Enzymes – des enzymes non-homologues isofonctionnelles)
[66] sont des enzymes qui catalysent les mêmes réactions biochimiques, mais qui ne sont pas
homologues, c’est à dire qu’elles n’ont pas évolué à partir d’un même gène ancestral. La plupart
du temps, elles ont des repliements structuraux différents, preuve d’une convergence évolutive
résultant de la nécessité des organismes à acquérir une fonction précise. On retrouve des NISE
dans des voies métaboliques essentielles comme dans la biosynthèse de la méthionine [67] ou du
coenzyme A (3 types d’enzyme réalisent l’activité pantothenate kinase dont une ne présentant
aucune homologie avec les deux autres types [68]). Un autre exemple pour illustrer les NISE est
l’activité enzymatique cellulase. Pour cette activité, catalysant la réaction de dégradation du
cellulose, il existe six versions différentes de la séquence avec des repliements très différents [66].](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-41-2048.jpg)
![36
L’acquisition d’une seule nouvelle fonction enzymatique dans un organisme est rarement
suffisante pour modifier profondément ses capacités métaboliques. Elle se fait de concert avec les
autres activités enzymatiques présentes dans l’organisme et par l’acquisition d’un ensemble
cohérent de fonctions catalysant une succession de réactions pour, par exemple, la dégradation
d’un nouveau composé de l’environnement en un métabolite d’intérêt pour l’organisme. Dans la
section suivante sont décrites les grandes théories sur les mécanismes d’acquisition de nouvelles
voies métaboliques par les organismes.
I.3.2 Grandes théories sur l’évolution des voies métaboliques
Il existe plusieurs grandes théories pour expliquer la façon dont les voies métaboliques sont
apparues et ont évolué. Les modèles correspondants à ces théories sont résumés dans la Figure
10 (partiellement inspirée de Schmidt et. al [69]).
Invention de novo des voies métaboliques
Le modèle le plus simple (voire simpliste) de l’évolution des voies métaboliques est celui de
l’invention de novo (Figure10a). Les voies métaboliques auraient pu apparaître et évoluer
spontanément, sans adapter ou réutiliser des enzymes préexistantes. Par exemple, un certain
nombre de d’ARNt synthétases semblent avoir initialement évolué d’une façon indépendante,
pour ensuite être impliquées dans différentes voies métaboliques comme celle de la traduction
des protéines et la transamidation ARNt-dépendante [70].
Synthèse rétrograde et synthèse progressive
La théorie sur l’évolution rétrograde des voies métaboliques par Norman Horowitz [71] est
historiquement la première a avoir été formulée (1945). Cette hypothèse soutient que la pression
de sélection sur une voie métabolique cible principalement la production fructueuse de son
produit final (Figure 10b). La formation du produit final à partir d’un métabolite intermédiaire
augmente la capacité vitale de l’organisme. Comme ce métabolite final peut dériver de
métabolites de plus en plus éloignés du point de vue chimique, la capacité vitale augmente et la](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-42-2048.jpg)
![37
voie métabolique évolue à rebours. Cette rétro-évolution semble être un bon modèle pour la
glycolyse [72] et la voie de biosynthèse du mandelate [73].
Une hypothèse alternative et moins connue que celle de la synthèse rétrograde est celle du
développement des voies de biosynthèse dans le sens avant [74] (aussi connue sous le nom de
celui qui l’a proposée, Sam Granick), où les composés terminaux ne joueraient aucun rôle dans
l’évolution. Granick proposa que la biosynthèse de certains produits terminaux pourrait être
expliquée par une évolution « vers l’avant » à partir de précurseurs relativement simples. Ce
modèle prédit que les composés biochimiques plus simples précèdent l’apparition des plus
compliqués. Par conséquent, les enzymes catalysant les étapes antérieures d’une voie métabolique
sont plus anciennes que celles catalysant les étapes suivantes. Pour que ce modèle puisse
fonctionner, il faudrait que les métabolites intermédiaires soient utiles à l’organisme, car
l’apparition simultanée de plusieurs enzymes catalysant des réactions consécutives est trop
improbable. Cette hypothèse peut fonctionner pour la biosynthèse de l’hème et de la chlorophylle
[74], mais ne fonctionne pas pour de nombreuses voies métaboliques comme la biosynthèse des
acides aminés ou des purines où les métabolites intermédiaires n’ont pas d’utilité apparente et
peuvent même être toxiques.
Spécialisation d’enzymes multifonctionnelles
Les voies métaboliques pourraient aussi évoluer à partir d’enzymes multifonctionnelles [64, 75]
(Figure 10c). A partir d’une enzyme multifonctionnelle catalysant plusieurs réactions consécutives
sur le même métabolite, la voie métabolique aurait pu évoluer avec la duplication et la
diversification de cette enzyme initiale vers des enzymes plus efficaces et plus spécialisées ne
catalysant chacune qu’une seule des étapes dans la voie. Des enzymes multifonctionnelles
actuelles, comme, par exemple, la carbamoyl phosphate synthase, sont utilisées dans de
nombreuses fonctions cellulaires et voies métaboliques, et pourraient être des précurseurs pour
de nouvelles voies métaboliques [76].
Duplication de voies métaboliques entières
De la même façon qu’une seule enzyme peut être dupliquée et se spécialiser, un bloc de gènes
participant à un même processus cellulaire peut aussi être dupliqué et se spécialiser, entrainant
naturellement la création d’une nouvelle voie métabolique [64, 77] (Figure 10d). Ce mécanisme
d’acquisition de nouvelles fonctions peut notamment être identifié en utilisant la génomique
comparative [78–80], notamment en observant une coévolution des opérons et des voies](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-43-2048.jpg)
![38
métaboliques. Par exemple, la voie de biosynthèse de l’histidine partage avec celles de la sérine et
du tryptophane plusieurs étapes qui possèdent un même type de transformation chimique et qui
sont catalysées par des enzymes homologues [77, 81]. Il est donc très probable que ces voies
métaboliques proviennent de duplications de voies ancestrales communes.
Recrutement enzymatique ou modèle d’évolution en « patchwork »
Les voies métaboliques pourraient aussi évoluer en « recrutant » des enzymes impliquées dans
d’autres voies métaboliques existantes, résultant en une mosaïque ou un « patchwork » d’enzymes
homologues qui catalysent des réactions dans différentes voies métaboliques [77, 82] (Figure 10e).
De nombreuse familles ou superfamilles d’enzymes catalysent des réactions similaires qui sont
rencontrées dans des voies métaboliques très différentes [83, 84], prouvant la plasticité des
réseaux métaboliques modernes [53]. Le recrutement des enzymes promiscuitaires dans les voies
métaboliques joue ainsi un grand rôle dans l’expansion du métabolisme [85]. Cette « versatilité »
enzymatique a été montrée à maintes reprises dont notamment chez Escherichia coli [86, 87].
Origine semi-enzymatique des voies métaboliques
Dans le but d’expliquer l’origine des toutes premières voies métaboliques, Lazcano et Miller [88]
ont proposé une hypothèse très différente des autres. Il est admis que la plupart des étapes des
voies métaboliques sont catalysées par des enzymes, mais certaines peuvent être naturellement
spontanées dans certaines conditions (température, pression, pH, présence/absence de molécules
particulières dans le milieu). Dans cette hypothèse, des enzymes très généralistes auraient permis
de modifier légèrement l’environnement de métabolites pour permettre aux réactions de se
dérouler spontanément. Il s’agirait alors d’étapes semi-enzymatiques dans les voies métaboliques
qui par la suite seraient remplacées par des étapes complètement enzymatiques au cours de
l’évolution, avec la spécialisation des enzymes (Figure 10f adaptée d’après Lazcano et Miller [88]).
D’après des études récentes [69, 79], le recrutement enzymatique semble être la principale force
motrice pour l’évolution de nouvelles voies métaboliques. La duplication de voies métaboliques
entières aurait aussi une grande importance dans l’évolution du métabolisme moderne. Les autres
hypothèses présentées semblent être des mécanismes évolutifs beaucoup plus rares ou
ancestraux. Il est important de noter également le rôle important du transfert horizontal de gènes
qui permet aux organismes microbiens d’acquérir rapidement de nouvelles compétences
métaboliques par échange de matériel génétique [89].](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-44-2048.jpg)
![39
Figure 10. Illustrations des grandes théories de l’évolution des voies métaboliques (adaptées d’après Scmidt et al. [69] et
Lazcano et Miller [88]). (a) Invention de novo des voies métaboliques, (b) Synthèse rétrograde, (c) Spécialisation
d’enzymes multifonctionnelles, (d) Duplication de voies métaboliques entières, (e) Modèle d’évolution en
« patchwork », (f) Modèle semi-enzymatique.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-45-2048.jpg)
![40
II. Représentation du métabolisme
En sciences, comme dans la vie de tous les jours, nous avons besoin de concepts et de structures
définis et communs à tous pour représenter les notions et les objets et communiquer d’une façon
efficace avec les autres individus. Comme nous l’avons vu dans la section précédente, le
métabolisme implique beaucoup d’acteurs de nature différente qui interagissent entre eux. Il est
donc nécessaire de codifier ces acteurs et leurs interactions. La quantité et la complexité des
données du métabolisme nécessitent l’utilisation des ordinateurs pour les intégrer et les
comprendre : c’est l’essence même de la bioinformatique.
Dans cette section seront décrits les différents niveaux et façons de représentation du
métabolisme. Dans un premier temps les différentes ressources de données publiques liées au
métabolisme seront passées en revue. Ensuite seront présentées diverses façons de classifier les
réactions chimiques catalysées par les enzymes : les activités enzymatiques.
Le métabolisme est souvent représenté sous la forme d’un graphe (Figure 11 d’après [90] et[120]).
En effet, ce type de structure permet d’intégrer à la fois des données sur les acteurs du
métabolisme (comme les métabolites, les réactions qui les transforment et les enzymes qui
catalysent ces réactions) et les interactions entre ces acteurs. Les troisième et quatrième parties de
cette section seront donc consacrées aux réseaux métaboliques.
Les études en biologie évolutive ont, à de très nombreuses reprises, démontré que le vivant est
modulaire, c’est à dire qu’il est composé, à tous les niveaux, d’unités conservées, ou modules,
ayant une existence propre et garantissant la cohérence de l’ensemble du système. A l’échelle
macroscopique, on pourra donner l’exemple de la transplantation médicale d’organes : un organe
est donc un des modules du système qu’est le corps d’un individu. A l’échelle microscopique, les
transposons, qui sont des petits morceaux d’ADN qui peuvent changer de place dans le génome
d’un organisme et même être échangés entre les organismes, pourront servir d’exemple de
modularité. La définition et la recherche des modules conservés de réactions dans les réseaux
métaboliques sont au cœur de cette thèse. La modularité du métabolisme et les concepts qui y
sont liés seront donc abordés dans la dernière partie de cette section.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-46-2048.jpg)

![42
II.1 Ressources de données métaboliques
Dans cette section seront présentées et décrites les différentes sources biologiques de données
publiques disponibles actuellement pour la communauté scientifique. La classification de ces
ressources en catégories bien distinctes est loin d’être évidente, car certaines d’entre elles sont
plutôt généralistes et contiennent beaucoup de types de données différentes (par exemple, des
données sur les molécules, les réactions, les enzymes et les voies métaboliques à la fois) et
d’autres ne contiennent qu’un seul type de données (par exemple uniquement des composés
chimiques).
II.1.1 Grandes bases de données sur le métabolisme
BioCyc & MetaCyc
BioCyc [91] est une collection de bases de données de génomes et de voies métaboliques (PGDB
– Pathway/Genome Data Base) et des outils pour comprendre ces données. MetaCyc [91–93] un des
PGDB de BioCyc, est une base de données curée de voies métaboliques expérimentalement
élucidées issues de tous les domaines du vivant. Au moment de l’écriture de ce manuscrit,
MetaCyc contient des données issues de 2600 organismes différents et 2260 voies métaboliques.
De plus, on y retrouve les métabolites, réactions, enzymes et gènes associés à ces voies
métaboliques. Le but de MetaCyc est de faire une description exhaustive du métabolisme via des
échantillons de voies métaboliques représentatives et expérimentalement élucidées. Les données
contenues dans MetaCyc sont accessibles au travers de son interface web (http://metacyc.org) ou
avec l’outil Pathway Tools [94, 95] qui permet une exploitation plus approfondie des données.
Les données des PGDBs peuvent aussi être utilisées directement en écrivant des programmes en
Java, Perl et Lisp. Les requêtes en Java et en Perl sont exécutées en utilisant les APIs (Application
Progam Interfaces) des systèmes appelés JavaCyc et PerlCyc [96].
Une des dernières nouveautés de MetaCyc est de proposer un atom mapping [97], c’est à dire le
marquage des atomes des molécules impliquées dans une réaction pour suivre leur flux au cours
de la transformation chimique.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-48-2048.jpg)
![43
Ce sont les données issues de MetaCyc qui ont été les plus utilisées pour les travaux présentés
dans cette thèse. Les données sur les voies métaboliques, les réactions et les métabolites ont été
extraites à l’aide de JavaCyc.
KEGG
KEGG [98–102] (Kyoto Encyclopedia of Genes and Genomes) est une des plus anciennes des bases de
données de réactions et de voies métaboliques. Ici, les voies métaboliques sont organisées en
cartes (maps) définies par objectif cellulaire et rassemblant tous les variants connus chez les
différents organismes. Dans cette base de données on retrouve tous les acteurs du métabolisme :
les métabolites (dans la section KEGG LIGAND), les réactions (KEGG REACTION), les
enzymes (KEGG ENZYME) et les voies métaboliques (KEGG PATHWAY et KEGG
MODULE). Il y a en plus des données sur les gènes et les génomes (KEGG GENES et KEGG
GENOME) ainsi que les groupes d’orthologues (KEGG ORTHOLOGY). Les cartes
métaboliques dans KEGG sont subdivisées en modules, qui sont des unités fonctionnelles
utilisées pour l’annotation et l’interprétation biologique des génomes.
Comparaison des bases de données MetaCyc et KEGG
La majeure différence entre KEGG et MetaCyc se trouve au niveau de la définition d’une voie
métabolique – il y a les « cartes » du côté de KEGG qui rassemblent pour tous les génomes
analysés, tous les variants possibles avec le même objectif cellulaire et, du côté de MetaCyc, des
voies métaboliques organisme (ou clade) spécifique. Dans KEGG, les voies métaboliques sont
généralement plus longues que dans MetaCyc (cf. Table 1). Les données dans MetaCyc sont
validées manuellement par des experts (ne travaillant pas nécessairement directement pour
MetaCyc), alors que dans KEGG une partie seulement est expertisée par des spécialistes internes
et les informations de l’autre partie sont inférées automatiquement. Une étude [103] comparant
les deux ressources a été publiée en 2013, et une partie de cette étude est résumée dans la Table 1.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-49-2048.jpg)
![44
Table 1. KEGG versus MetaCyc
Tableau de comparaison des bases de données de ressources métaboliques KEGG et MetaCyc. Adapté d’après [104].
Sont comparées les différentes statistiques sur les composés chimiques, les réactions et les voies métaboliques décrits
dans ces bases de données.
MetaCyc KEGG
Nombre de composés chimiques 11 991 15 161
Composés avec description 1 486 2 997
Longueur moyenne de la description 47,69 6,51
Nombre moyen de réactions associées à un composé 3,59 2,17
Nombre moyen de voies métaboliques par composé 1,78 0,67
Nombre de réactions 10 262 8 879
Nombre de réactions non-équilibrées 532 1 475
Nombre moyen de voies métaboliques associées à une réaction 0,84 0,90
Nombre de voies métaboliques 2 142 416
Nombre moyen de réactions par voie métabolique 5,73 19,10
BRENDA
BRENDA (BRaunschweig ENzyme DAtabase [105, 106]) est une ressource très complète sur les
enzymes, les réactions enzymatiques et les métabolites, contenant des données de très haute
qualité. Depuis peu de temps, on peut y retrouver aussi des informations sur les voies
métaboliques, mais celles-ci sont pour l’instant difficilement exploitables du point de vue
informatique. Les informations de cette base de données sont obtenues manuellement à partir de
la littérature, ainsi qu’en faisant de la fouille de données et de la fouille de texte et en utilisant des
algorithmes de prédiction.
Les données issues de BRENDA ont été particulièrement utiles pour l’étude sur les enzymes
orphelines présentée dans le premier chapitre de cette thèse.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-50-2048.jpg)
![45
RHEA
RHEA [107, 108] est une base de données de réactions non-redondantes annotées manuellement.
Elle est issue d’un projet collaboratif initié par l’EBI (European Bioinformatics Institute) et le
SIB (Swiss Institute of Bioinformatics). Les réactions y sont décrites en utilisant les espèces
chimiques issues de ChEBI (cf. section suivante pour la description de cette ressource), et sont
chimiquement équilibrées au niveau des masses et des charges (les structures chimiques y sont
normalisées au pH 7.3). Des références croisées avec les autres bases de données métaboliques
ainsi que des références bibliographiques sont associées aux réactions quand elles sont
disponibles.
Reactome
Reactome [109] est une base de données publique de réactions et voies métaboliques eucaryotes
(surtout humaines) manuellement validées par des experts. La particularité de cette ressource
consiste dans les très nombreuses références croisées avec les autres bases de données, avec un
accent particulier sur les données d’orthologie entre les espèces eucaryotes.
UniPathway
UniPathway [110] est une ressource pour la représentation et l’annotation de voies métaboliques
totalement validées manuellement par des experts et disponible en libre accès
(http://www.unipathway.org). Elle fournit une représentation explicite des réactions chimiques
spontanées et catalysées par des enzymes ainsi qu’une représentation hiérarchique des voies
métaboliques. Cette hiérarchie utilise des sous-voies linéaires comme des briques basiques pour
reconstruire des voies métaboliques plus grandes et plus complexes. Cette méthode permet ainsi
d’inclure des variants de voies métaboliques espèce-spécifiques plus facilement. Toutes les voies
métaboliques dans UniPathway possèdent des références croisées vers les autres ressources
métaboliques comme KEGG [98] et MetaCyc [111], ainsi que vers les ressources de protéines
comme UniProtKB [18] pour laquelle UniPathway fournit un vocabulaire contrôlé pour
l’annotation des activités enzymatiques et des voies métaboliques.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-51-2048.jpg)
![46
II.1.2 Bases de données de composés chimiques
En plus des ressources contenant plusieurs types d’acteurs du métabolisme, il existe aussi des
bases de données spécialisées uniquement pour les métabolites.
ChEBI
Chemical Entities of Biological Interest [112] (ChEBI) est une base de données non-redondante
de composés chimiques, de groupements chimiques (c’est à dire des parties d’entités chimiques)
et de classes d’entités chimiques annotés manuellement et d’intérêt pour le biologie. Elle est
maintenue par l’EBI. Cette base de données fournit aussi une ontologie chimique qui permet de
décrire les relations entre les molécules et leurs classes chimiques. On n’y trouve que des petites
molécules, donc les molécules (polymères) comme les acides nucléiques, les protéines et les
peptides n’y sont pas inclus. Certaines entrées dans ChEBI peuvent être marquées par trois
étoiles. Cela garantie un niveau de qualité pour l’entrée considérée : la molécule possède un
identifiant unique et stable ainsi qu’un nom unique et non-ambigu. Ces molécules sont aussi
associées à une structure bidimensionnelle, une description, une collection de synonymes incluant
les noms recommandés par l’IUPAC ainsi que des références bibliographiques quand les
molécules ont été citées dans une publication. Cette base de données propose un moteur de
recherche de molécule très performant, on peut y rechercher une molécule par son nom, sa
formule chimique, son identifiant (notamment SMILES ou InChi), sa structure si on dispose d’un
fichier mol, ou même en dessinant la molécule ou une partie de la molécule dans une application
mise à disposition.
PubChem
La base de données de petites molécules PubChem [113] est maintenue par le National Center
for Biotechnology Information (NCBI) aux Etats-Unis d’Amérique. Y sont décrites les molécules
et les complexes moléculaires, des échantillons moléculaires déposés par des chercheurs ainsi que
des molécules issues de bases de données payantes (mais qui ne sont toutefois pas en libre accès).
Le site web inclue un moteur de recherche assez complet ainsi que la description des structures
des molécules.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-52-2048.jpg)
![47
II.2 Classification des activités enzymatiques
Une enzyme est une protéine qui possède le pouvoir de catalyser des transformations chimiques,
c’est à dire qui possède une activité enzymatique. On confond souvent dans le langage courant la
classification des enzymes, qui est en fait une classification des protéines (selon, par exemple, leur
similarité de séquence, leurs domaines ou leur structure), et la classification des activités (ou
réactions) enzymatiques qui, en fait, catalogue les différents types de transformations chimiques
qui peuvent être catalysées par les enzymes.
La classification des objets et des notions est un caractère inhérent de l’espèce humaine. Au-delà
de cet aspect, la classification des réactions enzymatiques est nécessaire pour standardiser leurs
noms, leur type de transformation chimique, les molécules impliquées, les cofacteurs, ainsi que
toutes les autres informations pertinentes. La classification des réactions enzymatiques va de pair
avec la classification des enzymes qui les catalysent, mais dans le premier cas on classifie des
transformations chimiques et dans l’autre des séquences protéiques. Il est, bien sûr, très commun
de donner le nom des réactions aux enzymes, mais ce choix peut porter à confusion lorsqu’une
enzyme catalyse différentes réactions, ou la même réaction est catalysée par des enzymes qui
n’ont pas la même origine évolutive. Les difficultés de partage de travaux scientifiques avant l’ère
d’internet, qui ne sont pas encore totalement résolus, ont entrainé beaucoup de cas où les mêmes
enzymes étaient connues sous des noms différents, et, inversement, le même nom était parfois
donné à des enzymes différentes.
La classification de la Commission Enzymatique (EC) est la seule classification officielle des
activités enzymatiques [114]. Cette commission, crée en 1956 par l’Union Internationale de
Biochimie et de Biologie Moléculaire (IUBMB), a pour but de créer une nomenclature pour
décrire les activités enzymatiques, et résoudre ainsi le problème des réactions aux noms multiples
et de même noms pour des réactions différentes.
Ainsi, le numéro de Commission Enzymatiques (ou EC number) est un système de classification
numérique pour les réactions enzymatiques. Chaque EC number est aussi associé à un nom de
réaction précis.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-53-2048.jpg)
![48
Chaque EC number se compose de lettres « EC » suivies de quatre nombres séparés par des
points. Ces chiffres représentent une classification hiérarchique des activités. Les EC numbers
préliminaires (non-validés par la Commission Enzymatique) sont marqués avec un « n » dans le
quatrième niveau (par exemple EC 1.3.5.n3). Le premier chiffre, qui va de 1 à 6 et qui correspond
à la classe de l’activité enzymatique, définit son type :
1. Oxydoréductases : catalyse des réactions d’oxydation et de réduction ; il s’agit d’un
transfert d’atomes d’hydrogène et d’oxygène ou d’électrons d’une molécule à une autre
2. Transférases : effectuent un transfert d’un groupement fonctionnel d’une molécule à
une autre
3. Hydrolases : permettent la formation de deux produits à partir d’un substrat par
hydrolyse
4. Lyases : effectuent un ajout ou une ablation non-hydrolytique d’un groupement
fonctionnel
5. Isomérases : réarrangement intramoléculaire, c’est à dire des changements de
l’isomérisation au sein d’une seule molécule
6. Ligases : jointure de deux molécules par création d’une nouvelle liaison de type C-O, C-
S, C-N ou C-C
Le deuxième niveau de la classification EC réfère à la sous-classe, qui contient généralement
l’information sur le type des composés chimiques ou de groupements chimiques impliqués (c’est
à dire, par exemple, si la réaction se déroule sur des groupements aldéhyde ou oxo). Le troisième,
représentant la sous-sous-classe de la réaction, spécifie sa nature. Enfin, le quatrième chiffre est
un numéro de série utilisé pour identifier une activité individuelle au sein de la sous-sous-classe
[114] (Figure 12).
Figure 12. Description d’un EC number. Le 1.13.13.54 correspond à une ketosteroide
monooxygenase.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-54-2048.jpg)
![49
Les EC numbers sont répertoriés initialement dans une base de données officielle
(http://www.chem.qmul.ac.uk/iubmb/enzyme) et sont utilisées dans toutes les bases de données
qui contiennent des informations sur les enzymes et les réactions enzymatiques comme la base de
données ENZYME [115] qui fait le lien entre les EC numbers et des séquences de protéines.
Néanmoins cette classification présente quelques limites. La création d’un nouveau EC number
suite à la découverte d’une nouvelle activité enzymatique se fait lors des réunions de la
Commission Enzymatique. Désormais ces réunions se font tous les six mois (avant elles avaient
lieu tous les deux ans), mais ce délai provoque des décalages entre les connaissances accessibles
dans les publications, l’attribution d’un EC number permanent et son intégration dans les bases
de données. L’attribution d’un nouveau EC number officiel est donc manuelle, même si il y a des
méthodes computationnelles (décrites dans les sections suivantes) qui cherchent à automatiser le
processus. Une autre limite de ce système est que les EC numbers ne recouvrent que la moitié
des réactions enzymatiques connues (il y a un peu plus de cinq mille EC numbers au moment de
l’écriture de ce manuscrit et plus de onze mille réactions enzymatiques connues). De plus,
certaines réactions enzymatiques ne correspondent à aucune des six classes de la classification
[116].](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-55-2048.jpg)



![53
II.4 Réseaux métaboliques
Il existe plusieurs catégories de modèles pour décrire le métabolisme [117].
Tout d’abord, les modèles pour l’analyse structurelle du métabolisme. Ces modèles
regroupent principalement les modèles reposant sur la théorie des graphes. Ces derniers sont
basés sur les données qualitatives et sont utilisés pour analyser des propriétés topologiques du
réseau ainsi que les différentes interactions entre les entités qui y sont représentées.
Les modèles pour l’analyse des flux de matière dans le réseau, notamment avec des
techniques comme la « Flux Balance Analysis » [118]. Ce sont la plupart du temps des modèles à
base de contraintes qui prennent en compte la stœchiométrie des réactions afin de prédire la
formation d’une « biomasse » (c’est à dire la survie de la cellule) en fonction des inputs dans le
modèle, qui est une façon de représenter l’environnement de la cellule et surtout ce qui y rentre.
Les modèles pour l’analyse dynamique du métabolisme. Ces modèles sont orientés pour la
simulation du métabolisme et l’étude de ses propriétés dynamiques. Dans ce genre de modèles les
graphes peuvent être utilisés, mais étant donné qu’il s’agit d’étude de la dynamique, des
informations quantitatives sont requises, faisant que les réseaux ne sont que des intermédiaires
dans le processus de modélisation. Ce sont des modèles assez complexes à construire car
nécessitent des données dur la cinétique de chacune des transformations chimiques dans la cellule
[119].
Durant ma thèse je n’ai travaillé que sur les modèles pour l’analyse structurelle du métabolisme.
Ainsi, les sections suivantes seront consacrées à la description de l’utilisation des graphes pour
représenter le métabolisme ainsi qu’aux différentes techniques pour analyser ces graphes.
Le métabolisme est l’ensemble des interactions moléculaires qui se produisent dans un
organisme. Les molécules peuvent être divisées en deux grands types : les métabolites (molécules
souvent de petite taille et qui sont les briques cellulaires) et les enzymes qui catalysent la
transformation des métabolites. Il est commun de représenter le métabolisme d’un organisme,](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-59-2048.jpg)
![54
comme d’autres notions biologiques où l’interaction entre ses éléments est présente, sous forme
d’un réseau. Une belle illustration d’un tel réseau a été empruntée de [120] et est présentée en
Figure 11. La modélisation des réseaux en graphes mathématiques en bioinformatique en facilite
l’analyse. Un graphe est une structure utilisée pour modéliser des relations binaires entre les
objets d’une collection donnée. D’une façon formelle, un graphe G est défini par un couple (V,E)
où V est un ensemble fini de nœuds (ou sommets) et E est une partie de V2
est un ensemble
d’arêtes (en cas de graphe non-orienté) ou d’arcs (en cas de graphe orienté). Ainsi, un réseau
biologique est un ensemble de nœuds et d’arêtes (ou d’arcs si la direction de l’interaction existe
et/ou est connue) étiquetés. Ces étiquettes, ou labels, peuvent être qualitatifs, comme, par
exemple, des identifiants de gènes, de protéines, de réactions, ou quantitatifs, notamment des
poids ou des probabilités de transition sur les nœuds ou les arêtes. Il existe plusieurs types de
réseaux métaboliques, où les nœuds et les liens entre les nœuds représentent des entités
biologiques différentes [121].
II.4.1 Réseau de métabolites
Dans le réseau de métabolites, les nœuds représentent les composés chimiques et deux nœuds
sont liés par une arête si il existe une réaction qui permet la transformation du premier métabolite
en deuxième (c’est à dire si un des métabolites est le substrat et l’autre le produit).
II.4.2 Réseau de réactions
Dans le réseau de réactions, les nœuds représentent les réactions biochimiques (catalysées par des
enzymes ou spontanées) et deux nœuds sont reliés s’il existe un composé chimique produit par la
première réaction substrat de la deuxième.
II.4.3 Réseau d’enzymes
Dans le réseau d’enzymes, les nœuds correspondent aux enzymes. Elles sont reliées par une arête
si elles catalysent des réactions qui ont un composé chimique en commun. Ce type de réseau est](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-60-2048.jpg)

![56
Il existe d’autres façons de représenter le métabolisme sous la forme d’un réseau, mais elles sont
moins fréquemment étudiées et ne seront pas décrites ici.
Tous les métabolites n’ont pas la même fonction et ne sont pas présents en mêmes quantités ou
au même moment dans la cellule. Même si l’étude décrite ici se porte essentiellement sur un
modèle statique du métabolisme, qui représente tous les états possibles connus du métabolisme,
la question des composés ubiquitaires demeure importante.
II.4.5 Composés ubiquitaires et réseaux « petit-monde »
Dans toutes les façons de représenter le métabolisme, décrites précédemment, les réactions et les
métabolites sont considérés comme des acteurs équivalents. Or, comme décrit dans la première
section de ce chapitre, parmi les métabolites on trouve les cofacteurs (par exemple l’ATP et le
NAD), qui, bien que parfois présents dans les équations de réactions ne sont pas leurs
composants principaux. Interviennent, également, dans les réactions, des molécules ubiquitaires
comme par exemple l’eau (H2O), le dioxyde de carbone (CO2) et le dioxygène (O2). Ces
molécules sont souvent en excès dans le milieu cellulaire et elles se retrouvent impliquées dans de
très nombreuses réactions. Si on tient compte de ces composés ubiquitaires dans la modélisation
du métabolisme, on risque de se retrouver avec des réseaux trop connexes (pour un grand
nombre de couples (u, v) de sommets dans ce réseau, il existe un chemin de u à v) et concentrés
autour de ces métabolites. Ceci peut mener à de mauvaises interprétations, car on va notamment
connecter entre eux des réactions et des enzymes qui n’ont rien en commun à part un cofacteur.
Une étude publiée en 2001 [122] montre qu’une modélisation d’un réseau métabolique complet,
où tous les métabolites, mêmes les ubiquitaires, sont présents, exhibe des propriétés de réseaux
« petit monde ». Un réseau dit « petit monde » est un modèle mathématique utilisé pour
représenter des réseaux réels. Le coefficient de clustering de ces réseaux est élevé et la distance
moyenne entre deux nœuds est faible. Par exemple, les réseaux sociaux ont la propriété de petit
monde car dans la majorité des cas, deux nœuds (c’est à dire deux individus), peuvent être reliés
par un très faible nombre de connaissances intermédiaires. Dans le cadre de cette étude de 2001
sur le métabolisme de Escherichia coli, les auteurs montrent que l’on peut relier n’importe quelle
paire de métabolites de ce réseau par un chemin relativement court. Cependant, en se
positionnant du point de vue cellulaire, on ne s’intéresse pas simplement à relier des métabolites
entre eux via n’importe quel chemin possible, mais dans un ordre bien précis ayant un sens](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-62-2048.jpg)
![57
biologique. Comme l’a démontré une étude parue en 2004 [123], d’un point de vue biochimique,
la meilleure alternative est de se concentrer sur les motifs de changements structuraux des
métabolites d’intérêt et sur les flux d’atomes de carbone dans les voies métaboliques. L’auteur
démontre entre autres que le réseau métabolique de Escherichia coli n’est pas un réseau petit
monde, et que l’on a tout intérêt à retirer (ou démarquer) les composés ubiquitaires pour étudier
le métabolisme d’une façon optimale et calculer des chemins réalistes entre les composés.
Plusieurs techniques permettent de traiter ces métabolites gênants. La première consiste à tout
simplement retirer les métabolites les plus fréquents. Il faut toutefois fixer un seuil pour définir à
partir de quel moment un métabolite est « trop » fréquent. On court aussi le risque d’éliminer des
réactions essentielles dans lesquelles des molécules ubiquitaires interviennent comme composants
principaux (la synthèse de l’ATP à partir de l’ADP par exemple, ou la réaction qui permet
d’obtenir du dihydrogène (H2) à partir de deux protons).
Une autre méthode consiste à retirer les métabolites auxiliaires des réactions. Elle est plus
pertinente que la première car elle a l’avantage de ne pas retirer systématiquement les métabolites
ubiquitaires, considérant le contexte dans lequel ceux-là sont employés. Ainsi, en reprenant
l’exemple de la synthèse de l’ATP à partir de l’ADP, où ces métabolites sont les composés
principaux, ils ne seront pas retirés. Par contre, dans une réaction où l’ATP agit comme un
donneur de phosphate et d’énergie, il sera enlevé. La difficulté principale de cette méthode est de
définir systématiquement pour chaque réaction les composés principaux et auxiliaires. Cette
sélection peut se faire automatiquement en utilisant la notion de voie métabolique, où un
composé est principal (ou « primaire ») s’il est produit et consommé dans la voie. Dans la base de
données MetaCyc [124], lorsqu’une réaction fait partie d’une voie métabolique, les composés
chimiques sont marqués comme « primaires » ou « secondaires » selon si ils sont un des substrats
initiaux ou produits finaux, ou décrits comme composé intermédiaire dans la voie métabolique
[125, 126]. La distinction entre les métabolites principaux et auxiliaires peut aussi se faire
manuellement à partir de dessins de cartes métaboliques comme celles de KEGG [102].](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-63-2048.jpg)

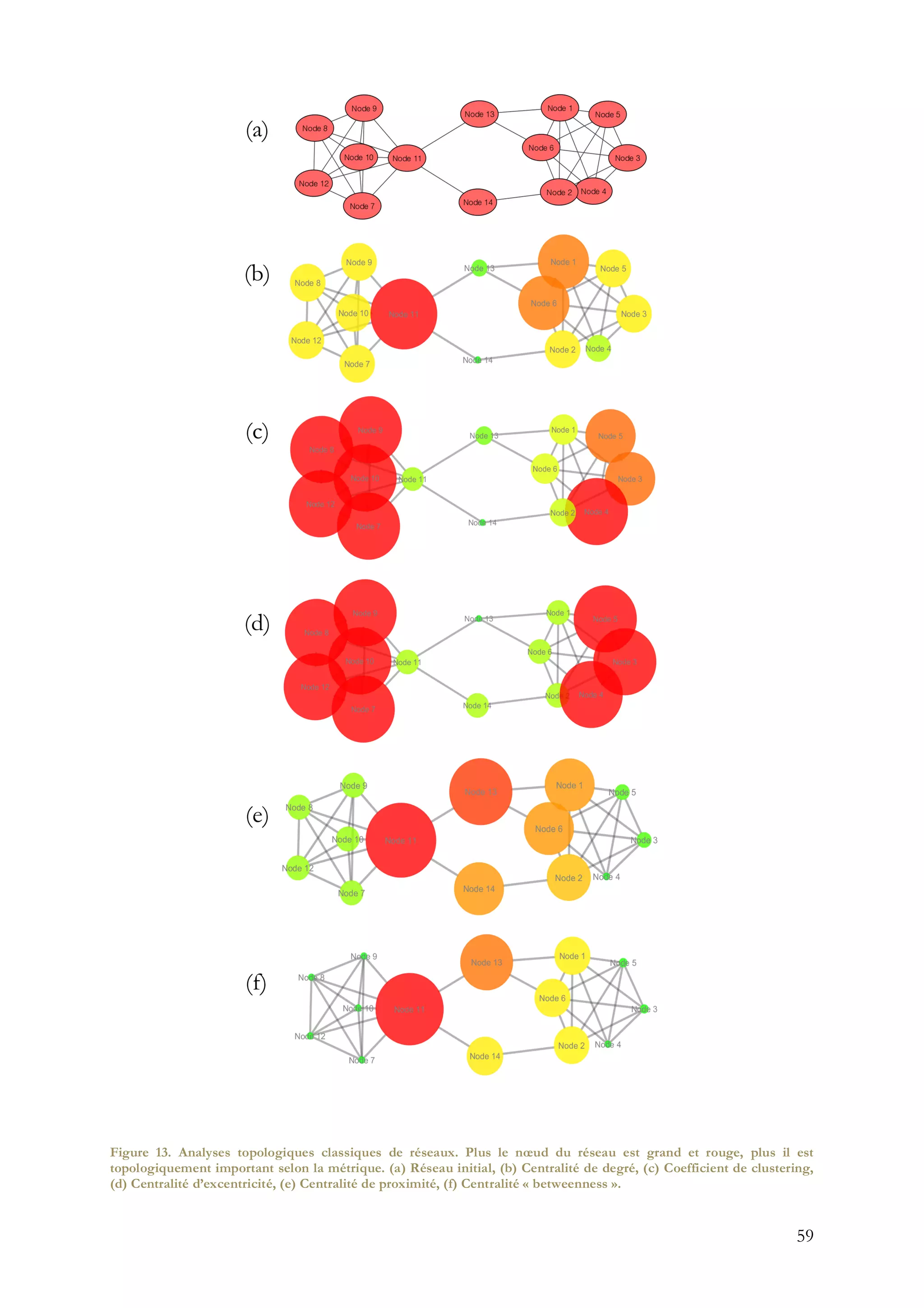
![60
II.5.2 Centralités
Les indices de centralité quantifient le sentiment intuitif que dans la plupart des réseaux certains
nœuds ou arêtes sont plus importants (ou plus centraux) que d’autres. Beaucoup d’indices de
centralité relatifs aux nœuds ont été introduits à partir des années 1940, comme la « degree
centrality » [127] ou la première « feedback centrality » [128]. Depuis, des dizaines de nouveaux
indices de centralités ont été publiés, car toutes les centralités ne représentent pas la même chose,
et il faut adapter cette mesure à chaque application. Ici seront présentés des indices de centralité
les plus classiques, qui ont cependant influencé la plupart des travaux dans ce domaine.
L’importance des nœuds et des arêtes dans un graphe est évaluée selon des valeurs réelles qui y
sont associées, et ces valeurs dépendent uniquement de la structure de ce graphe. Aussi, une
centralité doit rester invariante dans le cas de graphes isomorphiques et automorphiques.
Les indices de centralité peuvent être classés dans plusieurs catégories, décrites dans les sections
qui suivent.
Centralités de distances et de voisinage
Les centralités liées au voisinage des nœuds et aux distances qui les séparent évaluent
l’accessibilité d’un nœud. Dans un réseau, ces mesures permettent de classer les nœuds en
fonction du nombre de leurs voisins et/ou du coût nécessaire pour atteindre tous les autres
nœuds. La centralité basée sur la notion de voisinage est l’indice le plus basique. Les centralités
impliquant la notion de voisinage au sein d’un graphe sont plus complexes, et seront présentées
ensuite.
La « degree centrality », ou la centralité de voisinage, est l’indice de centralité le plus simple. Soit
v un nœud dans un graphe G(E,V) tel que v ∍ V. La « degree centrality » de v notée cD(v) est ce
qui est simplement défini comme le degré d(v) du nœud v si le graphe G n’est pas orienté (Figure
13b). Dans les graphes orientés, deux variantes supplémentaires de la centralité de degré sont
possibles : la « in-degree centrality » ciD(v) = d-
(v) et la « out-degree centrality » coD(v) = d+
(v).
La centralité de degré est une mesure locale car sa valeur pour un nœud donné est simplement
déterminée par le nombre de ses voisins. Les centralités impliquant la notion de distances dans un
graphe sont des mesures globales de centralité. Généralement ces mesures sont assimilées aux
problèmes de localisation des établissements (« Facility Location Problems »), car elles servent à](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-66-2048.jpg)

![62
plus courts chemins pour chaque paire de nœuds. Ceci peut être interprété comme une mesure
dans laquelle un nœud v contrôle la communication entre une paire de nœuds s et t.
Soit 𝛿&' 𝑣 la fraction de tous les plus courts chemins entre s et t qui contiennent le sommet v :
𝛿&' 𝑣 =
𝜎&'(𝑣)
𝜎&'
où 𝜎&' est le nombre total de plus courts chemins entre s et t, tels que 𝑠 ≠ 𝑣 ∈ 𝑉et 𝑡 ≠ 𝑣 ∈ 𝑉. Cette
fraction peut être considérée comme la probabilité que v est impliqué dans la communication
entre s et t. La centralité « betweenness » 𝑐3 𝑣 du nœud v est alors donnée par :
𝑐3 𝑣 = 𝛿&' 𝑣
'*+∈-&*+∈-
La centralité « betweenness » va donc être très élevée pour les nœuds par lesquels passent
beaucoup de chemins du graphe (Figure 13f).
Centralités basées sur les processus aléatoires
Les centralités basées sur les processus aléatoires sont utiles lorsqu’il n’est pas possible de calculer
tous les plus courts chemins dans un graphe. Dans ce type de cas, un modèle de marche aléatoire
fournit une façon alternative de traverser le graphe. Dans une marche aléatoire, une entité
« marche » d’un nœud à un autre, en suivant les arêtes du réseau. En étant sur un des nœuds,
cette entité choisit d’une façon aléatoire une des arêtes (sortantes si le réseau est orienté) du nœud
afin de la suivre jusqu’au nœud suivant. Le nombre de « pas » de cette entité doit être
suffisamment important pour que les résultats de la marche soient significatifs et reproductibles.
Globalement, plus le degré d’un nœud est important, plus l’entité marchant aléatoirement dans le
graphe risque d’y revenir souvent. La marche aléatoire donne aussi de très bons résultats en tant
qu’alternative à la centralité « betweenness », et permet aussi de repérer les nœuds par lesquels
transitent le plus de flux. La centralité de Markov [129], est quand à elle, basée sur le temps
moyen de premier passage (« mean first time passage » - MFPT), qui est le nombre attendu de nœuds
traversés en partant d’un nœud s jusqu’à la première rencontre du nœud t.
Le modèle de surfeur aléatoire, créé pour modéliser le comportement des utilisateurs d’Internet,
introduit un paramètre de « saut » dans la marche aléatoire. Il faut imaginer alors un utilisateur qui
« surfe » sur le Web, en allant d’une page à une autre en cliquant sur des liens hypertextes. Il peut
aussi passer d’une page à une autre sans cliquer sur un lien, parce qu’il connaît, par exemple,
l’adresse de la page par cœur. Il s’agit alors d’un saut car il n’y a probablement pas de lien entre
les deux pages. Ce type de modèle est très utile pour analyser des réseaux biologiques, que l’on](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-68-2048.jpg)
![63
sait « à trous » parce que des informations sont manquantes. Le paramètre de saut permet de
mieux gérer ces nœuds manquants dans le cadre de l’exploration d’un tel réseau.
Feedback
La centralité dite « feedback » (ou de « retour d’information ») est basée sur le principe
d’influence du voisinage : plus un nœud a de voisins, plus il est central, et plus il est central, plus
ses voisins le sont aussi.
Ce type de centralités, plus complexes que celles présentées précédemment, est très utilisé dans
l’analyse de réseaux internet, de réseaux sociaux, et, moins, pour l’instant, dans les réseaux
biologiques. Parmi les centralités « feedback » les plus connues, on retrouve l’indice de Katz
[130], la centralité de vecteurs propres de Bonacich [131], l’indice de Hubbell [132], PageRank
[133] et SALSA [134]. Les notions de « hubs » et « d’autorités » sont très importantes dans ces
centralités. Un hub est un nœud qui pointe vers beaucoup de bonnes autorités, et une autorité
est un nœud pointé par beaucoup de bons hubs.
Figure 14. Centralité PageRank. Plus un nœud est pointé par d’autres nœuds, plus il est influent. Plus un nœud
est influent, plus les nœuds qu’il pointe sont influents.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-69-2048.jpg)
![64
Ici ne sera présentée que la centralité PageRank [133]. Elle a, pendant très longtemps, été un des
ingrédients principaux du célèbre moteur de recherche Google. L’idée principale de cet
algorithme est de marquer une page internet en tenant compte de ses propriétés topologiques
(c’est à dire de sa position dans le réseau). Il s’agit bien d’une centralité feedback, car ici le score
d’une page web dépend du nombre et des scores de ses pages voisines. La Figure 14 représente
bien le fonctionnement de cette centralité. C’est cette centralité qui a été utilisée dans une partie
du travail réalisé pendant la thèse décrite dans ce manuscrit pour calculer l’importance des
réactions les unes par rapport aux autres du point de vue topologique. Cette centralité peut être
considérée comme « semi-globale », car elle permet de calculer des centralités par zones
d’influence de nœuds très autoritaires, qui définissent des régions autour d’eux.
Centralités sur les arêtes
Les centralités décrites dans les sections précédentes définissent l’importance d’un nœud par
rapport aux autres dans un réseau. La plupart de ces centralités peuvent aussi être calculées pour
les arêtes d’un réseau, et ce avec très peu de changements au niveau des algorithmes.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-70-2048.jpg)
![65
II.6 Modularité dans le métabolisme
De la molécule jusqu’à un organisme multicellulaire, toutes les entités biologiques peuvent être
décomposées en modules. La définition la plus simple d’un module est une unité d’un système
pouvant exister ou être décrit indépendamment. De nombreux chercheurs argumentent le fait
que la modularité est présente dans le monde vivant à tous les niveaux [135]. Une molécule est
composée de plusieurs atomes qui ont une existence propre indépendamment de cette molécule,
et peuvent être considérés comme des modules. La molécule elle-même peut être considérée
comme un module d’un complexe moléculaire ou d’un tissu. Les protéines peuvent être
découpées en domaines. Les organes d’un organisme sont les modules de celui-ci, la
transplantation d’organes en est un bon exemple.
En 1999, Hartwell et al. pressentent le fait que la biologie cellulaire va transiter de la simple étude
des molécules indépendantes vers l’étude de modules moléculaires accompagnée de l’essor de la
bioinformatique et de l’ingénierie du vivant [136]. Ils donnent de nombreux exemples de modules
dans les fonctions cellulaires, comme le mécanisme de synthèse des protéines, la réplication de
l’ADN, la glycolyse ou encore les processus de mitose permettant la distribution correcte des
chromosomes. Ces modules ont pu être reconstitués/reproduits in vitro ce qui est déjà un très bon
critère de validation en faveur de l’hypothèse de modularité.
Le métabolisme peut aussi être considéré comme modulaire. Les voies métaboliques, telles que
définies précédemment, peuvent être considérées comme des modules biochimiques du
métabolisme. On peut aussi retrouver des petits modules topologiques dans le réseau
métaboliques d’un organisme donné, pouvant être combinés d’une façon hiérarchique dans des
unités plus grandes [137]. L’identification de modules conservés dans le métabolisme est au cœur
de cette thèse. Les théories et les méthodes existantes sont présentées dans la quatrième section
de cet état de l’art, et celles développées lors de ce travail sont décrites dans le deuxième chapitre.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-71-2048.jpg)

![67
III.1 Annotation fonctionnelle des génomes
L’annotation fonctionnelle consiste principalement à assigner des fonctions aux séquences
protéiques codées par les gènes, notamment, pour les enzymes, à décrire leurs activités
enzymatiques et les voies métaboliques associées.
On peut distinguer trois différents niveaux de fonctions :
• les fonctions moléculaires, qui capturent le rôle biochimique ou structural de la
protéine
• les fonctions cellulaires, décrivant le rôle de la protéine dans un processus cellulaire de
plus haut niveau (implication dans une voie métabolique, par exemple, pour des enzymes)
• les fonctions phénotypiques, associant une protéine à un niveau systémique comme la
croissance cellulaire ou la virulence. Dans ce cas, la fonction moléculaire de la protéine
n’est pas forcément connue mais une modification/délétion du gène codant la protéine
impacte un processus cellulaire observable expérimentalement.
La description des fonctions se fait préférentiellement via du vocabulaire contrôlé et des
ontologies (comme les EC numbers, décrits dans la section II de ce chapitre, pour les enzymes),
même si beaucoup sont aussi décrites en texte libre par les experts annotateurs.
Pour les gènes codant des enzymes, le lien entre les gènes, les protéines qu’ils encodent et les
réactions que ces protéines catalysent est souvent retrouvé dans la littérature sous l’appellation
« association GPR » (Gene – Protein - Reaction) [138]. Ce mode de représentation permet faire la
distinction entre les isoenzymes (plusieurs gènes codant des enzymes différentes catalysant la
même réaction) et les enzymes multimériques et/ou multifonctionnelles (plusieurs gènes codant
des protéines formant un complexe protéique pour catalyser une ou plusieurs réactions). Avec ce
formalisme, il y a une connexion évidente entre la présence/absence d’un gène et la
présence/absence d’une fonction (c’est à dire d’une réaction) réalisée par la protéine.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-73-2048.jpg)
![68
III.1.1 Liens phylogénétiques et similarité de séquences
III.1.1.1 Liens phylogénétiques entre les gènes
Historiquement, l’homologie était utilisée par les naturalistes pour décrire des liens évolutifs
entre différentes espèces de plantes ou d’animaux. Des similarités entre la forme, la couleur et
l’utilisation des membres ou des organes permettait aux scientifiques d’identifier ces liens : on
comparait par exemple la structure des os du bras humain, de l’aile d’un oiseau et de la nageoire
d’un dauphin, qu’on disait homologues. Des traits dont l’utilité et la forme se ressemblent, mais
ne proviennent pas d’une même origine évolutive (comme l’aile d’un oiseau et celle d’un papillon)
sont dits analogues.
Ces notions sont aussi applicables en génétique. Deux gènes (ou produits de gènes) de deux
organismes différents sont dits homologues lorsqu’ils se ressemblent suffisamment du point de
vue moléculaire et qu’il y a des preuves suffisantes que les deux gènes ont évolué à partir d’un
même gène présent dans un ancêtre commun aux deux organismes. Des gènes analogues ont
des fonctions moléculaires similaires mais ont évolué séparément et ne présentent pas de
similarité de séquence notable. La notion d’homologie est utilisée pour l’annotation fonctionnelle
et suppose que des gènes homologues codent pour des protéines ayant des fonctions similaires ce
qui par de nombreux exemples peut se révéler inexact [11]. Il faut souligner ici que l’homologie
est un concept binaire, soit deux gènes sont homologues soit ils ne le sont pas. Il existe plusieurs
catégories d’homologie qui correspondent à des chemins évolutifs différents ayant mené à des
pressions de sélection différentes sur les gènes.
Un événement de spéciation est un évènement complexe qui mène à l’émergence de deux
nouvelles espèces à partir d’une seule espèce ancestrale. En raison de l’ascendance commune, la
plupart des gènes des deux nouvelles espèces possèdent des gènes ancestraux communs. Les
gènes ayant un ancêtre commun avec lequel ils n’ont été séparés que par des événements de
spéciations sont des gènes orthologues (Figure 15). Les gènes orthologues subissent
généralement la même pression de sélection dans leurs organismes respectifs, assurant ainsi la
conservation de leur fonction.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-74-2048.jpg)
![69
Les évènements de duplication de gènes entrainent la création de deux copies d’un même gène
au sein d’un même génome. Ces gènes peuvent évoluer sous différentes pressions de sélection,
car un seul des deux est nécessaire d’une façon vitale à la survie de l’organisme. Les gènes dans
cette configuration sont dits paralogues (Figure 15) et vu la pression sélective plus faible ou
différente entre les deux copies, la fonction n’est pas considérée comme systématiquement
conservée, même si la fonction peut demeurer similaire (des spécificités de substrats différentes
par exemple pour des enzymes).
Comme les événements de spéciation et de duplication de gènes ne sont pas linéaires dans le
temps et produisent des configurations assez complexes, deux termes supplémentaires pour
décrire la paralogie ont été introduits. Lorsque la duplication de gènes est ancienne (c’est à dire
qu’elle est survenue avant un évènement de spéciation), les gènes sont dits « out-paralogues ».
On les considère alors suffisamment éloignés l’un de l’autre pour avoir des fonctions différentes.
Si l’évènement de duplication est récent (c’est à dire qu’il n’y a pas eu a priori d’évènement de
spéciation après cette duplication), les gènes sont dits « in-paralogues » et sont considérés
comme étant suffisamment proches pour avoir une même fonction ou une fonction fortement
similaire (Figure 15).
L’évolution des génomes ne se fait pas uniquement dans le sens vertical, où les parents seuls
transmettent l’ensemble de l’information génétique à leur descendance. En effet, dans la nature, il
existe aussi un mode horizontal de transfert d’information génétique, où des morceaux d’ADN
sont transférés entre organismes de deux espèces différentes. Ce type de transmission géniques
survient la plupart du temps entre organisme unicellulaires et est particulièrement fréquent chez
les bactéries (même si des cas de transfert de gènes concernant les organismes pluricellulaires
complexes ont aussi été mis en évidence [139]). Les gènes dans cette configuration se nomment
xénologues (Figure 15).](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-75-2048.jpg)
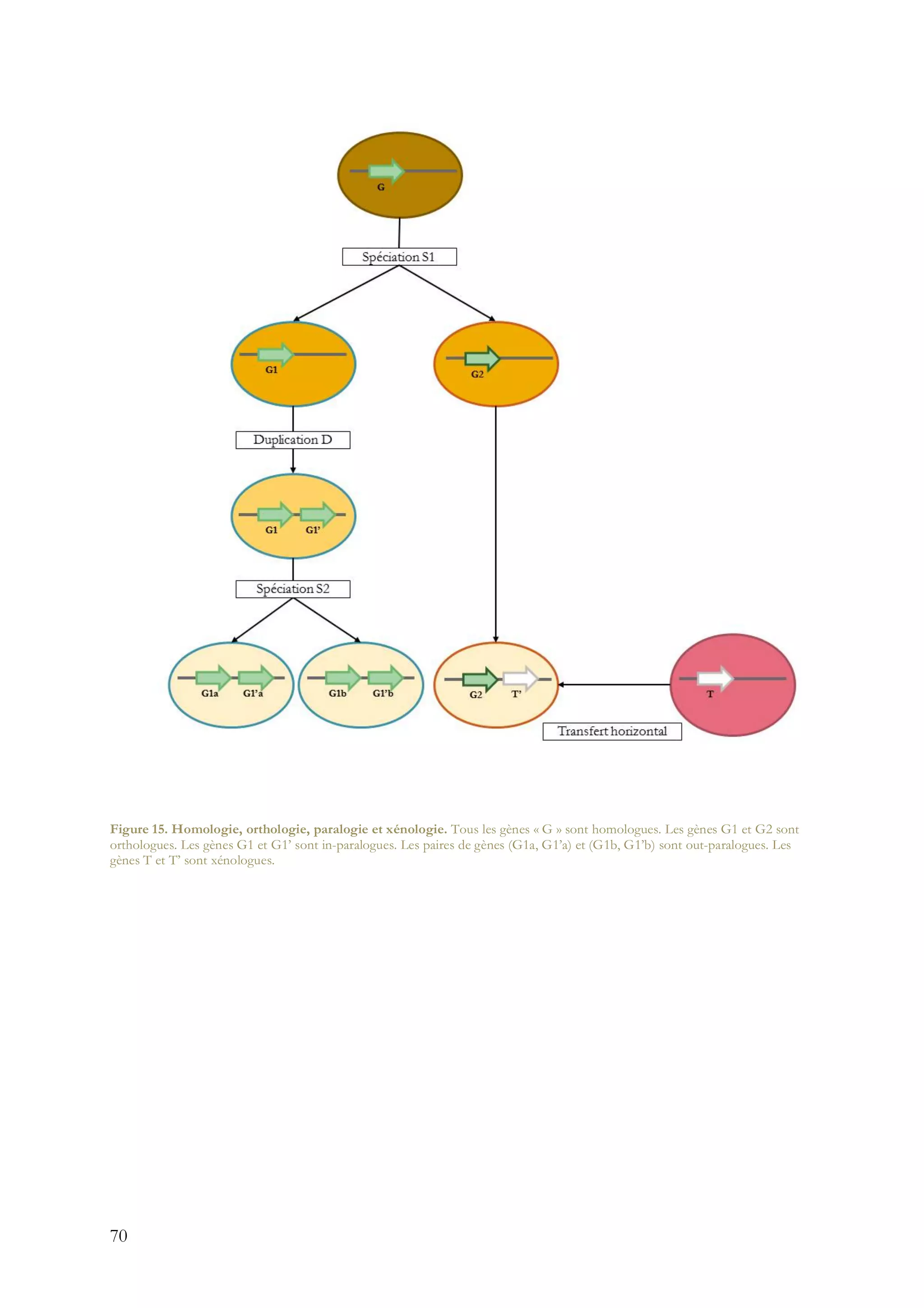
![71
III.1.1.2 Annotation fonctionnelle basée sur la similarité de séquences
La façon la plus classique et la plus rapide d’associer une fonction biologique à une séquence est
basée sur la comparaison des séquences des nouvelles protéines aux séquences de protéines déjà
connues. Ceci provient de l’hypothèse que des protéines homologues possèdent des fonctions
similaires et la même fonction si elles sont orthologues. La comparaison des protéines se fait via
la similarité de leurs séquences en acides aminés et, si elles sont suffisamment proches,
l’annotation est transférée de la protéine connue vers la nouvelle. La similarité entre les séquences
est calculée en utilisant des programmes comme FASTA [15] et BLAST [13] (PSI-BLAST [140]
en particulier pour les séquences d’acides aminés). Le problème de cette méthode provient du fait
que des protéines ayant des séquences relativement proches peuvent avoir des fonctions
différentes. Beaucoup d’annotations dans les bases de données publiques ne sont inférées qu’en
utilisant cette technique seule, ce qui conduit à beaucoup d’annotations erronées [11]. Par
exemple, toujours d’après [11], plus de 90% de certaines familles d’enolase ne sont pas
correctement annotées dans la plupart des bases de données publiques. Une étude récente [141]
qui a été réalisée pour estimer la sur-annotation par similarité de séquence dans les génomes
procaryotes, montre notamment que toutes les méthodes utilisées actuellement ont tendance à
beaucoup sur-prédire la fonction des protéines. Pour éviter les annotations erronées, la
comparaison de séquences protéiques peut (et doit) être associées à d’autres techniques
d’annotation fonctionnelle.
III.1.2 La base de données de protéines UniProt
L’entrepôt principal à l’heure actuelle de séquences protéiques est la base de données UniProt
[18]. Cette base de données est maintenue par le UniProt Consortium, constitué en 2002 et
regroupant les ressources et expertises de l’EBI (European Bioinformatics Institute) basé dans le
comté de Cambridge au Royaume-Uni, de PIR (Protein Information Ressource) basé à
Georgetown aux Etats-Unis d’Amérique et du SIB (Swiss Institute of Bioinformatics) en Suisse.
En plus d’être un entrepôt pour les séquences protéiques qui peuvent être déposées par les
équipes scientifiques du monde entier, UniProt propose diverses annotations qui peuvent y être
associées, telles que les fonctions, les ontologies, les références bibliographiques liées à la
séquence, le découpage de la protéine en domaines ou encore les liens vers d’autres séquences ou](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-77-2048.jpg)
![72
des bases de données plus spécialisées (cross-references). Cette énorme ressource est constituée de
plusieurs modules dont les objectifs scientifiques sont différents.
La partie de UniProt la plus connue et la plus utilisée est UniProt Knowledge Base (UniProtKB),
elle-même constituée de deux parties, SwissProt et TrEMBL. SwissProt est une base de données
de séquences de protéines de haute qualité d’annotation dont une partie est expertisée
manuellement. Le nombre d’entrées dans cette resource représente cependant moins de 1% du
total de séquences de UniProtKB. TrEMBL est une base de données dont les protéines sont
obtenues par la traduction automatique de séquences codantes (CDS) de l’ENA et dont
l’annotation est réalisée d’une façon automatique. Jusqu’en avril 2015, UniProtKB contenait
l’intégralité des protéines issues des projets de séquençage des génomes. Ces protéomes (i.e., un
protéome correspond à l’ensemble des séquences protéiques d’un organisme qui sont prédites à
partir de son génome) représentaient une quantité d’information trop importante (près de 100
millions d’entrées) pour être gérée convenablement par le consortium. Depuis la mise à jour du
27 mai 2015, UniProtKB ne contient plus que des protéines de protéomes dits « de référence » :
un seul protéome de référence est gardé parmi les groupes de protéomes se ressemblant entre
eux à plus de 90% dans leur contenu en séquence
(http://www.uniprot.org/help/2015/04/01/release). Le nombre d’entrées est ainsi redescendu à
50 millions.
La base de données UniParc est une collection regroupant l’ensemble des séquences de protéines
d’une manière non-redondante et sert également d’archive pour les anciennes séquences. Depuis
la mise à jour mentionnée ci-dessus, elle contient aussi toutes les protéines des protéomes qui ne
sont plus intégrés dans UniProtKB. Cependant, cette base de données ne contient pas
d’annotations sur les séquences.
III.1.3 Domaines fonctionnels et familles de protéines
Une des façons d’améliorer la prédiction de fonction des protéines est d’étudier leur composition
en domaines structuraux et/ou fonctionnels. L’hypothèse guidant cette approche est que certains
domaines sont des unités fonctionnelles, et ceux-ci sont très conservés au cours de l’évolution.
Souvent, une protéine est constituée de plusieurs domaines, un seul domaine principal peut ainsi
porter la fonction moléculaire ou, alors, c’est la combinaison de ces domaines qui permettra de
réaliser la fonction. Des méthodes comme MKDOM [142], PRIAM [143] et Pfam [144] ont été](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-78-2048.jpg)
![73
développées pour découper les protéines en domaines, trouver comment les identifier (parfois,
quelques acides aminés placés à des endroits spécifiques suffisent pour déterminer un domaine et
une fonction enzymatique) et y associer une activité biologique. La ressource InterPro [145, 146]
permet de regrouper et hiérarchiser ces différentes méthodes au sein de mêmes entrées
caractérisées par des signatures correspondant à des résultats des méthodes intégrées dans
InterPro. Certaines méthodes, comme EnzML ou Pfam2GO [19], se basent sur la composition
en domaines d’une séquence et leurs combinaisons pour identifier au mieux la fonction
biologique.
Pfam
La base de données de Familles de Protéines (Pfam) [147] est basée sur la recherche de domaines
conservés dans les séquences protéiques. La présence d’un domaine donné (ou d’un ensemble de
domaines aussi appelé « architecture ») est utilisée pour définir les familles de protéines. Les
domaines sont détectés dans les protéines en se basant sur des alignements multiples de
séquences qui sont utilisés ensuite pour construire des profils de modèles de Markov cachés
(HMM) représentant ces domaines. Ces profils permettent d’assigner à d’autres séquences de
protéines un ou plusieurs domaines Pfam via le logiciel HMMER [148]. Il existe deux types de
familles de protéines dans Pfam : les familles Pfam-A qui sont établies manuellement par des
experts et les familles Pfam-B dont les profils sont générées automatiquement et pas encore
validés. Cette section Pfam-B n’est pour l’instant plus maintenue (la dernière mise à jour date de
mai 2013). Les domaines Pfam ont une bonne couverture sur UniProtKB : 80% des protéines
sont associées à au moins un domaine.
Les domaines dont la fonction est encore inconnue sont désignés comme des DUFs (Domains of
Unknown Function) et représentent environ 25% des familles Pfam [144].
Il faut remarquer que dans Pfam, la taille des différentes familles de protéines est très variable,
ainsi que le niveau de résolution des domaines : certains domaines vont représenter toute une
famille d’enzyme (par exemple, PF00171 regroupe les enzymes de la famille des aldéhyde
déshydrogénases), d’autre vont décrire un sous-domaine structural d’une enzyme particulière (par
exemple, PF00712 représente la partie N-terminal de la chaîne beta de la DNA polymérase III).
Cette granularité variable pose donc des problèmes dans l’utilisation directe de Pfam pour prédire
des fonctions.
Néanmoins, les familles Pfam ont été beaucoup utilisées dans le cadre de cette thèse, notamment
pour relier des protéines de fonction inconnue à des transformations chimiques.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-79-2048.jpg)
![74
InterPro
InterPro [146] est un entrepôt intégratif pour plusieurs méthodes de définition de signatures
(domaines, motifs, familles) de protéines. En plus d’intégrer diverses informations sur les familles
de protéines, les domaines et les sites fonctionnels, InterPro propose un outil, InterProScan qui
permet de prédire les signatures issues de différentes sources à partir d’une séquence.
PRIAM
La méthode PRIAM [143] est dédiée à l’identification des gènes codant pour des enzymes et leurs
activités enzymatiques en utilisant des règles combinant des « profils » spécifiques à l’activité
enzymatique construits à partir de collections de séquences enzymatiques connues. PRIAM utilise
la classification en EC numbers pour les activités enzymatiques et les protéines annotées de
SwissProt pour construire les profils PSSM (Position‐Specific Scoring Matrices) de référence via le
programme MKDOM [142]. Ces profils sont comparables à des domaines protéiques. PRIAM
permet ainsi d’assigner des fonctions aux nouvelles séquences en se basant sur la détection de
similarité de profils via le logiciel PSI-BLAST [140].
Cette approche a été utilisée dans l’étude sur les enzymes orphelines (Chapitre I de cette thèse)
pour trouver des séquences candidates pour les enzymes orphelines de séquences.
Il existe aussi d’autres ressources permettant de classifier les protéines en familles de protéines
équivalogues (i.e. protéines homologues ayant leurs fonctions conservées), comme FIGFam
[149], TIGRFam [150], FunFams [151] ou encore HAMAP [21], mais elles ne seront pas
abordées ici.
III.1.4 Contexte génomique pour l’annotation fonctionnelle
Les différentes méthodes de contexte génomique sont décrites plus tard dans cette section. Elles
peuvent être utilisées dans le cadre de l’annotation fonctionnelle. Par exemple, chez les
procaryotes, les gènes impliqués dans une même fonction cellulaire ont tendance à être proches
sur le chromosome, voire être co-transcrits sous l’influence d’un même promoteur (on appelle](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-80-2048.jpg)
![75
cette structure « opéron »). La conservation de cette co-localisation au cours de l’évolution
s’appelle la synténie. Cette information de contexte d’un gène peut être utilisée pour y inférer
une fonction [152, 153].
L’information sur la fusion de deux gènes au cours de l’évolution peut aussi être utilisée pour
relier fonctionnellement des gènes homologues non fusionnés [154, 155].
Le phénomène de coévolution des protéines repose sur la tendance observée des protéines
fonctionnellement reliées à évoluer de façon corrélée. En prenant un grand nombre de génomes,
un profil de présence/absence dans chacun d’entre eux est établi pour chaque protéine. Ce profil
correspond généralement à un vecteur booléen, où « vrai » signifie la présence d’un homologue
de la protéine dans le génome correspondant, et « faux » son absence. Les protéines sont alors
classées en fonction de la similarité de profils phylogénétiques et leurs fonctions déterminées en
conséquence [156].
III.1.5 Analyse de la structure des protéines
L’étude de la conformation structurale des protéines, ainsi que la comparaison de leurs structures
est aussi une méthode d’annotation fonctionnelle. Bien que prometteuse, elle ne s’est pas encore
révélée suffisamment efficace pour être appliquée à grande échelle, mais il s’agit d’un domaine
relativement nouveau et dynamique. Il se pourrait donc que dans un avenir relativement proche
cette méthode prouvera son efficacité [157]. En effet, la structure d’une enzyme, et
particulièrement de sa poche catalytique (l’endroit où la transformation chimique des molécules
est catalysée), est directement liée à la fonction qu’elle effectue.
En théorie, des enzymes n’ayant aucune homologie de séquence mais présentant le même
arrangement en 3D des acides aminés dans les poches catalytiques, ont de forte chance de
catalyser la même réaction. C’est par exemple le cas de la subtilisine et de la chymotrypsine [158].
Ainsi, les logiciels de comparaison de sites actifs vont rechercher les motifs tridimensionnels
connus (c’est à dire répertoriés dans des bases de données de sites actifs) se trouvant dans la
protéine de fonction inconnue.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-81-2048.jpg)
![76
Cependant, la plupart des logiciels ne vérifient pas que le motif tridimensionnel trouvé se trouve
bien dans la poche (ce motif peut aussi être enfoui dans la protéine et non-accessible aux
métabolites). Selon les enzymes étudiées, les logiciels ne peuvent repérer qu’un motif de trois
résidus. Celui-ci n’est souvent pas assez spécifique d’une activité donnée, comme par exemple la
triade catalytique Serine-Histidine-Aspartate, qui est retrouvée dans un très grand nombre
d’hydrolases et de transférases. D’autres logiciels (comme, par exemple, SALSAs [159] et ASMC
[160]) comparent les structures des sites actifs de familles d’enzyme et recherche le motif
tridimensionnel consensus de sous-familles potentielles. Ces méthodes révèlent ainsi la diversité
des réactions possibles au sein d’une famille et par conséquent aide à affiner l’annotation
fonctionnelle et spécifique des enzymes.
Il est aussi possible de faire de la prédiction ab initio de compatibilité d’une poche catalytique et
d’un métabolite d’un point de vue géométrique et énergétique, grâce à l’amarrage moléculaire
(aussi appelée « docking » moléculaire). C’est en testant in silico plusieurs milliers de métabolites
dans une poche catalytique d’une protéine de fonction inconnue par amarrage, que, par exemple,
Fan et al. ont découvert une activité pterin deaminase [161]. La limite la plus importante des
méthodes basées sur la comparaison des structures protéiques est le manque de structures
résolues expérimentalement (par cristallographie aux rayons X ou par résonance magnétique
nucléaire) qui sont couteuses et assez longues à obtenir. La modélisation d’une structure par
homologie apparaît donc comme un bon compromis. Aussi, la prédiction d’activité grâce à
l’amarrage moléculaire est limitée par le nombre restreint de métabolites répertoriés dans les
banques.
En combinant les approches de comparaison de séquences, de contexte génomique et de
structure, la qualité de l’annotation fonctionnelle automatique peut être largement améliorée
[162]. Cette efficacité a été démontrée récemment par Bastard et al. [163] qui, grâce à une
approche combinant plusieurs méthodes informatiques et des résultats expérimentaux de criblage
enzymatique ont réussi à annoter la famille Pfam de protéines de fonction inconnue DUF849
comme étant des enzymes réalisant le clivage de β-keto acides (3-keto acides). Ils ont aussi pu
définir des sous-familles pour lesquelles ils ont associés 14 nouvelles réactions enzymatiques
spécifiques.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-82-2048.jpg)
![77
III.1.6 Systèmes d’annotation à base de règles
Des méthodes combinant plusieurs approches d’annotation fonctionnelle d’une façon
« intelligente » ont aussi été développées. Appelées « systèmes à base de règles », ce sont des
méthodes d’annotation fonctionnelle automatique basées sur plusieurs méthodes d’annotation
fonctionnelle et d’un système de décision. La méthode publiée en 2008 par Azé et al. [164], par
exemple, considère l’annotation d’une protéine en termes de hiérarchie fonctionnelle, et propose
un ensemble de règles qui prédisent la ou les classes fonctionnelles pour une protéine.
Des méthodes plus simples ont été développées au sein du consortium UniProt. Les règles
(HAMAP et UniRule) sont basées sur des propriétés simples des protéines (longueur de la
séquence en acides aminés, par exemple), ainsi que sur leur composition en domaines et leur
appartenance taxonomique, et servent à annoter automatiquement les protéines de la base de
données UniProtKB [21].
Une autre méthode, INFAES, publiée en 2015 par Xavier et al. [165] est un système expert à base
de règles qui mime le raisonnement d’un être humain pour l’inférence d’une annotation
fonctionnelle. Ce système intègre les connaissances sur la biologie ainsi que les heuristiques sur
l’utilisation des méthodes automatiques d’annotation fonctionnelle. Très souple, il permet une
intégration continue de nouvelles connaissances, et est aussi très performant (il a montré
notamment de bons résultats en comparaison avec les résultats du concours CAFA [166] qui
rassemble des équipes du monde entier travaillant sur les problèmes liés à l’annotation
fonctionnelle).
III.1.7 Systèmes d’annotation communautaire
En dehors des différentes technologies automatisant l’annotation fonctionnelle de grandes
quantités de données, l’annotation fonctionnelle des gènes et des protéines devrait aussi être
gérée par la communauté scientifique. Ainsi, lorsqu’un chercheur remarque une erreur
d’annotation dans les bases de données publiques, l’édition de l’annotation devrait être facilitée.
Certains auteurs [11, 144, 167, 168] proposent notamment un système d’éditions expertes basé
sur le modèle de Wikipédia pour permettre à la communauté d’écrire et de rectifier les
annotations. Ce travail de curation nécessite des environnements informatiques intégrés, appelés](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-83-2048.jpg)
![78
plateformes d’annotation (comme Microscope [169] ou SEED [170] par exemple) qui fournissent
de puissantes interfaces graphiques pour aider les experts à nettoyer ou à compléter les
annotations générées par les méthodes automatiques.
III.1.8 Cas des protéines multifonctionnelles
Les protéines multifonctionnelles sont des enzymes capables de jouer plusieurs rôles dans le
métabolisme en catalysant des réactions (parfois très) différentes. Plusieurs sortes de
multifonctionnalité sont connues actuellement. Certaines enzymes sont capables de catalyser une
même réaction chimique sur plusieurs composés chimiques différents, c’est la promiscuité de
métabolites [56]. D’autres enzymes sont capables de catalyser différentes transformations
chimiques en utilisant le même site catalytique, c’est la promiscuité de réactions [171]. On peut
aussi avoir des protéines constituées de deux ou plus domaines fonctionnels avec différents sites
actifs [172]. L’association de plusieurs domaines au sein d’une protéine, qui résulte généralement
d’un événement de fusion de gènes au cours de l’évolution, peut notamment faciliter la
conversion des substrats et la régulation des flux métaboliques. Il existe aussi des protéines
multifonctionnelles assez particulières, appelées « moonlighting enzymes » [44, 45]. Ces protéines ont
la capacité de changer d’activité enzymatique en fonction des conditions environnementales, de
leur localisation cellulaire, du type de la cellule (dans le cas d’organismes multicellulaires), des
concentrations en ligands ou en cofacteurs, ou en formant des complexes avec d’autres protéines.
Il existe une base de données dédiée aux enzymes multifonctionnelles répertoriant leurs
différents types : MultitaskProtDB [173].
Les enzymes multifonctionnelles sont assez difficiles à annoter, car la plupart des méthodes ne
cherchent à associer qu’une seule fonction à une séquence. De plus, hormis les enzymes multi-
domaines, la recherche des autres fonctions est assez complexe et nécessite souvent des données
expérimentales.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-84-2048.jpg)

![80
III.2.1 Clusters de gènes
III.2.1.1 Opérons
Un opéron est une unité génomique contenant un groupe de gènes co-localisés sur le même brin
d’ADN et souvent associés à une même fonction cellulaire sous contrôle d’un même promoteur
(Figure 16a). Les gènes d’un opéron sont co-transcrits en un seul ARN messager, appelé ARN
polycistronique. Environ 60% des gènes chez les procaryotes sont regroupés en opérons [174].
Chez les eucaryotes, les opérons sont beaucoup plus rares : des transcrits polycistroniques ont
tout de même été observés, par exemple chez le nématode et chez la drosophile [175, 176]. Les
opérons sont souvent conservés entre différentes espèces, même s’il peut y avoir des
réarrangements génomiques (gains, pertes, duplications de gènes) [177].
Il a été remarqué que les gènes d’un opéron sont fréquemment impliqués dans une même
fonction cellulaire. Par exemple, un opéron peut contenir des gènes codant des enzymes
catalysant des réactions d’une même voie métabolique. Il est donc intéressant d’explorer
l’information contenue dans les opérons pour prédire de nouveaux processus biologiques comme
des voies métaboliques et améliorer l’annotation des protéines.
Méthodes de prédiction des opérons
Une première hypothèse pouvant être formulée pour la détection d’opérons est que la distance
entre les gènes d’un même opéron est plus faible qu’entre les gènes appartenant à des unités de
transcription différentes, puisqu’ils sont co-transcrits et que la présence de divers signaux de
transcription n’est pas nécessaire. Cette hypothèse a été confirmée en étudiant les opérons
connus de Escherichia coli, rassemblés dans la base de données RegulonDB [178, 179]. La distance
intergénique est le critère le plus informatif dans la prédiction des opérons [180–182]. Ainsi, la
prédiction des opérons peut être vue comme la recherche des limites des unités de transcription,
où la distance entre les gènes adjacents est faible et il n’y a pas de gènes sur le brin opposé de
l’ADN. Les groupes de gènes correspondant à cette description sont appelés des directons.
Une autre hypothèse de base est que les opérons vont avoir tendance à être conservés dans les
organismes procaryotes. Des résultats d’investigation en génomique comparative [183, 184]
montrent que les gènes adjacents sur le même brin d’ADN ont tendance à rester proches dans les
génomes d’espèces différentes, contrairement aux gènes sur les brins opposés. Ainsi, la](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-86-2048.jpg)
![81
comparaison de la conservation de gènes entre différents organismes permet une prédiction de
grande qualité des opérons dont on ne dispose pas de données expérimentales sur les unités de
transcription [183].
Figure 16. Clusters de gènes.
(a) Structure d’un opéron procaryote. La séquence régulatrice contrôle l’expression des multiples régions codantes (en
rouge). Le promoteur, l’opérateur et l’enhancer (en jaune) régulent la transcription de cette région en ARNm. Les
régions non-traduites de l’ARNm (en bleu), régulent la traduction en protéines. Image adaptée de Wikipedia
(https://en.wikipedia.org/wiki/Operon).
(b) Groupes de synténie conservée entre les génomes A et B. Ces groupes de synténie sont détectés avec un algorithme
utilisant le concept de multigraphe [190,191], qui permet l’association de plusieurs gènes homologues entre les
génomes, ainsi que la détection d’évènements de fusion, duplication, insertion, inversion et réarrangement de gènes.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-87-2048.jpg)
![82
Des méthodes de prédiction des opérons plus complexes et très divers ont été développées ces
dernières années. On pourra notamment citer des méthodes intégrant des données
expérimentales comme des données d’expression via des micro-puces à ADN [185], ou du
séquençage d’ARN [186], des méthodes utilisant l’apprentissage artificiel comme des réseaux
bayésiens [187] ainsi que l’utilisation des algorithmes génétiques [188].
Une approche simple basée sur la première hypothèse présentée ici a été appliquée dans le cadre
de cette thèse pour prédire des opérons potentiels (directons) d’une façon systématique dans un
grand nombre de génomes procaryotes. Cette analyse sera présentée dans le chapitre 3 de ce
manuscrit.
III.2.1.2 Synténies conservées
Du point de vue de la génomique, la synténie est la présence simultanée (et éventuellement dans
le même ordre) sur le même chromosome de deux ou plusieurs gènes dans plusieurs organismes
(Figure 16b). Elle permet de conclure qu’une région génomique dans deux ou plusieurs
organismes provient d’une seule région génomique ancestrale. Les régions synténiques peuvent
appartenir à des organismes différents, et sont donc dérivés d’évènements de spéciation, ou au
même organisme et ont pour origine des évènements de duplication (on pourra donner l’exemple
de polyploïdie – duplication de chromosomes entiers – chez les plantes). Un bloc synténique (ou
groupe de synténie, ou synton) comprend l’ensemble des gènes en synténie.
Les analyses de synténie sont une façon pratique de comparer les organismes et d’étudier
l’évolution des génomes. Elles permettent de détecter la conservation de fonctions biologiques
[189, 190], d’identifier des réarrangements de génomes [191], aider à l’annotation fonctionnelle
des génomes [152] et même prédire des erreurs d’assemblage de génomes après le séquençage.
Il existe un grand nombre d’outils de détection et de visualisation de synténie entre les génomes,
on citera, notamment, cette méthode basée sur le recherche de composantes connexes maximales
dans un multigraphe [192, 193], Cinteny [191] et Proteny [194]. Les blocs synténiques sont
facilement visibles avec les outils de visualisation de génomes les plus simples, comme Artemis
Comparison Tool [195], ou intégrés dans des plateformes pour une aide à l’annotation, comme
dans MicroScope [169].](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-88-2048.jpg)
![83
III.2.2 Profils phylogénétiques
Un profil phylogénétique (parfois aussi appelé « profil phylogénomique ») est un vecteur
décrivant la présence/absence de familles de gènes dans un ensemble d’organismes. La
comparaison des vecteurs de présence/absence de gènes entre différents organismes permet
d’établir une dépendance fonctionnelle entre les gènes : deux gènes impliqués dans un même
processus biologique ont beaucoup de chance d’être soit tous les deux présents, soit tous les deux
absents dans un organisme, la perte de l’un d’entre eux pouvant entrainer la perturbation, voire la
perte, du processus. En 1999, Pellegrini et. al [156] étaient les premiers à proposer l’utilisation des
profils phylogénétiques pour mesurer cette dépendance inter-génique. Beaucoup de variantes de
la méthode ont été proposées depuis, utilisant notamment des mesures différentes de similarité
de gènes ou des vecteurs pondérés à la place de vecteurs booléens. Les profils phylogénétiques
sont principalement utilisés comme des indicateurs de la co-évolution des gènes plutôt que
comme des outils directs pour l’annotation fonctionnelle, même s’ils peuvent l’améliorer.
III.2.3 Rosetta stone (fusions/fissions de gènes)
La fusion de gènes permet la création de gènes hybrides à partir de deux gènes initialement
séparés. Ce mécanisme joue un rôle important dans l’évolution de l’architecture génique. En
effet, lorsque ce genre d’altération génique n’est pas létale pour l’organisme, la fusion de gènes
entraine l’apparition de nouvelles fonctions ou une augmentation d’efficacité des fonctions
métaboliques déjà existantes (via le « metabolic channeling » par exemple [196]), en ajoutant un
module peptidique pour former une protéine multimérique. C’est aussi un bon indice par rapport
à l’implication des deux gènes dans une même fonction cellulaire dans différents organismes. Les
évènements de fission de gènes, où un gène ancestral constitué de plusieurs domaines est séparé
en deux gènes fonctionnels sont beaucoup plus rares [197]. On appelle « Rosetta stone » un
triplet constitué d’un gène fusionné dans un génome et de deux gènes séparés et homologues au
premier dans un autre génome, car ce genre de structure permet de « déchiffrer » des interactions
possibles entre les produits de ces gènes [198, 199]. Beaucoup d’autres travaux ont inclus les
évènements de fusion et de fission de gènes dans les analyses de génomique comparative [197,
200, 201]. L’analyse de ces évènements fait désormais partie des méthodes de référence dans
l’analyse du contexte génomique.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-89-2048.jpg)
![84
III.3 Reconstruction de réseaux et modèles métaboliques
L’information génomique disponible à partir du séquençage d’un génome complet permet la
reconstruction d’un réseau métabolique entier et spécifique de l’organisme. Comme nous l’avons
vu dans les sections précédentes, il peut y avoir différents types de réseaux métaboliques, centrés
sur les métabolites, les réactions ou les enzymes, orientés ou non, contenant des arêtes simples ou
des hyperarêtes.
Pour reconstruire le réseau métabolique d’un organisme donné, son génome doit être
fonctionnellement annoté. Ceci signifie que chaque gène (lorsque c’est possible) doit être associé
à une fonction biologique, plus précisément, à une activité enzymatique pour les gènes codant des
enzymes. On peut ainsi déduire toutes les capacités métaboliques de l’organisme en traduisant les
activités enzymatiques prédites en réactions pouvant être catalysées dans l’organisme. Les autres
données ‘omiques’ sur l’organisme, comme le transcriptome (données qualitatives et quantitatives
sur les ARNs), le protéome (données qualitatives et quantitatives sur les protéines), le
métabolome (données qualitatives et quantitatives sur les métabolites) et le bibliome
(informations issues de la littérature) permettent d’améliorer la qualité du réseau construit [202].
La reconstruction de réseaux métaboliques à partir de génomes complets comprend quatre
grandes étapes fondamentales : la reconstruction automatique à partir des annotations
fonctionnelles des gènes, la curation de cette reconstruction, sa conversion en un modèle
informatique et l’intégration d’autres données ‘omiques’ pour affiner le modèle. Ces différentes
étapes, ainsi que les données utilisées, sont représentées sur la Figure 17 (adaptée d’après [202]).
Etape 1 : Reconstruction automatisée à partir d’un génome complet
Le point de départ pour toutes les reconstructions métaboliques est le génome annoté d’un
organisme donné. Les données d’annotation fonctionnelle peuvent être trouvées dans des
banques généralistes de génomes (Genbank ou EMBL), des banques généralistes de protéines
(UniProtKB) ou dans des ressources spécialisées pour un organisme (comme Ecogene [203] pour
E. coli K-12 ou la « Pseudomonas Genome Database » [204] pour les Pseudomonas). Elles
peuvent également être issues de plateformes d’annotation ou être produites localement en
utilisant différentes méthodes d’annotation fonctionnelle. Ces multiples sources d’annotations ne](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-90-2048.jpg)
![85
facilitent pas la reconstruction. De plus, la plupart du temps, seuls les EC numbers, avec leurs
limites (cf. section « Classification des activités enzymatiques »), sont disponibles pour décrire les
activités enzymatiques avec un vocabulaire contrôlé. A partir de ces fonctions prédites, un
ensemble de réactions enzymatiques potentiellement présentes dans l’organisme est projeté sur
des voies métaboliques de référence qui peuvent être issues de bases de données généralistes
(comme KEGG [102] ou MetaCyc [91]) ou spécifiques d’une espèce (EcoCyc [205] pour E. coli
par exemple). Cette reconstruction par homologie suppose que les voies métaboliques sont
conservées entre les organismes et a pour but de prédire si une voie métabolique existe ou non
dans un organisme étant donné un ensemble d’activités enzymatiques prédites. Quelques
méthodes facilitant cette reconstruction automatique de réseaux métaboliques existent, on pourra
notamment citer PathwayTools [94] et SEED [170].
Ces méthodes sont relativement rapides mais une annotation fonctionnelle correcte des protéines
est cruciale pour une reconstruction de bonne qualité. Pour établir correctement les associations
GPR (cf. début de section), une difficulté supplémentaire est d’être capable de faire la différence
entre des protéines qui sont des isoenzymes et des protéines formant un complexe protéique. Les
cas d’enzymes multifonctionnelles et de promiscuité sont également difficiles à appréhender pour
définir un bon ensemble de réactions pouvant être catalysés dans un organisme. Cette étape
permet d’obtenir une structure appelée GENRE (GEnome-scale Network REconstruction).
Etape 2 : Curation de la reconstruction automatique
Bien que l’extraction automatisée de réactions métaboliques des bases de données à partir des
annotations fonctionnelles permet d’obtenir une collection initiale de réactions biochimiques que
l’organisme est capable de réaliser, elle ne permet pas d’établir certaines caractéristiques
organisme-spécifiques, comme des réactions ou des voies métaboliques non représentées dans les
bases de données généralistes ou la localisation subcellulaire des enzymes. Ce type d’informations
requiert la connaissance experte de l’organisme ; ainsi, le réseau métabolique reconstruit
automatiquement nécessite une curation manuelle. Celle-ci est nécessaire pour ajouter et corriger
les informations que les procédures automatisées manquent ou placent mal. Cette étape est
souvent assez laborieuse et peut prendre beaucoup de temps, nécessitant la recherche
d’informations spécifiques dans la littérature spécialisée ou directement auprès des spécialistes.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-91-2048.jpg)
![86
Etape 3 : Conversion du réseau métabolique reconstruit en modèle
informatique
Avant qu’une reconstruction puisse être utilisée pour les calculs, notamment pour les calculs de
capacités physiologiques de l’organisme, la conversion de cette reconstruction en une
représentation mathématique doit être faite. Cette conversion traduit un GENRE en un modèle
mathématique à l’échelle d’un génome – GEM (GEnome-scale Model). La représentation d’un
réseau dans un format mathématique permet le déploiement d’un large éventail d’outils de calcul
pour analyser les propriétés de celui-ci. Ces outils de calculs permettent l’évaluation des
propriétés systémiques du réseau, ainsi que des fonctions que le réseau peut accomplir sous des
Figure 17. Etapes et données pour la reconstruction d’un réseau métabolique à partir d’un génome complet (image extraite
de Feist et al. [202]). La reconstruction de modèles métaboliques à partir de génomes complets peut être divisée en quatre phases
majeures successives. Une des caractéristiques de ce processus de reconstruction est son raffinement itératif dirigé par les données
expérimentales des trois dernières phases. Pour chaque phase, des types de données spécifiques sont nécessaires. Ces données peuvent
être très différentes en fonction de la phase, allant des données à haut débit (comme les données de métabolomique ou de phénomique)
aux données issues d’analyses détaillées caractérisant des composants individuels (par exemple, données biochimiques pour une réaction
particulière). Les modèles intermédiaires générés par chaque phase de la reconstruction peuvent être utilisés et appliqués pour répondre à
une quantité croissante de questions, mais c’est bien la version finale du modèle qui a le plus d’applications.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-92-2048.jpg)
![87
contraintes physico-chimiques. Cette approche a mené au développement des méthodes de
reconstruction et d’analyses à base de contraintes, dont la boite à outils COBRA [206] est
l’exemple le plus connu. Ce type d’approches permet d’étudier notamment le comportement de
l’organisme dans des conditions de croissance spécifiques ou des conditions environnementales
particulières.
Etape 4 : Utilisation de modèles métaboliques et intégration des données
‘omiques’
Les données ‘omiques’ qui évaluent un très grand nombre d’interactions au travers de différentes
conditions peuvent être utilisées pour raffiner et développer le contenu métabolique d’un modèle.
Ces types de comparaisons et d’analyses permettent d’améliorer la compréhension du
fonctionnement de l’organisme dans différentes conditions environnementales. On pourra
notamment donner l’exemple de l’utilisation de données de croissance cellulaire sur des milieux
définis via la technologie Biolog (http://www.biolog.com), ou des données issues de la
métabolomique et de dosages enzymatiques in vitro systématiques qui ont mené à la découverte de
nouvelles réactions et voies métaboliques comme par exemple dans cette étude de Saito et al.
[207]. La confrontation de données expérimentales aux prédictions du modèle permet ainsi de
valider le modèle. En cas d’incohérences, le réseau métabolique reconstruit doit être amélioré (cf.
étape 2).
Malgré les avancées grandioses des connaissances sur l’organisation et le fonctionnement des
organismes vivants, beaucoup de parts d’ombre demeurent. Ces lacunes dans les connaissances
actuelles sur le métabolisme sont présentées dans la section suivante.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-93-2048.jpg)
![88
III.4 Lacunes dans les connaissances enzymatiques
Les connaissances sur les enzymes et les activités enzymatiques sont très diversifiées et produites
par des scientifiques issus de domaines différents. La caractérisation des activités enzymatiques
est plutôt du ressort de la (bio)chimie avec par exemple des applications en biocatalyse, alors que
l’étude des protéines enzymatiques et des gènes qui les encodent implique plutôt la biologie
moléculaire, la protéomique, la génomique et la biologie structurale. La multiplicité des approches
et des représentations des données, les difficultés de communication entre les différents
domaines scientifiques, ainsi que les limites technologiques font qu’il existe des lacunes dans les
connaissances. Dans cette partie, seront présentés le problème des activités enzymatiques
« orphelines » de séquences, les causes et les conséquences de ce problème.
En 2004, Richard J. Roberts a lancé un appel pour une action communautaire pour l’annotation
de gènes de fonction inconnue dans les génomes microbiens [208]. La même année, Peter Karp
proposa une approche complémentaire, aussi via un appel à la communauté scientifique, qui
consistait à essayer d’associer au moins une séquence protéique à chaque activité enzymatique
biochimiquement caractérisée [1]. Il a proposé de combiner les approches bioinformatiques et
des stratégies « de paillasse » pour identifier et valider des protéines candidates issues de données
génomiques. Il a été notamment mis en avant que parmi les 3736 activités enzymatiques (EC
numbers) listées dans la base de données ENZYME [115], 1437 (c’est à dire 38%) d’entre elles
n’avaient aucune séquence protéique associée, même en combinant différentes sources
d’annotation de protéines (SwissProt [23], TrEMBL [18], PIR (Protein Information Ressource
[209]), CMR (Comprehensive Microbial Ressource [210]) et BioCyc [124]). Comme la
classification EC n’inclue pas toutes les activités enzymatiques connues et que certaines
annotations protéiques ne sont pas associées avec les bons EC numbers, Peter Karp a estimé
alors que cette estimation pouvait être biaisée. Ces activités enzymatiques sans séquences
associées ont été baptisées « activités enzymatiques orphelines de séquences » (ou « enzymes
orphelines » pour faire court) en 2005 [211] par Olivier Lespinet et Bernard Labedan.
Ces activités enzymatiques orphelines sont répertoriées dans la base de données dédiée,
ORENZA (http://www.orenza.u-psud.fr) [4], qui existe depuis 2006, ainsi que, depuis peu dans](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-94-2048.jpg)
![89
le « Orphan Enzymes Project » (http://www.orphanenzymes.org) initié par Alexander Shearer
[212, 213].
Au sein de la classification EC, les activités orphelines se répartissent plutôt uniformément dans
les 6 grandes classes : il y en a le moins parmi les ligases (21%) et le plus parmi les
oxydoréductases et les transférases (respectivement 37% et 38%) [214].
Elles ont tendance à provenir des organismes autres que les 10 organismes modèles les plus
étudiés (37% des enzymes orphelines proviennent des organismes modèles contre 63% des
organismes non-modèles [214]) Par exemple, seulement 4% des enzymes orphelines ont pour
organisme source initiale Escherichia coli. Par ailleurs, 75% d’activités annotées avec des EC
numbers incomplets (où il manque un ou plusieurs digits) sont orphelines de séquence [214].
L’existence des enzymes orphelines pause ainsi un problème dans les analyses du métabolisme.
En effet, parmi les 124 voies métaboliques bien connues en 2006 issues de KEGG [102] et de
MetaCyc [91], seulement 24 ne contiennent aucune enzyme orpheline [2].
Les activités enzymatiques orphelines peuvent être classifiées comme « locales » et « globales »
[215]. Les enzymes orphelines globales, celles décrites précédemment, n’ont aucune séquence
représentative associée dans aucun des organismes. En revanche, les enzymes orphelines
locales représentent des activités pour lesquelles on n’a pas de séquence représentative associée
dans un organisme ou clade (groupe d’organismes) d’intérêt, bien qu’une ou plusieurs séquences
protéiques catalysant la réaction peuvent être connues dans d’autres organismes.
L’existence de ces enzymes, dont les protéines qui les catalysent sont inconnues, pose notamment
un gros problème lors de l’annotation fonctionnelle des séquences et de la reconstruction de
réseaux métaboliques à partir de génomes complets. Aussi, les enzymes orphelines de séquences
pourraient être importantes pour des applications industrielles et pharmacologiques [3] (synthèse
de nouveaux médicaments par exemple), c’est pourquoi il peut être intéressant de découvrir les
protéines qui les réalisent, pour pouvoir les maitriser et les utiliser. Dans la section suivante sont
décrites différentes méthodes permettant d’explorer le métabolisme et pour, notamment, associer
des séquences aux activités enzymatiques orphelines.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-95-2048.jpg)

![91
Il existe un grand nombre de représentations de métabolites (cf section I de cette partie du
manuscrit) et autant de façons de décrire les réactions qui les transforment. Dans les sections
suivantes, sera présentée une sélection de méthodes de représentation, de classification et
d’utilisation des réactions enzymatiques.
IV.1.2 Reaction Pairs et Reaction Class de KEGG
KEGG [98] est une ressource très complète sur les génomes et sur le métabolisme au sein de
laquelle un grand nombre de méthodes sont développées.
Chacune des réactions présentes dans la base de données KEGG est découpée en un ensemble
de paires substrats-produits. Pour chaque paire, les molécules sont comparées entre elles avec une
représentation en motifs RDM ayant pour but de déterminer les atomes du centre réactionnel
(atomes R), les atomes adjacents au centre réactionnel (atomes D) et les atomes qui changent au
cours de la réaction (atomes M) [216]. Cette comparaison est basée sur une représentation de
sous-structures de molécules appelée KCF/KCF-S [42] qui rassemble 68 types d’atomes avec une
distinction particulière des groupements chimiques fonctionnels et des environnements
atomiques. La signature d’une réaction en motif RDM (Figure 18) pour chaque paire de
molécules est nommée RPair. Les RPairs sont utilisés pour calculer des classes de réactions
(RClass), qui rassemblent les réactions partageant les mêmes RPair. Les RClass sont ensuite
utilisés pour prédire un EC number pour de nouvelles réactions (deux algorithmes ont été
développés dans ce cadre, MUCHA [217] et E-zyme [218]).
Figure 18. Motifs RDM permettant de décrire les changements atomiques dans les molécules au cours d’une réaction
(image extraite de Kotera et al. [216]). Ces motifs sont utilisés dans la base de données KEGG. Les types KEGG d’atomes
permettent l’identification de l’endroit de la molécule où se déroule la réaction ainsi que les changement opérés au cours de celle-ci.
Ces atomes permettent de définir un motif de conversion chimique. Trois types d’atomes sont définis : les atomes du centre
réactionnel (atomes R), les atome qui sont impliqués dans la différence de structure (atomes D) et les atomes qui ne changent pas
au cours de la réaction (atomes M).](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-97-2048.jpg)
![92
IV.1.3 Signatures moléculaires de réactions (RMS)
Comme évoqué précédemment (cf. section I.2.1), la signature moléculaire (MS) [41] permet une
représentation canonique des molécules en sous-graphes circonvoisins d’un atome dans une
structure moléculaire jusqu’à un diamètre prédéfini, aussi appelé hauteur. Ces sous-graphes,
encodés en format SMILES, sont calculés pour chaque atome de la molécule pour un diamètre
donné.
Une signature moléculaire pour une réaction métabolique (« RMS » pour Reaction Molecular
Signature) est obtenue par la différence entre les signatures des produits et des substrats. Ce
système d’encodage des réactions en signatures permet d’avoir plus ou moins de précisions sur la
sous-structure chimique autour des atomes impliqués dans la transformation en jouant sur la
hauteur des signatures moléculaires (les hauteurs élevées permettent une plus grande précision,
les plus basses étant moins précises). Le processus de création des RMS est illustré en Figure 19
(extraite de l’article de Carbonell et. al [29]).
Les RMS ont été utilisées lors du travail décrit dans cette thèse pour encoder et regrouper les
réactions de la base de données MetaCyc.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-98-2048.jpg)
![93
Figure 19. Processus de création d’une signature moléculaire de réaction (RMS) (image extraite de Carbonell et al. [29]).
(A) Processus de calcul d’une signature moléculaire pour le 6-aminohexanate. La première étape est le calcul de la signature pour
chacun des atomes. Dans l’exemple présenté, la signature atomique du carbone du groupement carboxyle est calculée jusqu’à la
hauteur 2. A hauteur 0 (en bleu), le graphe moléculaire est enraciné à l’atome n’est représenté que par cet atome. A hauteur 1 (en
vert) est donnée la représentation canonique de l’atome de carbone central et de ses voisins immédiats. Le processus est répété
pour les hauteurs suivantes : à hauteur 2 (en orange) ce sont les voisins des voisins qui sont pris en compte. Les signatures des
atomes sont calculées pour tous les atomes de la molécule.
(B) Processus de création d’une signature moléculaire pour la réaction 6-aminohexanoate hydrolase. La signature de réaction
contient la différence entre les signatures des produits et des substrats. Ici, la RMS a été calculée pour la hauteur 1.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-99-2048.jpg)
![94
IV.1.4 Cartographie des atomes (Atom Mapping)
L’atom mapping (« cartographie des atomes » en français) d’une réaction chimique est la
bijection des atomes réactants vers les atomes des produits qui spécifie le terminus de chaque
atome réactant. Concrètement, il s’agit de suivre le devenir de chaque atome des molécules
impliquées dans la réaction. Historiquement, plusieurs méthodes, souvent basées sur
l’isomorphisme de graphes, ont été utilisées pour calculer les atom mappings, mais ici une seule
sera présentée, celle qui est implémentée dans MetaCyc [97].
L’atom mapping de MetaCyc est basé sur une métrique minimisant les distances d’édition entre
atomes (MWED) et qui s’avère être très efficace. Concrètement, des poids sont assignés à
presque toutes les liaisons atomiques de tous les substrats et les produits de la réaction. Ces poids
représentent la tendance des liaisons atomiques à être rompues, créées ou à changer de type (la
transformation d’une liaison simple en liaison double par exemple). Un cout basé sur ces poids
est associé à chaque type de changement de liaison. La distance d’édition de l’atom mapping est la
somme des coûts. Ce type de modélisation de réactions chimiques s’avère assez efficace et peu
coûteux en terme de complexité computationnelle (Figure 20).
IV.1.5 EC-BLAST et autres méthodes basées sur la comparaison
de fingerprints moléculaires
EC-BLAST [219] est un algorithme et un outil pour la recherche de similarités quantitatives entre
les réactions enzymatiques. Les résultats de cette méthode sont disponibles sur un site web
(http://www.ebi.ac.uk/thornton-srv/software/rbl). Il y a trois niveaux de similarité possibles qui
sont calculés suivant : les changements de liaisons entre les atomes des molécules impliquées dans
Figure 20. Cartographe des atomes pour une réaction de monooxygénation de type Baeyer-Villiger issue de MetaCyc.
L’atome 70 de la molécule de dioxygène est inséré dans le lien carbone-carbone des atomes 17 et 19.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-100-2048.jpg)
![95
une réaction, les changements au niveau du centre réactionnel et la similarité de structure des
molécules. EC-BLAST utilise l’atom mapping pour calculer les changements de liaisons et permet
également d’aider à classifier les activités enzymatiques en EC numbers. Le fonctionnement de
EC-BLAST est décrit en Figure 21. Les trois niveaux de similarité sont décrits par des vecteurs
booléens, communément appelés « fingerprints ».
Une autre méthode de comparaison de réactions biochimiques basée sur les fingerprints est
RxnSim [220]. Elle utilise des signatures moléculaires des participants d’une réaction encodées
dans un ensemble de vecteurs binaires. Cet ensemble est construit en utilisant trois méthodes
pour capturer les signatures moléculaires à des niveaux différents de granularité. L’avantage de
cette méthode est de comparer les réactions sur la base des similarités entre les substrats et les
produits en plus de leur transformation chimique.
L’avantage des méthodes basées sur les fingerprints est que ceux-ci sont relativement faciles à
construire à partir des données structurales des molécules impliquées dans les réactions, et qu’il
est computationnellement facile de les comparer entre eux. Leur plus gros désavantage réside
dans leur limitation descriptive, car il faut définir chaque caractéristique qu’une molécule
biologique pourrait avoir pour la marquer ensuite comme présente ou absente dans la molécule
Figure 21. Description du workflow EC-BLAST (image extraite de Rahman et al. [219]).](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-101-2048.jpg)
![96
considérée, et cette description de toutes les possibilités peut être assez fastidieuse et requiert une
expertise humaine importante.
IV.1.6 Mécanisme réactionnel enzymatique
Le concept de similarité des réactions est surtout étudié du point de vue des transformations
chimiques associées aux réactions, mais pas en termes du mécanisme réactionnel. La méthode de
mesure quantitative de similarité de réactions basée sur leur mécanisme explicite a été publiée en
2007 par O’Boyle et al. [221] et c’est la seule réellement efficace pour le moment. La différence
entre une transformation chimique d’une réaction et son mécanisme est que le mécanisme
présente en plus l’ordre des modifications des liaisons interatomiques, étape par étape. Deux
approches complémentaires sont utilisées par cette méthode pour mesurer la similarité entre les
étapes réactionnelles : une approche basée sur des fingerprints (représentés par des vecteurs) qui
incorporent les informations sur chaque étape mécanistique, et une approche basée uniquement
sur l’ordre des modifications des liaisons atomiques. La similarité globale de deux mécanismes
réactionnels est calculée en utilisant un algorithme d’alignement simple sur les fingerprints.
Il existe une base de données de mécanismes enzymatiques qui classifie les enzymes selon le
mécanisme utilisé pour catalyser les réactions – MACiE [222]. Une analyse de cette base de
données, en utilisant les résultats de classification des réactions selon leur mécanisme, a permis
une identification de mécanismes chimiques convergents (enzymes d’origines évolutives
différentes réalisant des transformations avec le même mécanisme). Cette analyse a d’ailleurs
souligné que la classification EC ne couvre pas la similarité de transformation chimique [221].
IV.1.7 Description des réactions avec MOLMAP
Le descripteur MOLMAP (molecular maps of atom-level properties) [223] est relativement récent et
semble de plus en plus utilisé pour décrire les réactions. Ce descripteur moléculaire permet de
définir les types des liaisons covalentes par rapport à leurs propriétés physico-chimiques et
topologiques. Ainsi, le descripteur MOLMAP d’une molécule représente les types de liaisons
dans cette molécule. Par ailleurs, le descripteur MOLMAP d’une réaction, de la même façon que](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-102-2048.jpg)
![97
les RMS [41], se définit comme la différence des MOLMAPs des produits et des substrats de la
réaction. Il permet d’encoder d’une façon numérique les changements dans les liaisons
interatomiques au cours de la réaction. Ce système permet ainsi de classifier des réactions sur la
base des modifications de liaisons qu’elles engendrent dans les molécules participantes. Ce
système a notamment été utilisé pour assigner d’une façon automatisée des EC numbers aux
réactions enzymatiques [224].
IV.2 Méthodes pour détecter des protéines pour les
enzymes orphelines
Le problème des enzymes orphelines pourrait être en partie résolu avec des techniques de fouille
de littérature, car seulement 80% de ces activités seraient vraiment orphelines de séquence [5], les
20% restantes ont leur séquences manquantes à cause du décalage dans les connaissances dans les
bases de données publiques et d’erreurs d’annotation.
Il existe plusieurs façons d’identifier des protéines candidates pour les enzymes vraiment
orphelines de séquences.
L’hypothèse que des enzymes participant à une même processus biologiques (i.e. une voie
métabolique) partagent une histoire évolutive commune, est à l’origine de l’utilisation des profils
phylogénétiques pour trouver des séquences candidates pour les enzymes orphelines [6]. La
méthode des profils phylogénétiques se base sur le fait que des protéines, ayant des vecteurs de
présence/absence similaires dans un ensemble d’espèces, sont souvent fonctionnellement liées
[156]. Ainsi, si deux protéines co-occurrent fréquemment dans des génomes, qu’une d’entre elles
est de fonction inconnue et l’autre catalyse une réaction métabolique voisine d’une réaction
orpheline, il y a de fortes chances que la protéine de fonction inconnue catalyse en fait la réaction
orpheline.
Une autre approche, basée également sur le contexte génomique, est de combiner les contextes
de co-localisation chromosomique et métaboliques [225, 226]. En effet, et c’est particulièrement
le cas chez les bactéries et archées, des gènes participant à un même processus cellulaire sont](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-103-2048.jpg)
![98
souvent co-localisés sur le chromosome dans des structures en opérons. En détectant des
métabolons [193], c’est à dire des groupes de gènes co-localisés codant pour certains des enzymes
catalysant des réactions voisines dans le réseau métabolique (i.e. liées entre elles par des
métabolites), on peut réussir, là aussi, à associer des gènes de fonction peu ou pas connue à des
gaps métaboliques (c’est à dire à des activités orphelines).
Un des problèmes de ces méthodes utilisant le contexte métabolique vient du fait que
généralement, dans les voies métaboliques, les réactions voisines de réactions associées à une
activité enzymatique orpheline sont elles aussi orphelines. Par conséquent, ces méthodes donnent
de bons résultats uniquement dans les cas où très peu d’enzymes orphelines sont présentes dans
une voie métabolique et qu’elles sont entourées d’enzymes non-orphelines.
Les données expérimentales post-génomiques, telles que celles issues de la transcriptomique
quantitative, de la protéomique, les structures tridimensionnelles ou encore les données de
phénotypes de croissance, peuvent aussi s’avérer très utiles pour associer des séquences aux
activités enzymatiques orphelines [7]. Il est notamment important de prendre en compte
simultanément les informations dans les organismes procaryotes et eucaryotes, pour trouver des
enzymes homologues partagées dans les différents règnes, ce qui pourrait aussi être utile dans
l’association de séquences à des activités enzymatiques orphelines locales [7].
Il n’existe donc pas encore de méthode parfaite qui permettrait de retrouver des séquences
protéiques candidates pour l’intégralité des enzymes orphelines mais, en combinant différentes
méthodes et approches présentées dans cette section, un certain nombre d’entre elles ont déjà été
résolues.
Dans le premier chapitre de cette thèse sont présentées différentes statistiques sur les enzymes
orphelines, de nouvelles perspectives pour l’association de séquences à ces activités et de
nouvelles définitions dans les lacunes sur les connaissances enzymatiques.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-104-2048.jpg)
![99
IV.3 Recherche de chemins et de motifs dans le réseau
métabolique
La représentation mathématique du métabolisme sous la forme d’un réseau facilite sa
manipulation et son exploration. Cette exploration peut notamment consister à rechercher des
voies métaboliques dans le réseau, ou encore des structures biologiquement importantes qui sont
indépendantes du reste (modules) ou répétées (motifs). Dans cette section, seront présentées les
différentes méthodes de recherche de telles structures.
IV.3.1 Recherche de voies métaboliques
Trois approches sont possibles pour trouver de nouvelles voies métaboliques :
- la rechercher de sous-graphe ou de chemins dans le réseau métabolique
- la rétrobiosynthèse
- l’alignement de voies métaboliques qui utilise la similarité d’enchainements de réactions
entre des voies connues et de nouvelles voies potentielles.
Les trois approches sont présentées dans les sections suivantes.
IV.3.1.1 Recherche de sous-graphes ou chemins
L’analyse de données variées, expérimentales (e.g. transcriptomique, protéomique) ou non (e.g.
profils phylogénétiques, les opérons ou les groupes de synténie), permet la détection de groupes
de gènes/protéines dont les fonctions peuvent être reliées. Ces fonctions (i.e. activités
enzymatiques) peuvent ainsi être projetées sur le réseau métabolique de l’organisme étudié pour
déterminer des sous-graphes connexes pouvant correspondre à des voies métaboliques [227,
228]. Il existe plusieurs variations dans ces méthodes, en fonction du type des données
disponibles (données sur les gènes/protéines, ou sur les métabolites) et des approches
informatiques (utilisation d’hypergraphes ou de graphes pondérés).](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-105-2048.jpg)
![100
La pondération d’un réseau métabolique en fonction du degré de ses nœuds et la recherche de
chemins de score le plus bas est une méthode qui s’est montrée efficace pour la découverte de
voies métaboliques dans un réseau biparti [229]. La comparaison des chemins trouvés grâce à
cette technique pour la dégradation de l’arginine avec les voies métaboliques réelles en a prouvé la
cohérence.
Les modes élémentaires, introduits en 1999 par Schuster [230], sont aussi une bonne technique
pour trouver des voies métaboliques dans un réseau. Il s’agit de déterminer un ensemble minimal
de réactions pouvant opérer à l’état stable du système et où toutes les réactions irréversibles
procèdent dans la direction appropriée. Pour être qualifiée de mode élémentaire, une voie
métabolique doit respecter l’équilibre stœchiométrique et ne doit pas pouvoir être décomposée en
sous-chemins plus petits respectant cette propriété.
L’atom tracking (le suivi des atomes) est aussi un bon moyen de trouver des voies métaboliques
cohérentes dans un réseau métabolique. Des algorithmes [231, 232], étant donné un métabolite
de départ et un d’arrivée, recherchent des chemins basés sur la conservation des atomes en
suivant leurs échanges dans un réseau métaboliques. Ces méthodes permettent de trouver des
voies métaboliques linéaires, mais aussi ramifiées.
Ces méthodes de recherche de sous-graphes ou chemins dans un réseau métabolique se limitent
uniquement à l’univers des réactions décrites dans le réseau et ne peuvent donc pas trouver des
voies métaboliques composées de nouvelles réactions.
IV.3.1.2 Rétro(bio)synthèse
La biosynthèse est un processus biologique dont les étapes sont catalysées par des enzymes,
transformant les substrats dans des produits complexes. C’est un processus naturel faisant partie
du métabolisme. L’émergence de l’ingénierie métabolique, où le génome d’un organisme est
spécialement modifié pour lui faire acquérir de nouvelles compétences métaboliques, permet de
créer des organismes capables de synthétiser des métabolites d’intérêt pour des applications
industrielles ou pharmaceutiques, qu’ils ne pourraient pas synthétiser naturellement.
La rétrobiosynthèse est une technique de résolution de problèmes dans le design de ces
nouvelles voies métaboliques. Elle consiste à décomposer récursivement le composé chimique](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-106-2048.jpg)
![101
d’intérêt en précurseurs, en suivant des chemins de transformations jusqu’à des molécules
disponibles dans le commerce à moindre coût ou naturellement produites par l’organisme
modifié. Dans le cas de l’ingénierie métabolique, la rétrobiosynthèse consiste à appliquer des
transformations chimiques réverses (c’est à dire des réactions catalysées par des enzymes dans le
sens réverse) au produit souhaité, en suivant des chemins jusqu’aux substrats endogènes à
l’organisme modifié. Le but final est d’identifier les gènes des enzymes à insérer dans l’organisme
pour le rendre capable de synthétiser une molécule d’intérêt. Un exemple de voie de
rétrobiosynthèse est celle de la production du taxol dans la levure [29].
Souvent, la synthèse d’un composé chimique va avoir plus d’un chemin de synthèse possible. La
rétrobiosynthèse permet de sélectionner les meilleurs chemins, notamment grâce à l’étude du
rendement catalytique des enzymes et son optimisation.
Ainsi, les approches de rétrobiosynthèse permettent de trouver de nouvelles voies métaboliques.
Deux d’entre elles sont présentées dans ce manuscrit.
Le framework BNICE (Biochemical Network Integrated Computational Explorer) [233] permet de
générer de nouvelles réactions biochimiques à partir d’un ensemble de règles de réactions
enzymatiques et d’un ensemble de composés chimiques de départ. Cette technique permet, à
partir de nos connaissances sur les activités enzymatiques, de simuler toutes les façons dont les
composés chimiques peuvent être transformés, ce qui peut permettre la découverte et le design
de nouvelles voies métaboliques. L’algorithme M-path [234] fonctionne aussi sur ce principe. A
partir des connaissances sur les métabolites et les réactions enzymatiques disponibles dans les
bases de données publiques, il permet de générer des voies métaboliques et des réactions
enzymatiques potentielles.
RetroPath [235] est un pipeline automatisé qui permet l’exploration des possibles circuits
métaboliques à partir des signatures moléculaires des métabolites et des réactions (RMS) [236] et
de sélectionner les meilleures voies métaboliques possibles en fonction des contraintes
souhaitées. Les molécules potentielles pouvant être produites par les réactions données sont
énumérées et permettent l’assemblage de nouvelles voies métaboliques (synthétiques). Intégré
dans une approche globale comprenant aussi la recherche de gènes codant pour des enzymes
pouvant catalyser les réactions d’intérêt, et la prédiction du potentiel promiscuitaire de ces
enzymes grâce à l’apprentissage artificiel, il s’agit d’une méthode efficace de prédiction ab initio de
chemins métaboliques.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-107-2048.jpg)
![102
Il faut cependant se rappeler qu’une bonne modélisation du réseau métabolique est nécessaire
pour découvrir efficacement de nouvelles voies métaboliques. En effet, les métabolites
ubiquitaires et secondaires ainsi que le sens des réactions, peuvent poser problème et entrainer
des prédictions fausses.
IV.3.1.3 Alignement de voies métaboliques
A la fin du siècle dernier, des approches de comparaison et d’alignement de voies métaboliques
entre les organismes ont commencé à émerger [237]. Depuis, des méthodes de plus en plus
élaborées ont été publiées pour comparer et aligner efficacement, et surtout automatiquement, les
voies métaboliques.
Il est important d’être capable de détecter à la fois une topologie similaire entre des voies
métaboliques, mais aussi de prendre en compte les étiquettes sur les nœuds (les enzymes que ces
nœuds représentent). L’algorithme MetaPathwayHunter [238], notamment, permet d’aligner les
voies métaboliques sur ces deux critères simultanément.
L’alignement des voies métaboliques en se basant sur la structure des molécules chimiques
impliquées dans les réactions peut aussi s’avérer très efficace. Il s’agit de mesurer la similarité de
structure des métabolites. Ces structures peuvent être représentées par différents descripteurs
moléculaires qui sont comparés ensuite sous la forme de fingerprints en utilisant des métriques
comme le coefficient de Tanimoto ou de Jaccard. Cette méthode a, notamment, été appliquée par
Tohsato et al. [239] pour mettre en évidence des similarités entre les voies de biosynthèse du
glucose, du mannose et du galactose chez Escherichia coli. L’alignement des molécules entre voies
métaboliques permet aussi faire du mapping d’une molécule d’une voie métabolique donnée sur
plusieurs molécules d’une autre voie métabolique, ce qui serait biologiquement plus correct. Cette
approche, combinée à la comparaison de topologie de voies métaboliques intégrée dans SubMAP
[240] a été testée sur les données de KEGG et permet d’aligner très efficacement des voies
métaboliques entre elles, et est donc un bon outil de recherche de nouvelles voies métaboliques
par ce biais.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-108-2048.jpg)
![103
La comparaison des modifications subies par les molécules au cours des réactions peut aussi être
utilisée pour aligner les voies métaboliques entre elles [241]. Les voies métaboliques peuvent
d’ailleurs aussi être directement alignées sur les réactions (et non pas sur les molécules et/ou leur
modifications), à condition de pouvoir aligner une réaction sur plusieurs autres et ainsi prendre
en compte la variabilité enzymatique inter-espèces (CAMPways [242]).
La détection de similarités entre voies métaboliques par leur alignement permet aussi de détecter
des séquences répétées de réactions similaires dans le réseau métabolique (motifs) ainsi que des
ensembles de réactions relativement indépendants du reste de ce réseau (modules). Ces deux
notions, ainsi que les méthodes orientées spécialement vers leur détection, sont présentées dans
la section suivante.
IV.3.2 Motifs dans le métabolisme & modules de réactions
Des blocs fonctionnels réalisant la même chimie sont souvent retrouvés dans les réseaux
métaboliques. On peut donc supposer que l’évolution du métabolisme peut se faire par blocs
conservés de transformations chimiques qui se diversifient en termes de réactions spécifiques
[243]. C’est d’ailleurs autour de cette constatation que s’est construit le travail présenté dans cette
thèse. Ces blocs fonctionnels peuvent être perçus de deux façons différentes dans les
représentations mathématiques du métabolisme : comme des motifs et comme des modules. La
différence entre ces deux notions est illustrée dans la Figure 22. Concrètement, il faut retenir
qu’un motif est répété et qu’un module est autonome. Dans un réseau métabolique, un module
correspondrait à un sous-graphe qui aurait plus de connections entre ses éléments qu’avec les
autres éléments. Pour comprendre la notion de motif dans un réseau métabolique, il faut
imaginer que les nœuds partageant une même propriété (métabolites appartenant à une même
classe chimique ou réactions effectuant le même type de transformation sur les molécules, par
exemple) sont coloriés de la même façon. Le même enchainement d’un ensemble de couleurs
répété à différents endroits du réseau sera considéré comme un motif. Les motifs sont donc des
outils très pratiques pour détecter des cooccurrences fréquentes d’un ensemble de
transformations chimiques qui peuvent être considérés comme des modules conservés.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-109-2048.jpg)
![104
Dans les deux cas, la recherche de telles sous-structures topologiques peut s’apparenter à la
recherche d’ensembles de réactions et/ou de métabolites d’importance biologique, ce qui
ressemble beaucoup à la recherche de voies métaboliques. Il existe un certain nombre de
définitions et méthodes de recherche de modules et de motifs dans les réseaux métaboliques,
quelques unes sont présentées dans les sections suivantes.
Figure 22. Motif vs module.
IV.3.2.1 Motifs dans le métabolisme
Dans un réseau biologique, un « motif » est souvent défini comme un ensemble de connections
qui se retrouve de manière exceptionnelle dans un réseau (c’est à dire qui apparaît
significativement plus souvent qu’un ensemble aléatoire de connections). Dans ce cas, où seule la
topologie des connections entre les nœuds compte, on parle de « motifs topologiques » [244,
245].
Une définition améliorée d’un motif, particulièrement adaptée aux réseaux métaboliques, a été
proposée par la suite par Vincent Lacroix [25]. Dans le contexte d’un graphe de réactions, tous
les nœuds ne sont pas équivalents. On peut les distinguer par classes fonctionnelles (qu’on peut](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-110-2048.jpg)
![105
aussi appeler « couleurs » pour imager et généraliser le concept). La topologie exacte de
l’ensemble des nœuds n’a alors qu’une importance secondaire, tant que les nœuds sont connectés.
Ici, un motif, que l’on appellera « motif coloré », est un multi-ensemble de classes fonctionnelles
de réactions prises dans toutes les classes fonctionnelles de réactions possibles apparaissant dans
le réseau. Plus le motif est fréquent, plus il a d’occurrences dans le réseau, et plus il a donc une
signification biologique importante. La recherche de motifs, topologiques comme colorés, est un
problème difficile du point de vue computationnel (NP-complet) [246].
Cette figure présente un exemple de voies impliquées dans la biosynthèse d’acides aminés (Figure
23). Dans la biosynthèse de la valine, de la leucine et de l’isoleucine, on constate que l’on retrouve
des nœuds appartenant aux mêmes classes fonctionnelles de réactions (dans l’exemple présenté
dans la figure, les réactions sont classées ensemble si elles sont toutes les deux annotées avec les
mêmes trois premiers nombres d’EC numbers).
IV.3.2.2 Modules dans le métabolisme
Un module réactionnel est un ensemble conservé de transformations chimiques. Un motif de
réactions conservé dans un réseau métabolique est finalement un outil pour détecter des modules
de transformations conservés. Ces modules peuvent être considérés comme des briques de
construction d’un réseau métabolique et reflètent une logique chimique d’un enchainement de
Figure 22. Exemple d’un motif dans le métabolisme (image extraite de Lacroix et al. [25]). Dans la biosynthèse de la
leucine, valine et isoleucine, une partie des réactions impliquées sont annotées avec des EC numbers similaires (au
moins les trois premiers nombres des EC numbers identiques).](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-111-2048.jpg)
![106
réactions dans le métabolisme. Les limites des modules correspondent souvent aux voies
métaboliques ou à des sous parties. Deux méthodes de recherche de modules seront présentées
ici.
La détection des RModules [27] dans les voies métaboliques de KEGG est basée sur la
classification des réactions selon leur RClass (cf. section IV.1.2). Les RClass étant trop précises
pour décrire les réactions, Muto et al. ont comparé les RClass en utilisant des fingerprints pour
obtenir au final 376 groupes de réactions (et 1190 singletons) ayant des RClass similaires. Les
voies métaboliques de KEGG ont ensuite été alignées à partir d’un calcul de tous les chemins
possibles de réactions (de longueur de 2 à 8 réactions) convertis en groupes de RClass. Ils ont
obtenu entre 88 (longueur 8) et 928 (longueur 2) chemins conservés. Cependant, cette méthode
demande une curation manuelle car la classification des réactions selon les groupes de RClass ne
garantit pas la conservation de la transformation chimique entre des réactions d’un même groupe
du à l’utilisation des fingerprints.
Une curation manuelle a donc été réalisée par les auteurs pour arriver au final à une liste de 34
modules conservés (http://www.kegg.jp/kegg/reaction/rmodule.html).
L’identification de modules conservés de réactions peut aussi se baser sur l’homologie des
enzymes qui catalysent des réactions. Ainsi, un module réactionnel peut être défini comme au
moins deux réactions successives catalysées par des enzymes homologues dans des voies
métaboliques alignables par rapport à leur similarité de réactions. Cette définition a notamment
permis d’identifier des similarités réactionnelles et enzymatiques dans le catabolisme des purines,
ce qui a entrainé la découverte d’une nouvelle voie de dégradation [26].](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-112-2048.jpg)
![107
IV.4 Visualisation des réseaux
Une partie de l’analyse de réseaux et de voies métaboliques peut se faire en visualisant les
données. Il existe un certain nombre d’outils qui permettent de visualiser d’une façon efficace les
données sous forme de réseaux. Tout d’abord, les grandes ressources de données métaboliques,
KEGG [98] et BioCyc [91] proposent une visualisation des voies métaboliques. Cependant, pour
une analyse globale d’un réseau métabolique, le visualiser en entier est plus intéressant. Les deux
ressources proposent donc des cartes métaboliques globales, où l’utilisateur peut colorier les
nœuds, mais il y a un manque d’interactivité et de possibilité d’édition des réseaux affichés.
Plusieurs logiciels, permettant à l’utilisateur d’interagir, d’éditer et d’analyser directement les
réseaux, existent.
Cytoscape [247], le plus populaire dans la communauté bioinformatique, est codé en langage
Java et présente de nombreux avantages. La possibilité d’intégrer au logiciel diverses applications
développées par la communauté en fait un outil d’analyse, en plus d’être un outil de visualisation.
Il offre aussi la possibilité d’interactions directes avec les grandes bases de données publiques
biologiques en croisant les données très facilement. Son plus gros défaut vient de sa
consommation de ressources mémoires de l’ordinateur sur lequel il est exécuté, ce qui peut
ralentir fortement les interactions humaines avec le logiciel. Tulip [248] est un autre logiciel de
visualisation particulièrement bien adapté à de très grandes quantités de données. Ecrit en langage
C++, il offre un certain nombre de possibilités pour l’exploration rapide de réseaux biologiques,
notamment le croisement efficace avec les bases de données biologiques publiques. Gephi [249]
le dernier présenté ici, est un logiciel de visualisation et d’analyse de graphes qui utilise un moteur
de rendu tridimensionnel qui permet l’affichage des réseaux en temps réel et d’en accélérer
l’exploration.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-113-2048.jpg)
![108
Limites : Réactions métaboliques non-
enzymatiques
Il est convenu que les réactions transformant les petites molécules dans le métabolisme sont
spontanées ou catalysées par des protéines enzymatiques. Cependant, il existe des enzymes non-
protéiques, qui catalysent avec succès des réactions métaboliques. Leur présence peut expliquer
notamment l’existence d’activités enzymatiques orphelines. Elles sont aussi un grand challenge
pour la reconstruction métabolique à l’échelle génomique, car elles sont difficiles à prédire avec
les moyens actuels. Parmi les catalystes non-protéiques, on retrouve principalement les
ribozymes (aussi appelées RNA catalytique ou RNAzymes et qui sont des complexes
moléculaires constitués d’ARN pur ou d’une association entre des molécules d’ARN et des
peptides), des glycolipozymes [250, 251] qui sont des molécules composées d’un sucre et d’un
lipide et ayant une activité assimilée à une activité enzymatique et les DNAzymes [252]
(molécules d’ADN capables de repliement et de catalyse). Les ribozymes sont assez largement
étudiées, car sont considérées comme les vestiges du « monde à ARN » par les défenseurs de
cette théorie de l’évolution. De nombreuses publications [253–255] peuvent être consultées pour
en apprendre plus sur cette partie passionnante du métabolisme. Par ailleurs, le prix Nobel de
Chimie 1989 a été décerné à Thomas R. Cech et Sidney Altman pour la découverte des propriétés
catalytiques de l’ARN. Les glycolipozymes, par contre, sont encore très méconnues et n’ont été
découvertes qu’au début des années 2010 [250]. Elles auraient une activité liée au transport
transmembranaire, mais beaucoup de travail reste encore à faire pour comprendre comment elles
fonctionnent réellement, si elles sont fréquentes dans la nature et pour éventuellement établir une
stratégie pour en découvrir de nouvelles. Quand aux DNAzymes, ce sont des constructions
artificielles à partir d’ADN, sélectionnées pour leurs capacités d’auto-repliement, de fixation et de
catalyse de ligands. La recherche dans ce domaine est relativement récente (on parle pour la
première fois d’oligomères d’ADN ayant une fonction catalytique dans les années 1990 [256]) et
reste relativement discrète.
Pour conclure cette partie sur les réactions métaboliques non-enzymatiques, je voudrais évoquer
l’une des branches de la biologie de synthèse en plein développement, le XNA et les XNAzymes
[257]. Les XNA, pour « xeno-nucleic acids » sont des polymères génétiques synthétiques
composés de briques non-naturelles comme des sucres et des nucléobases alternatifs ou
connectés entre eux par une structure chimique différente. Les aptamères (oligonucléotides](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-114-2048.jpg)
![109
synthétiques capables de fixer un ligand) de XNA sont capables de se replier, de fixer des ligands,
sont plus résistants que l’ADN et l’ARN et sont aussi capables de catalyser des réactions
métaboliques [258]. De plus, un certain nombre de systèmes génétiques synthétiques constitués
de XNA supportent les notions d’hérédité et peuvent évoluer [259]. Toutes ces caractéristiques
font des XNAzymes des outils alternatifs très intéressants pour la biologie de synthèse.
L’avenir de l’étude du métabolisme réside donc non seulement en la compréhension de plus en
plus précise de son fonctionnement, mais aussi à la création de nouvelles briques de celui-ci.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-115-2048.jpg)

![111
Chapitre I
Actualisation des connaissances sur
les activités enzymatiques orphelines
de séquences
Les activités enzymatiques orphelines de séquences (surnommées aussi « enzymes orphelines »)
sont des activités enzymatiques connues et validées expérimentalement dans au moins un
organisme, mais pour lesquelles aucune protéine n’est connue pour les catalyser. Environ 20%
des activités enzymatiques annotées par un EC number sont orphelines de séquences. Ces
lacunes dans la connaissance sur les enzymes sont problématiques pour plusieurs raisons. En
effet, lors de la reconstruction des réseaux métaboliques à partir de génomes entiers, l’absence
d’association séquence-réaction laisse des trous dans les modèles métaboliques et engendre donc
des prédictions erronées. Aussi, l’absence de gène associé à ces activités orphelines ne permet pas
de produire l’enzyme en laboratoire par des techniques de biologie moléculaires et complique
ainsi une caractérisation biochimique fine. De même, cette lacune ne facilite pas l’utilisation ou la
modification de l’activité enzymatique dans des applications en ingénierie métabolique ou en
biologie de synthèse.
Dans ce premier chapitre, est présenté une revue complète des enzymes orphelines, publiée en
juin 2014 dans le journal Biology Direct. Un cas particulier d’activités enzymatiques orphelines, les
enzymes orphelines « locales » (par opposition aux classiques, qui elles sont « globales »), est
réintroduit et développé. Ces activités ont des séquences connues qui leur sont associées dans un
groupe taxonomique donné, mais pas dans un autre alors que l’activité a été également
caractérisée. Pour déterminer si un candidat homologue aux enzymes connues pourrait être
présent dans ces organismes orphelins, une stratégie simple, basée sur la méthode PRIAM [143],
a été appliquée. Cette méthode utilise des profils spécifiques à une activité enzymatique (plus](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-117-2048.jpg)
![112
sensibles et spécifiques qu’une simple comparaison de séquence par BLAST [13] pour détecter
par similarité de séquences des protéines candidates. Finalement, une étude de la relation entre les
familles de protéines et les activités enzymatiques auxquelles elles sont associées a été réalisée.
Une réflexion sur la promiscuité enzymatique et la multifonctionnalité des protéines conclut cette
revue sur les enzymes orphelines.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-118-2048.jpg)
![REVIEW Open Access
Profiling the orphan enzymes
Maria Sorokina1,2,3*
, Mark Stam1,2,3
, Claudine Médigue1,2,3
, Olivier Lespinet4,5,6
and David Vallenet1,2,3*
Abstract
The emergence of Next Generation Sequencing generates an incredible amount of sequence and great potential
for new enzyme discovery. Despite this huge amount of data and the profusion of bioinformatic methods for
function prediction, a large part of known enzyme activities is still lacking an associated protein sequence. These
particular activities are called “orphan enzymes”. The present review proposes an update of previous surveys on
orphan enzymes by mining the current content of public databases. While the percentage of orphan enzyme
activities has decreased from 38% to 22% in ten years, there are still more than 1,000 orphans among the 5,000
entries of the Enzyme Commission (EC) classification. Taking into account all the reactions present in metabolic
databases, this proportion dramatically increases to reach nearly 50% of orphans and many of them are not
associated to a known pathway. We extended our survey to “local orphan enzymes” that are activities which have
no representative sequence in a given clade, but have at least one in organisms belonging to other clades. We
observe an important bias in Archaea and find that in general more than 30% of the EC activities have incomplete
sequence information in at least one superkingdom. To estimate if candidate proteins for local orphans could be
retrieved by homology search, we applied a simple strategy based on the PRIAM software and noticed that
candidates may be proposed for an important fraction of local orphan enzymes. Finally, by studying relation
between protein domains and catalyzed activities, it appears that newly discovered enzymes are mostly associated
with already known enzyme domains. Thus, the exploration of the promiscuity and the multifunctional aspect of
known enzyme families may solve part of the orphan enzyme issue. We conclude this review with a presentation of
recent initiatives in finding proteins for orphan enzymes and in extending the enzyme world by the discovery of
new activities.
Reviewers: This article was reviewed by Michael Galperin, Daniel Haft and Daniel Kahn.
Keywords: Orphan enzyme activities, Enzyme discovery, Metabolic pathways, Enzyme promiscuity, Data survey,
Biological databases, Local orphan enzymes
Review
New progress in sequencing technologies generates
thousands of new sequences each day. With the large
public sequence databases combined with efficient bio-
informatic methods, it is possible to predict the function
of some new proteins mainly by comparative genomics
approaches. Nevertheless, millions of protein entries are
not assigned reliable functions due to the lack of trust-
worthy annotations and the drawbacks of homology-based
predictions [1]. This shortcoming illustrates our limited
knowledge of the functional diversity in the protein world
and restricts the analyses of an organism starting from its
genome. This is particularly the case for enzymatic activ-
ities that can be predicted by gene functional assignments
and used as a starting point to reconstruct genome-scale
metabolic models.
The first enzyme was discovered and isolated in 1833 by
Anselme Payen [2]. It was the first time a non-living com-
pound was shown to have properties of an organic catalyst,
a discovery which shook the scientific community. This
enzyme was named “diastase” (now called α-amylase) and
the suffix –‘ase’ will be henceforth used to refer to enzymes.
Since then, the number of discovered enzymes has continu-
ally increased, thanks to the experimental work of chemists
and biologists. In the beginning of enzymology, the naming
of enzyme was not systematic. Many different enzymes
* Correspondence: msorokina@genoscope.cns.fr; vallenet@genoscope.cns.fr
1
Direction des Sciences du Vivant, Commissariat à l’Energie Atomique (CEA),
Institut de Génomique, Genoscope, Laboratoire d’Analyses Bioinformatiques
pour la Génomique et le Métabolisme, 2 rue Gaston Crémieux, 91057 Evry,
France
2
CNRS-UMR8030, 2 rue Gaston Crémieux, 91057 Evry, France
Full list of author information is available at the end of the article
© 2014 Sorokina et al.; licensee BioMed Central Ltd. This is an Open Access article distributed under the terms of the Creative
Commons Attribution License (http://creativecommons.org/licenses/by/4.0), which permits unrestricted use, distribution, and
reproduction in any medium, provided the original work is properly credited. The Creative Commons Public Domain
Dedication waiver (http://creativecommons.org/publicdomain/zero/1.0/) applies to the data made available in this article,
unless otherwise stated.
Sorokina et al. Biology Direct 2014, 9:10
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-119-2048.jpg)
![were given similar names and, on the other hand, the same
enzymes had several names. An Enzyme Commission,
whose first meeting took place in 1961, was created to give
rules and recommendations that could be implemented for
the systematic naming of enzymes [3]. Enzyme activities
are nowadays classified with EC (Enzyme Commission)
numbers, a nomenclature maintained by the IUBMB
(International Union of Biochemistry and Molecular
Biology) [4-6]. To be integrated into the EC classification,
an activity must be observed and biochemically character-
ized without the necessity to identify the associated protein
that catalyzes the reaction.
Since 2003, several teams around the world have no-
ticed that many EC numbers have no identified coding
sequences for the enzymes catalyzing the corresponding
activities (Figure 1). In order to fill the missing knowledge
between genes and their function, Richard J. Roberts
called, in 2004, for a community action for the annotation
of genes of unknown function in microbial genomes [7].
The same year, Peter Karp proposed an enzyme genomic
initiative to associate at least one protein sequence for
every biochemically characterized enzymatic activity [8].
He noticed that many EC numbers (38% among 3,736
entries) were lacking an associated nucleic or protein
sequence in public databases, a problem that hadn’t been
really considered before by the scientific community. He
observed that his estimation could be biased as the EC
classification does not cover all known enzymatic activities.
Figure 1 Orphan enzyme chronicles. Studies on orphan enzymatic activities in the past ten years.
Sorokina et al. Biology Direct 2014, 9:10 Page 2 of 16
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-120-2048.jpg)
![Indeed, in sequence databases, some entries are missing an
EC number even if a correct textual description of the
enzymatic activity is annotated. He proposed to take
advantage of the numerous accessible sequenced genomes
and to cross this genetic information with published exper-
iments that have characterized the enzymatic activities.
This first data mining step should identify some candidate
proteins which could be experimentally validated.
In 2005, sequence-lacking enzymatic activities were
named “orphan enzymes” by Bernard Labedan and Oliv-
ier Lespinet in an open letter [9]. They conducted a
similar analysis to that of Peter Karp and showed that
42% of the EC numbers were orphan enzymes (1,625 EC
numbers among 3,820). One of the main surprises of
this study was the fact that 200 organisms had orphan
enzymes, despite the availability of their complete gen-
ome. They also noticed that, in several cases, the protein
catalyzing the enzymatic activity had been identified but
not sequenced. The following year they published two
exploratory articles on orphan enzymes [10,11]. The
proportion of orphan enzymes was updated, giving a
slight decrease of 3% (39% of orphans, 1,525 EC entries
among 3,877). They pointed out that a number of path-
ways (~100) had at least one orphan enzyme. They also
made several remarks on the use of EC numbers. More-
over, they created a public database, called ORENZA,
listing all orphan enzymes present in the EC nomencla-
ture and allowing users to perform queries by tracking
them between organisms and pathways [10].
In 2007, Lifeng Chen and Dennis Vitkup carried out a
very detailed review on the historical accumulation of
orphan enzyme activities and a wide range of statistical
analyses on their distribution across different classifications
[12]. They found 1,360 orphans, representing 34% of the
4,003 valid EC entries. They investigated the number of
biochemical characterizations per year of discovery and
noticed that it decreased in the 1970s and 1990s. A study
of the relation between orphan enzymes and their pathway
neighbors was conducted: 39% of network neighbors for
orphan activities were orphan themselves, compared with
29% for neighbors of non-orphan activities. They also
noticed that a majority of orphan activities were found in
the most studied organisms. Finally, they pinpointed a
possible bias in the EC classification because many reac-
tions in metabolic databases were not associated with any
EC number. Considering this limitation, they estimated that
up to 50% of all know biochemical reactions were orphan.
Here, we present an extended review on orphan
enzyme activities by updating previously conducted
surveys and performing new analyses. We first update
the estimation of the number of orphan enzymes and
interpret their decrease in the light of past and recent
enzyme activity discoveries. As the EC classification
does not totally cover all known activities, we briefly
introduce two main metabolic databases and analyze
their content to estimate orphans at the reaction level.
Also, an analysis of their connectivity in metabolic net-
work is made. The concept of orphan enzymes is then
extended to local orphans (i.e. activities which have no
representative sequence in a given clade, but have one
in other organisms) and an analysis is made at the
superkingdom level to estimate their number and to
evaluate if candidate proteins for local orphans could
be retrieved by sequence homology. Finally, we expose
the notion of promiscuity and multifunctionality in the
enzyme world and explore the relation between protein
domains and catalyzed activities. In conclusion, we
present some new initiatives and concepts of interest to
reduce the number of orphan enzymes but, also, to extend
the landscape of enzymes by finding new activities.
An updated view of orphan enzymes
In this study, we estimated the number of orphan enzymes
by using EC numbers present in the IntEnz [13] and
UniProt [14] databases (versions of February 2013).
UniProt is a resource of proteins where enzymatic activ-
ities are described using the EC classification. Only valid
and complete EC entries were considered without taking
into account deleted or transferred entries. We also
considered as valid entries the nearly 100 provisional
EC numbers of IntEnz waiting to be confirmed by the
IUBMB. It appears that 22.4% of the enzymatic activities
are orphans; among the 5,096 EC numbers, 1,143 entries
have no associated protein in UniProt. As noticed previ-
ously [12], the proportion of orphan enzymes is not uni-
formly distributed across the different classes of the EC
nomenclature: in EC class 1 the fraction is 25%, 26% in
class 2, 19% in class 3 and 4, 15% in class 5 and 13% in
class 6 (Additional file 1: Figure S1.1 and Additional file 2:
Table S2.1 for the complete list of orphan EC numbers).
In comparison with the first study made by Peter Karp
in 2003 [8], we observe a significant decrease in the
number of orphan activities (−294 EC entries) while
the number of EC entries has increased considerably
(+1,360 entries) in the last ten years. To interpret this
result, we performed a survey of the EC classification
dynamics in terms of entry creations and updates (Figure 2).
Since 2010, more than 800 EC numbers have been created
and a substantial number of old entries have been
re-classified (i.e. deleted or transferred to another entry).
Over the last few years, the EC commission has consider-
ably enhanced its activity and increased the coverage of
the EC classification in terms of number of new enzymatic
activities. Before the year 2000, the EC classification was
not updated regularly each year, whereas new EC numbers
are now created several times a year, suggesting that the
Enzyme Commission tries to minimize the time between
the publication of a new activity and its EC attribution.
Sorokina et al. Biology Direct 2014, 9:10 Page 3 of 16
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-121-2048.jpg)
![Nevertheless, many of these new EC entries correspond to
older biochemical characterizations as depicted in Figure 3,
where the delay between activity discoveries and corre-
sponding EC creations is shown. This pitfall limits the
search of enzymes in public databases since EC numbers
are the only standardized way for scientists to publish an
enzymatic activity associated with a protein sequence.
Moreover, many recently characterized activities have
no associated protein entries, see Figure 4. We can sup-
pose that the annotations of the corresponding proteins
were not updated accordingly with the correct complete
EC numbers. This delay of knowledge in databases,
which was reported by Yannick Pouliot and Peter Karp
in 2007 [15], remains the case today and it impacts the
evaluation of orphan enzymes because numbers of
recently discovered enzymes are wrongly considered as
orphans. These authors defined a strategy in order to
determine which orphans might be salvageable and
extrapolated that around 18% of them can be solved
with a literature search. At the time of writing, this type
of analysis was applied to a wide list of orphan EC
numbers [16]. The authors found protein sequences
for about 270 activities among 1,122 putative orphan
enzymes that were extracted from databanks in 2009.
Using their results and the current knowledge in data-
banks, protein entries for 112 false orphans could be
updated with the corresponding activities and literature
evidences.
To get a better view of the dynamics of the enzyme
discovery in the past century, we computed the number
of characterized activities over the years, represented by
the solid red curve in Figure 5. As previously reported
by Chen et al. [12] several phases can be observed.
The 1930s and 1940s correspond to the beginning of
biochemistry with a few numbers of characterized
enzymatic activities. The 1950s and 1960s then saw an
explosion of newly discovered activities due to tech-
nical progress in biochemistry and scientists’ increas-
ing interest in this new field. This golden age of
biochemistry took place in parallel with the progress in
DNA knowledge and the emergence of molecular biol-
ogy. These two complementary disciplines synergized
in the 1980s and 1990s as shown by a second peak of
enzymatic activities in Figure 5. Simultaneously, the
number of activities associated for the first time with a
protein sequence increased considerably (dashed green
curve in Figure 5). Before this period, the purification
and the direct sequencing of proteins were laborious
and very few enzyme sequences were determined as it
required highly purified polypeptides and was limited
to short polypeptides. The improvements in molecular
biology techniques, like DNA sequencing and expression
cloning, permitted quick association between nucleic
sequences (i.e. genes) and enzymes, whether the latter
was long-known or recently discovered. The emergence of
whole-genome sequencing projects and then, the Next
Figure 2 EC classification evolution over years. (a) Snapshot of EC number status by year of creation. This barplot represents the number of
created EC numbers over years and the proportion of nowadays active entries in red and transferred/deleted entries in pink. (b) Dynamics of the
EC entry creations and status changes over years. This barplot represents the number of EC entry modifications over years: creation (yellow), transfer
(light red) and deletion (dark red).
Sorokina et al. Biology Direct 2014, 9:10 Page 4 of 16
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-122-2048.jpg)
![Generation Sequencing (NGS) technologies should have
eased the discovery of associations between genes and
enzymatic activities. Unfortunately, since the year 2000
the number of newly discovered activities is not main-
tained at the established level and starts to dramatically
decrease (Figure 5). It may be due to difficulties in
publishing such biochemical characterizations, and also
to the fact that funding is now directed towards other
priorities. The gap between the number of sequences
present in public databases and the number of cha-
racterized enzymes continues to increase dramatically
[17-19]. In 2010, Hanson et al. pointed out the dual
problem of increasing number of proteins of unknown
function produced by genome projects, facing the orphan
enzymes missing sequence information [20]. They
suggested using experimental data and comparative
genomics in order to predict candidate genes.
Orphan enzymes in the metabolic world
It is important to distinguish the terms “enzyme” and
“enzymatic activity”. The first designates a protein able
to catalyze a chemical reaction and the second one the
chemical reaction catalyzed by the enzyme. Therefore, an
EC number does not represent the enzyme itself, but only
the activity. As a consequence, non-homologous isoen-
zymes (i.e. with different ancestral origin) may share the
same EC number as they catalyze the same enzymatic
reaction. In the case of substrate promiscuity, different
EC numbers may exist to give precision to the nature of
transformed compounds. Otherwise, only one EC number
may be available and represents a generic transformation
that could occur on different substrates (e.g. alcohol
dehydrogenase, hexokinase). The promiscuity aspect of
enzymes is extensively described below. Besides, a same
chemical transformation may be represented by different
Figure 3 Delayed knowledge in the EC classification. Heatmap of the number of EC entries reported by the year of the activity discovery
(X axis) versus the year of the corresponding EC entry creation (Y axis). The square’s shade of gray is proportional to the number of EC entries.
A delay can be observed between the discovery of an activity and the creation of the corresponding EC number.
Sorokina et al. Biology Direct 2014, 9:10 Page 5 of 16
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-123-2048.jpg)
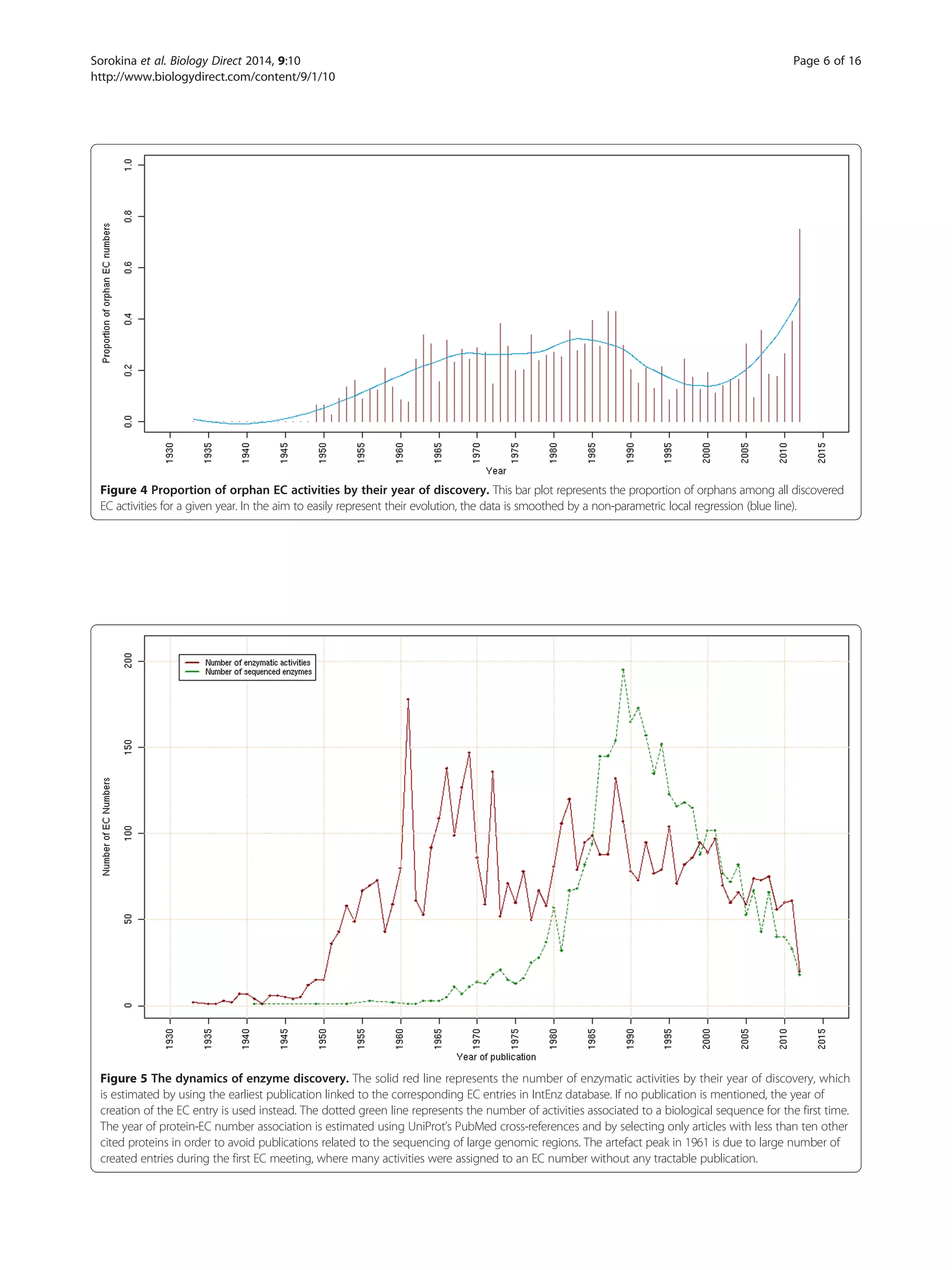
![EC numbers when, for example, different cofactors are
used. This multiplicity between related activities and EC
numbers may lead to discrepancies in databases and mask
some orphan enzymes. Another point, reported by Green
et al. [21], is the ambiguity in the use of incomplete EC
numbers that could lead to enzyme annotation errors in
public databases. This is because incomplete EC numbers
don’t distinguish between the lack of knowledge of the
exact substrate specificity of an enzyme and the lack of an
official EC number to describe the given activity. Conse-
quently, the use of EC numbers may have introduced
some biases in our survey. It should be noticed that the
UniProt consortium is making improvements in the repre-
sentation of the enzymatic activities through Rhea [22]
and UniPathway [23] databases, which are focused on the
definition of chemical reactions and metabolic pathways,
respectively.
To complete our survey at the chemical reaction level,
we performed a study on orphan enzymes using two
metabolic databases, named KEGG (version 65.0) [24]
and MetaCyc (version 17.0) [25]. The comparison of
these two databases was extensively reviewed in a recent
publication [26]. As a difference with EC nomenclature,
KEGG and MetaCyc make a clear distinction between
the chemical reactions and the enzymatic activities.
MetaCyc has adopted a formal representation of the
relation between proteins and chemical reactions they
can catalyze and thus deals with the multiplicity of
enzymatic activity-reaction relations. For example, if an
enzyme is able to catalyze the same chemical transform-
ation on a wide range of substrates (i.e. the substrate
promiscuity of the enzyme), the different chemical reac-
tions will be explicitly linked to the enzymatic activity
description. In other cases, an EC entry may give only a
general description of the overall reaction whereas the
different steps of this chemical transformation may be
more precisely described using several reaction steps.
The results of our analysis are summarized in Table 1.
About twice as many reactions are found in the two
pathway databases in comparison to the ~5,000 EC
entries. This high number of reactions is partly due to
the multiple relations between enzymatic activities and
reactions described above: in KEGG and MetaCyc, there
is an average of 1.15 and 2.2 reactions per EC number,
respectively. Conversely, a large proportion of these
reactions correspond to enzymatic activities not de-
scribed by a complete EC entry, reflecting the previously
mentioned delay between an activity discovery and its
official classification by the commission. In KEGG and
MetaCyc, there are 4,588 and 4,497 reactions not
linked to a complete EC number, respectively. As a
consequence and as noted previously [12,27], the per-
centage of orphan enzymes may be underestimated
using only the EC classification. It increases consider-
ably when the estimation is made at the reaction level
using metabolic resources: in KEGG and MetaCyc,
48% and 39% of the reactions are lacking associated
protein or nucleic sequences, respectively.
Enzymes are classically studied through metabolic
pathways, which are groups of activities taking part in a
same biological process. In this survey, we studied the
orphan enzyme content and their connectivity at the
pathway level. As described previously [26], there are
several key differences between the way the databases
represent the notion of a pathway: KEGG pathways are a
kind of mosaic of similar pathways predicted in different
species; in MetaCyc, the overall reactions in a pathway
are supposed to occur in a defined group of species.
Therefore, there are 12 times more pathways in Meta-
Cyc than in KEGG, as MetaCyc attempts to provide
distinct pathway variants for a given metabolic process
(Table 1). An important fraction of pathways (87% in
KEGG and 36% in MetaCyc) contains at least one
orphan activity. There is no pathway in KEGG containing
only orphan enzyme activities, whereas it is the case
for about a quarter of the MetaCyc pathways. This is
explained by the very large number of reactions in
KEGG pathways in comparison to MetaCyc (80 on aver-
age per pathway versus 4). Considering pathways contain-
ing a mix of orphan and non-orphan activities in KEGG
and MetaCyc, an average of 26.0% and 39.5% of the
reactions per pathway corresponds to orphan enzymes,
respectively (Table 1). These statistics show that an im-
portant proportion of pathways are still not completely
resolved at the gene level, which limits in silico recon-
structions of genome-scale metabolic models [28,29].
To cope with this problem, computational tools were
developed to find candidate genes for these missing
enzymes by using genome and metabolic context-based
methods [30-32]. The concept of these methods and the
illustration of integrated tools using genomic and post-
genomic data to link gene and function have been
Table 1 Statistics on orphan reactions in KEGG and
MetaCyc metabolic databases
MetaCyc KEGG
Total number of non-spontaneous reactions 10126 9148
Number of orphan reactions 3929 4348
Number of reactions in a pathway 6873 6271
Number of orphan reactions in a pathway 1833 1716
Number of orphan reactions having a non
orphan pathway neighbour
915 1223
Number of pathways 2002 150
Average number of reactions per pathway 4 80
Number of pathways with only non orphan
reactions
1264 19
Number of pathways with only orphan reactions 155 0
Sorokina et al. Biology Direct 2014, 9:10 Page 7 of 16
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-125-2048.jpg)
![reviewed recently [33]. Another illustration is presented
through the MicroScope platform as a combination of
CanOE and phylogenetic profile methods [32,34]. Actu-
ally, these in silico predictions have not raised a lot of
orphan cases despite the sophistication of the methods
and their relative independence from classical sequence
based methods. As many orphan enzymes (1,223 reactions
in KEGG and 915 in MetaCyc) have pathway neighbors
that are orphans themselves, one difficulty is the definition
of correct genomic contexts including candidate genes
and known enzymes. Furthermore, there is some part of
the metabolism with a lot of missing knowledge like gly-
can and lipid pathways. For example, a number of orphan
enzymes still exist in ether lipid metabolism, even if some
recent progresses were made [35].
Local orphan enzymes
From a taxonomic point of view, we propose to make
the distinction between global and local orphan en-
zymes. Orphan enzymes were previously defined as ac-
tivities having no associated gene in any organism,
which we called here global orphans. In addition, a
local orphan is an experimentally observed activity in at
least one organism of a given clade with only associated
sequences in organisms from other clades [36,37]. To
illustrate this concept at the superkingdom level, we
present here the example of the EC number 4.1.1.12,
the aspartate 4-decarboxylase, which catalyzes the
transformation of an L-aspartate in an L-alanine by
releasing a molecule of CO2. In UniProt, 327 bacterial
proteins are annotated with this EC number, including
two SwissProt entries, but no eukaryotic or archaeal
sequences can be found. Nevertheless, the aspartate
4-decarboxylase activity has been characterized in vari-
ous mammalians (e.g. rat, pig, chicken) [38], making the
EC number 4.1.1.12 a local orphan activity in eukary-
otes. For the Archaea, there is no associated sequence
and no literature evidence of its presence in this super-
kingdom. Thus, the aspartate 4-decarboxylase activity
could be considered as absent in the Archaea.
To conduct a survey of local orphans, a resource
of characterized activities in identified organisms is
required and should be exhaustive enough to gather
all the biochemical knowledge published in the past
century. We used the BRaunschweig ENzyme DAta-
base (BRENDA, version 2013), which is one of the
major public resources on enzymes and enzymatic
activities, and contains a very large spectrum of infor-
mation related to them [39]. BRENDA is based on the EC
number classification and gathers valuable information
about biochemical experiments that were extracted from
the literature. In complement to BRENDA that contains
only manually annotated data, the FRENDA (Full Refer-
ence ENzyme DAta) and AMENDA (Automatic Mining
of ENzyme DAta) subsections are based on an automatic
text-mining of article abstracts and provide an exhaust-
ive collection of organism-specific enzyme information.
BRENDA was used in our survey to extract, for each
enzymatic activity, a set of organisms for which the
activity was observed. In combination with UniProt
data, the proportion of global and local orphan enzymes
at the superkingdom level was then estimated (Figure 6;
the lists of local orphan and not observed EC numbers
are available in Additional file 2: Tables S2.2 and S2.3
for Bacteria, S2.4 and S2.5 for Eukaryota, and, S2.6 and
S2.7 for Archaea). Interestingly, we found that the pro-
portion of orphan enzymes is higher in Eukaryota than
in Bacteria (26% and 18%, respectively). Among the one
thousand orphan activities in eukaryotes, a third corre-
sponds to local orphans (31%) whereas the fraction is
lower in Bacteria (21%). These slight differences could
reflect a higher difficulty in experimental procedures to
identify genes or proteins in eukaryotes. In Archaea, the
low number of enzymatic activities (1,322 EC numbers),
which are reported in BRENDA and UniProt, clearly
illustrates our limited knowledge of metabolism of this
superkingdom. In our study, the proportion of archaeal
orphan enzymes is thus clearly underestimated. Indeed,
new specific enzymatic activities need to be discovered as
their chemistry shows many differences from other forms
of life. Nevertheless, a high proportion of reported
orphans in Archaea (77%) are local orphans, suggesting
either homolog proteins could be candidates for these
activities or specific isoenzymes have emerged during
their evolution. A similar analysis was conducted by
adding FRENDA/AMENDA data (Additional file 1:
Figure S1.2). Surprisingly, the number of orphan en-
zymes considerably increased in each superkingdom
with a high proportion of local orphans (52% for
Eukaryota and Bacteria, and 91% for Archaea). These
results should be taken with caution as FRENDA/
AMENDA data is not subjected to manual curation
(e.g. we found false-positive local orphans for Bacteria
that correspond to heterologous expressions of eukaryotic
proteins in Escherichia coli BL21). Nevertheless, this
analysis demonstrates that, in addition to the 22.4% of
global orphan, the proportion of EC numbers which are
local orphans in at least one superkingdom is consider-
able and is estimated between 9.5% (BRENDA alone)
and 33.5% (including FRENDA/AMENDA). Despite the
observed decrease of orphans at a global level, this high
number of enzyme activities (>30%), for which no or
incomplete sequence information is available, remains
problematic in our knowledge of metabolism.
Two reasons may explain this high proportion of local
orphan enzymes. Firstly, non-homologous isofunctional
enzymes, referred as NISE [40], may remain to be
discovered. They correspond to proteins that evolved
Sorokina et al. Biology Direct 2014, 9:10 Page 8 of 16
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-126-2048.jpg)
![independently, but catalyze the same biochemical reac-
tions. Therefore, these analogous enzymes cannot be
detected by classical comparative genomics approaches,
as they do not share any detectable sequence similarity.
Secondly, candidate homologous proteins may exist for
local orphans but remain to be experimentally confirmed
and annotated in databanks. To address this second point,
we conducted a preliminary analysis to find homologous
proteins for all local orphan enzymes in a given superking-
dom. For that purpose, we applied the PRIAM software
(release of March 2013) [41] against all UniProt proteins
from the Eukaryota, Bacteria and Archaea superkingdoms
(see Additional file 1: Figure S1.3). PRIAM relies on a set
of profiles (i.e. position-specific scoring matrices), which
are supposed to be characteristic of protein modules
sharing same enzyme activities (i.e. same EC numbers).
We found that PRIAM is able to retrieve candidate
proteins for a non-negligible fraction of local orphans
previously defined using BRENDA data: 30% for Archaea
and Bacteria, and 59% in Eukaryota (Table 2; the lists of
candidate proteins for local orphan and not observed EC
numbers are available in Additional file 3: Tables S3.1 and
S3.2 for Bacteria, S3.3 and S3.4 for Eukaryota, and, S3.5
and S3.6 for Archaea). Even if these predictions cannot be
transferred directly without supplementary bioinformatics
analyses or experiments, they give strong clues on protein
candidates for local orphan enzymes. Another interesting
feature is the substantial number of putative candidates
for activities that have never been seen in a given super-
kingdom (“not observed” columns in Table 2). Only 21%
of not observed EC numbers in Archaea have candidate
proteins whereas the total number of known enzymatic
activities is low in this superkingdom (n = 1,322, Figure 6).
This result is in agreement with the specificity of their
metabolism, which may be a reservoir of new enzyme
families and pathways. Conversely, the percentages of
potentially resolvable local orphans and not observed
enzymes in eukaryotes are higher than the two other
superkingdoms, at 59% and 46% respectively. This sug-
gests that the set of common enzymes between Bacteria
and Eukaryota may be underappreciated in protein
databanks and could be partially solved by a curation
Figure 6 Orphan and non-orphan EC number distribution across superkingdoms. The green pie chart represents the proportion of orphan
EC activities among all valid entries. Other pie charts represent the proportion of orphan activities among each superkingdom. An activity is
considered as present in a superkingdom if at least one protein is annotated with corresponding EC number or the activity has been observed in
an organism according to BRENDA database. The number and percentage of local and global orphans are given for each superkingdom. The
small amount of characterized EC numbers in Archaea shows the obvious lack of knowledge about their metabolism.
Sorokina et al. Biology Direct 2014, 9:10 Page 9 of 16
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-127-2048.jpg)
![effort of eukaryotic genome annotations. As already illus-
trated, comparative genomics analyses between prokary-
otes and eukaryotes are successful in finding common
and specific enzymes in shared pathways [20]. These
homology-based predictions of enzymatic functions
could be also completed by probabilistic annotation of
metabolic networks to increase the accuracy of this
strategy [42].
Enzyme promiscuity and protein families
Multifunctional enzymes are enzymes capable of playing
several roles in metabolism by catalyzing different trans-
formations that may occur in different pathways. Several
kinds of multifunctionality can be observed. Some
enzymes may show broad substrate specificity. This
substrate promiscuity is a feature of enzymes able to
catalyze the same chemical reaction on a variety of
related compounds [43]. Other enzymes may catalyze
different chemical transformations. One can observe
proteins having two or more functional domains with
different active sites [44]. The association of several
domains within a protein, which is generally the result
of a gene fusion event during evolution, may facilitate
substrate conversion and regulation of the metabolic
fluxes. Another origin of this catalytic promiscuity is
the special case of moonlighting enzymes [45]. These
proteins switch between activities under environmen-
tal changes according to their cellular localization,
expression in a novel cell type, ligand or cofactor con-
centrations, oligomerization or complex formation with
other proteins. A repository of multitasking proteins
was recently set up and several examples of moonlight-
ing enzymes may be explored [46].
The proportion of multifunctional enzymes may be
underestimated [47,48] and only a few enzymes are
described as multifunctional in databases: among
the ~250,000 enzymes in Swiss-Prot, 5% are associated
with two or more EC numbers and 3% with EC num-
bers having different classification at third-level. This
proportion should dramatically increase when we will find
a simpler way to detect them. Recently, a bioinformatic
method based on reaction molecular signatures was pro-
posed to predict catalytic and substrate promiscuity [49].
Using this method, a complementary study showed
that highly promiscuous enzymes are more likely to be
widespread in the tree of life [50]. Because multifunctional
enzymes are so difficult to discover and annotate,
they represent an interesting and relatively unexplored
reservoir to find sequences for orphan enzymes. Quite
often, biochemists discover a “new” activity performed
by an enzyme known to catalyze other type of reac-
tions [45]. The point is that the characterization of a
novel protein generally leads to the discovery of only
one function, but does not automatically include a
search for all possible additional functions. Nevertheless,
the characterization of supplementary in vitro activities
does not necessarily imply the elucidation of bona fide
in vivo functions.
To explore the potential promiscuity of enzymes in a
broader way, we conducted an analysis of enzyme activity/
domain associations among all known enzymes using
Pfam as a resource of domains [51]. We show that since
the 1990s and despite the increasing number of available
complete genomes in the last few years, the proportion
of newly discovered activities associated to new do-
mains (i.e. domains that were not previously associated to
an enzyme) is continuously decreasing (Figure 7). Thus,
the exploration of the functional diversity of known
enzyme domains may be a good approach for finding
proteins for new or orphan activities. Conversely, 22%
of protein domains in Pfam remains without function
and could be a reservoir of new enzyme families, con-
siderably extending the enzyme world. A recent study
successfully led to the discovery of new activities and
pathways through the exploration of the enzymatic
diversity of a protein family of unknown function [52].
On the structural side, a majority of enzyme activities
are performed by a relative small number of protein
superfamilies [53]. Indeed, we can observe an import-
ant diversity between the presence of a structural
domain and the number of potential activities: using
CATH as a resource of structural domains [54], there
Table 2 Potential candidates for local orphan enzymes retrieved by PRIAM
Archaea Bacteria Eukaryota
local orphan EC not observed EC local orphan EC not observed EC local orphan EC not observed EC
Total number 79 3774 133 1521 299 1348
Number of predictable 56 2247 115 817 150 718
Number of predicted 17 475 35 203 88 333
Percent of predicted 30% 21% 30% 25% 59% 46%
Number of candidate 400 9406 2929 11451 2996 9727
Not observed EC numbers correspond to entries than have never been associated to a protein or an organism in the superkingdom. Predictable EC numbers are
entries having an associated PRIAM profile. A predicted EC number is an entry for which PRIAM detected a significant hit with at least one protein sequence
(see Additional file 1: Figure S1.3).
Sorokina et al. Biology Direct 2014, 9:10 Page 10 of 16
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-128-2048.jpg)
![is an average of 6.37 EC numbers per CATH domain
and of 27.20 CATH domains per EC class at third-
digit. These observations reflect the importance of
convergence in the evolution of enzymes [55]. In 2010,
Omelchenko et al. found 185 enzyme activities with at
least two structurally unrelated proteins [40]. The
amount of NISE may even be revised upwards, as to
our knowledge a systematic research of all potential
structures performing the same activity has not been
carried out. These complex relations between protein
families and enzymatic activity diversity can introduce
barely detectable, but easily spreadable, misannotations
using homology based bioinformatics strategy during
the annotation process [1]. Complementary analyses
combining structural modeling, ligand docking and
active site comparisons could lead to more accurate
predictions and may open new ways to find candidate
proteins for orphan enzymes.
Conclusion
Despite an observed decrease of the number of orphan
enzyme activities over the last ten years, the orphan
enzyme challenge remains important: more than 30% of
the enzymatic activities reported in the EC classification
have no or incomplete sequence information. Though
NGS, combined with improvements in sequence analysis
methods, produces an exponential growth of genomic
data, an explosion in the number of newly discovered
activities has not occurred unlike the 80’s when the
democratization of molecular biology techniques took
place. This lack of knowledge is obviously problematic
in the overall comprehension of metabolism and in
potential biotechnological applications like biocatalysis.
As shown in our survey and as previously reported
[20], a more systematic use of comparative genomics
across superkingdoms may help to solve part of the local
orphans. For the global ones, a delay of knowledge in
databases still exists and could be resolved by intensive
bibliographical searches. In this way, the Orphan
Enzyme Project initiative [56] recently conducted a
systematic analysis of databases and publications, and
found protein sequences for about 270 presumed
orphans among an initial list of 1,122 activities established
in 2009 [16]. Similarly to what is done for protein struc-
tures with the PDB [57] and nucleic sequences by the
INSDC (International Nucleotide Sequence Database
Collaboration) [58], the design of a central and common
scientific framework to submit enzymes with their activ-
ities is of priority to reduce the loss of knowledge
between publications and databases. Indeed, collabora-
tive initiatives were recently established to discover new
activities and enzymes: the Enzyme Function Initiative
[59] which addresses the challenge of assigning reliable
functions to enzymes discovered in bacterial genome
projects, and the COMBREX project [60], connecting
computational and experimental biologists to improve
protein annotation and proposing grants to experimen-
tally validate new functions. These kinds of projects
combining in silico and wet lab strategies should lead to
a breakthrough in the discovery of new enzymes and
activities since classical sequence based methods have
lost momentum in function prediction. In fact, several
recent studies have successfully applied this approach by
exploiting mass-spectrometry or high throughput enzym-
atic assay experiments and computational methods using
sequence similarity networks, genomic contexts, structural
Figure 7 Proportion of EC activities with new protein domains. This bar plot represents the proportion of EC numbers having at least one
new Pfam domain which was never associated to any enzyme before, by year of discovery. An EC number is considered to be associated to a
new domain if this domain has never been seen associated to any other EC number discovered previously. Only EC numbers with at least one
associated sequence were taken into account.
Sorokina et al. Biology Direct 2014, 9:10 Page 11 of 16
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-129-2048.jpg)
![modeling with metabolite docking and active site com-
parison [52,61,62]. Another field of research concerns
non-protein enzymes. The most well-known are ribo-
zymes and all kinds of protein-RNA complexes, like
ribosomes, that are a real challenge to study and ex-
tremely hard to discover [63,64]. The existence of active
RNA has been known for a long time, but expertize in
this area is far from being as exhaustive as in classical
biochemistry. More recently, the discovery of a glyco-
lipid playing a “membrane protein integrase” role in
Escherichia coli has pushed back the limits of known
catalytic activities [65]. After all, not only should we
enlarge the limits of potential catalysts, but also enlarge
the limits of the known metabolites. Progress in meta-
bolomics will certainly catalyze the discovery of numer-
ous chemical compounds orphan of activities.
Reviewers’ comments
We thank the reviewers for their comments. We have
revised the manuscript taking into account their remarks.
Reviewer 1 (First Round): Dr. Michael Galperin
The paper by Sorokina et al. addresses an important
question and includes some interesting results. However,
I think that in order to justify publication in Biology
Direct, the paper needs to be much better written. The
current version is something intermediate between a
review and a regular research paper and does not make
for either a good review or a good research paper. As an
example, I would suggest moving Figure 1 to Supple-
mentary Materials (it is not a new result) and moving
Figure S2 into the main text (it is a new result).
Authors’ response: Our article is not a regular research
article but a review paper written in a format similar to
previous studies listed in Figure 1. It includes updated
analyses of existing data from public databanks that
substantially enhance our knowledge about orphan
enzymes. We thus decided not to move Figure 1 to Sup-
plementary Materials as it resumes previous studies.
Figure S2 (now S1.2) is an estimation of orphan enzymes
at the superkingdom level based on non-curated data
from FRENDA and AMENDA whereas Figure 6 was
made using manually curated data. Therefore, we prefer
not to move Figure S1.2 to the main text.
In addition, I am afraid that the current version of the
manuscript does not really benefit the scientific community
as it simply enumerates the enzymes in each category
without providing the specific lists of these enzymes. I
could support publication of this paper only after the
authors include (at least as Supplementary Materials) the
lists of global and local orphans from Figure S2. Unless
this is done, the data in Figures 2, 3 and 4 cannot be
independently verified and the entire manuscript cannot
be considered acceptable for publication.
Authors’ response: We added the lists of global and
local orphans and proteins in Supplementary Materials
2 and 3.
Finally, the entire paper looks like a promotion for the
Orphan Enzymes Project [http://www.orphanenzymes.
org, ref. 49]. However, according to the Orphan Enzymes
web site, this project is also the subject of an upcoming
paper “Finding sequences for over 270 orphan enzymes”
(currently in press). The reviewers should have been
provided the text of that other paper to ensure that there
was no significant overlap between the two.
Authors’ response: We have no relation or contact with
the Orphan Enzymes Project and had not access to their
upcoming paper at the time of writing the present article.
This article is now published and sentences were included
in the main text to present their work.
To help revision of this manuscript, I provide below
some specific examples of the poorly formulated sen-
tences. However, the entire text must be carefully revised
and made less descriptive and more concise.
1. The Abstract needs to be revised to clearly explain
what are the new results communicated in this
work. Right now, the new results seem to start from
“Besides, we extended our study”? Please rewrite the
first 4 sentences of the Abstract to explain what
exactly was the goal of this work and what exactly
has been done.
2. The statement in the Abstract “We developed a
simple strategy to rescue these local orphan
enzymes” is totally enigmatic and has to be deleted
or reformulated.
3. The last sentence of the Abstract does not seem
relevant to the rest of the text. Please either delete
or at least reformulate.
Authors’ response: Part of the abstract has been
rewritten according to the reviewer suggestions.
4. The Introduction could (and should) be made more
compact and succinct. That said, the last paragraph
of the Introduction contains a much better
description of the work presented in this paper than
the Abstract does.
Authors’ response: We removed the definition of the
EC nomenclature but we think that it is important to
keep a description of previous analysis reviews on
orphan enzymes in the introduction.
5. Citations of the enzyme and EC number databases
in the Introduction and other sections of the paper
present are unfortunately biased. The authors
should, at the very least acknowledge the official
web sites of the EC classification, the IUBMB list
(http://www.chem.qmul.ac.uk/iubmb/enzyme/) and/
or the ExplorEnz (http://www.enzyme-database.org,
PMID: 18776214) as well as the ENZYME database
Sorokina et al. Biology Direct 2014, 9:10 Page 12 of 16
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-130-2048.jpg)
![(http://www.expasy.org/enzyme/ PMID: 10592255),
That would also make it unnecessary to explain the
organization of the EC system in the Introduction
section. INSDC should be cited (PMID: 23180798).
The section on Enzyme promiscuity should probably
mention the availability of the MultiTaskDB (http://
wallace.uab.es/multitask/, PMID: 24253302).
Authors’ response: Suggested references have been
added.
Reviewer 2 (First Round): Dr. Daniel Haft
The manuscript submission by Sorokina et al., “Profiling
the Orphan Enzymes”, functions fairly well as a review
article on the chronology of the growth of EC numbers
with and without associations with specific sequences.
The authors define a problem space - identifying enzymes
that have no representative in some superkingdom -. They
introduce a strategy for generating lists of candidate
sequences to fill the void. The revised form of the manu-
script now provides lists of these candidate sequences in
supplementary materials, rather than their count only, and
it clearly warns that the associations offered by their tech-
nique are in no way validated.
The strategy relies on PRIAM, an update from March
2013. But there is no discussion of how PRIAM itself is
formed and whether its design could be appropriate to
the task. PRIAM was described in 2003, and relies on
MKDOM. Therefore, PRIAM requires an unsupervised
domain definition algorithm to find signature regions
one enzyme has but another enzyme lacks. The domain
could be a C-terminal extension with no relevance to
enzyme function, and could be eukaryotic only, but
PRIAM would make it a signature. Should this method
be used to identify probable “local orphan enzymes” in
the archaea? Not without validation.
Other homology strategies might do as well PRIAM or
better, such as searching for bi-directional best BLAST
hit matches that link a known exemplar of enzyme func-
tion in one superkingdom to a homolog in another
superkingdom. The PRIAM strategy itself could have
been benchmarked somewhat be seeing how much its
predictions vary from one version to the next. Readers
are strongly cautioned that the output from the PRIAM
strategy should be viewed only as anecdotal evidence,
appropriate to a review article, that simple homology
methods could generate lists of sequences that contain
candidates to represent the first extension into a new
superkingdom of enzymatic activities that have been
assigned to sequences in other superkingdoms.
Authors’ response: This strategy is not a methodo-
logical development but just a way to estimate if candi-
date proteins for local orphans could be retrieved by
homology search. We agree that PRIAM profiles have
limitations but, as far as we know, it is one of the best
tools to track potential conserved domains which are
enzyme specific and have a wide coverage of Swiss-Prot
enzymes. BBH cannot be computed for all the Swiss-Prot
enzymes as many of them are not from complete organ-
isms. As mentioned in the manuscript: “these [PRIAM]
predictions cannot be transferred directly without supple-
mentary bioinformatics analyses or experiments”.
As a review, the manuscript did not do justice to the
methods that might be used to find orphan enzymes in
general, or domain orphans. In particular, Yamada et al.
(ref 27) struck me as a landmark demonstration of data
mining combined with comparative genomics for finding
complete sequence orphans. The method would work
even better for superkingdom orphans. Because that
work followed predictions with validations, it represents
a standard that should be discussed in any review article
on matching sequences to orphan EC numbers.
Authors’ response: We introduce the main methods of
finding candidate genes for global or local orphans and
some of their limitations. But, we do not wish to develop
more deeply these methods for three reasons: (1) a
complete review of these methods would require a dedi-
cated article (2) a methodological review should be done
by a third party since authors of the paper are involved in
methodological developments on this topic (i.e. the CANOE
method was published the same year as Yamada et al.
paper) (3) a review has recently been published and
presents a practical description of these methods (El
Yacoubi et al. 2014, a reference to this paper was added
in our article). For information, the two experimentally
tested enzymes in Yamada et al. are not supported by
enough evidence to validate that they are good can-
didates for the two orphan activities: (1) the two tested
activities are amino acid transaminases, which are
known to have in vitro substrate promiscuity (2) the can-
didate protein (UniProt AC Q8R5Q4) for the histidine
transaminase activity has a TIGRFAM result corre-
sponding to HisC protein (TIGR01141), which catalyzes
the transamination of imidazole acetol-phosphate in the
context of the histidine biosynthesis. Furthermore, the
corresponding gene (TTE2137) is in the hisGDCBHAFI
operon confirming that this protein should be involved
in the histidine biosynthesis and not in the degradation
process via the histidine transaminase activity. (3) the
candidate protein (UniProt AC Q8DTM1) shares more
than 50% of amino acid identity with biochemically
characterized aspartate aminotransferases (UniProt ACs
P23034, Q59228). This activity is more coherent with
the asparaginyl tRNA synthetase genomic context than
the asparagine aminotransferase activity proposed by
Yamada et al., an activity described only in eukaryotes
for asparagine degradation. These two cases are really
good examples to illustrate the difficulty in interpreting
in vitro activities to elucidate bona fide in vivo functions.
Sorokina et al. Biology Direct 2014, 9:10 Page 13 of 16
http://www.biologydirect.com/content/9/1/10](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-131-2048.jpg)





![116
Chapitre II
Construction d’un modèle réduit du
métabolisme pour l’identification de
modules conservés
Le métabolisme est très souvent représenté informatiquement sous la forme d’un réseau. Le
choix du type de réseau (réseau de composés, réseau de réactions, réseau biparti ou autre) dépend
forcément du but de l’analyse, et de ce que l’on veut découvrir ou mettre en évidence.
L’hypothèse principale qui a orienté les développements décrits dans ce chapitre est la
conservation d’enchainements de transformations chimiques au cours de l’évolution. Le but ici
est d’identifier des ensembles de transformations chimiques conservés et éventuellement inédits
qui peuvent servir de base pour la découverte de nouvelles voies métaboliques.
La première étape a été de construire un réseau de réactions rassemblant toutes les réactions
connues et présentes dans au moins une voie métabolique de la base de données généraliste
MetaCyc [91]. Seules les réactions décrites dans une voie métabolique ont une définition de
composés chimiques « primaires » et « secondaires ». Cette information est nécessaire pour ne pas
relier deux réactions entre elles via des métabolites secondaires, qui sont souvent des cofacteurs
ubiquitaires. Dans ce réseau, deux réactions sont reliées entre elles si il existe un métabolite
primaire produit par une et consommé par l’autre. Il s’agissait avant tout de construire un réseau
regroupant toutes les connaissances disponibles sur le métabolisme, indépendamment de la
notion d’organisme ou d’espèce.
Ce réseau orienté de réactions, construit à partir de données de MetaCyc, contient environ 6 000
nœuds et 11 000 arcs. Il a un diamètre (distance maximale parmi les distances entre toutes les
paires de nœuds dans le graphe) de 47 ce qui est relativement faible et montre la relativement
forte connectivité des nœuds dans ce réseau (Figure 24) On y retrouve cependant un grand](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-137-2048.jpg)



![120
encodées par chaque RMS. Parmi les centralités globales, celle qui a été envisagée en premier lieu
est la centralité « betweenness » car elle représente la quantité d’information qui passe par chaque
nœud du réseau, ce qui pourrait s’apparenter aux flux d’atomes de carbone lors des
transformations chimiques, par exemple. Elle n’a toutefois pas été retenue car, paradoxalement,
elle est trop globale. En effet du point de vue biologique, un flux d’atomes de carbone décrit dans
les voies métaboliques est en général inférieur à une dizaine de réactions. Nous avons aussi essayé
de calculer la centralité betweenness pour chaque nœud sur un sous-graphe de diamètre 10
autour de ce nœud. Cette technique ne donnait pas de résultats significativement différents de la
centralité betweenness globale et résultait aussi en la perte du sens même apporté par cette
centralité. Nous nous sommes alors tournés vers les centralités dites de « hubs et d’autorités »,
très utilisées dans les analyses de réseaux sociaux et dans les réseaux de pages web. Le principe de
ces centralités est assez simple : un nœud qui pointe vers un grand nombre d’autres nœuds (qui a
un degré sortant assez grand) est un hub. Par exemple, les pages web annuaires, populaires dans
les années 1990 et début 2000, et qui ont pour seul but de pointer vers d’autres pages web
(souvent contre rémunération et/ou pour des raisons commerciales ou frauduleuses), sont des
hubs. En contrepartie, un nœud qui est pointé par beaucoup d’autres nœuds (qui a un degré
entrant important) est une autorité. C’est le cas par exemple de pages Wikipédia populaires. Parmi
les différentes centralités suivant le principe des hubs et des autorités, la centralité Page Rank
[133] a été retenue ici. Cette centralité est à la base du célèbre moteur de recherche Google et
apporte une amélioration à la notion d’autorité : plus un nœud est influent (plus son autorité est
grande) plus ses voisins directs sortants sont influents (les amis des personnes influentes sont
influentes). On parle aussi de centralité « feedback ». Dans ce cas présent, cette particularité est
intéressante, car elle permet de propager l’importance d’un nœud, et peut faire ressortir plus
naturellement les chemins dans lesquels des nœuds importants du point de vue topologique se
succèdent. Les centralités basées sur la marche aléatoire, comme le « web surfer » ou la centralité
de Markov n’ont pas été essayées, mais, avec du recul, elles ne sont pas aberrantes et pourraient
avoir un sens intéressant dans le contexte du réseau métabolique de transformations chimiques.
Un certain nombre de chemins conservés de transformations chimiques ont été identifiés grâce
aux trois scores. Certains de ces chemins font partie de voies métaboliques connues, d’autres ne
correspondent à rien de connu pour le moment, et restent donc à analyser.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-141-2048.jpg)
![Sorokina et al. BMC Bioinformatics (2015) 16:385
DOI 10.1186/s12859-015-0809-4
RESEARCH ARTICLE Open Access
A new network representation of the
metabolism to detect chemical transformation
modules
Maria Sorokina1,2,3*, Claudine Medigue1,2,3 and David Vallenet1,2,3
Abstract
Background: Metabolism is generally modeled by directed networks where nodes represent reactions and/or
metabolites. In order to explore metabolic pathway conservation and divergence among organisms, previous studies
were based on graph alignment to find similar pathways. Few years ago, the concept of chemical transformation
modules, also called reaction modules, was introduced and correspond to sequences of chemical transformations
which are conserved in metabolism. We propose here a novel graph representation of the metabolic network where
reactions sharing a same chemical transformation type are grouped in Reaction Molecular Signatures (RMS).
Results: RMS were automatically computed for all reactions and encode changes in atoms and bonds. A reaction
network containing all available metabolic knowledge was then reduced by an aggregation of reaction nodes and
edges to obtain a RMS network. Paths in this network were explored and a substantial number of conserved chemical
transformation modules was detected. Furthermore, this graph-based formalism allows us to define several path
scores reflecting different biological conservation meanings. These scores are significantly higher for paths
corresponding to known metabolic pathways and were used conjointly to build association rules that should predict
metabolic pathway types like biosynthesis or degradation.
Conclusions: This representation of metabolism in a RMS network offers new insights to capture relevant metabolic
contexts. Furthermore, along with genomic context methods, it should improve the detection of gene clusters
corresponding to new metabolic pathways.
Keywords: Metabolic network, Reaction signatures, Graph reduction, Pathway conservation, Chemical
transformation modules
Background
In bioinformatics, metabolism is generally modeled
by directed networks where nodes represent reactions
and/or metabolites and edges the product/substrate
exchanges between reactions [1]. Metabolic network
reconstruction of a given organism generally starts with
its genome annotation that predicts enzymatic activities
from coding sequences and, therefore, the correspond-
ing reactions and metabolites of the network. However,
*Correspondence: msorokina@genoscope.cns.fr
1Direction des Sciences du Vivant, Commissariat à l’Energie Atomique et aux
Energies Alternatives (CEA), Institut de Génomique, Genoscope, Laboratoire
d’Analyses Bioinformatiques pour la Génomique et le Métabolisme, 2 rue
Gaston Crémieux, 91057 Evry, France
2CNRS-UMR8030, 2 rue Gaston Crémieux, 91057 Evry, France
Full list of author information is available at the end of the article
two main bottlenecks limit today this reconstruction by
homology: the difficulty in associating correct functions
to genes and the lack of experimental characterization
of enzyme activities for which proteins are sometimes
unknown, i.e. orphan enzymes [2].
Subgraphs of these networks are often used to repre-
sent metabolic pathways that group sets of connected
reactions involved in a same biological process. Sev-
eral hypotheses on the origin and evolution of metabolic
pathways have been proposed, including patchwork evo-
lution by enzyme recruitment in new metabolic path-
ways [3, 4], retrograde synthesis which postulates that
metabolic pathways are constructed starting from the
final metabolite [5], and the theory on metabolic path-
way duplication [6]. Despite their differences, these
© 2015 Sorokina et al. Open Access This article is distributed under the terms of the Creative Commons Attribution 4.0
International License (http://creativecommons.org/licenses/by/4.0/), which permits unrestricted use, distribution, and
reproduction in any medium, provided you give appropriate credit to the original author(s) and the source, provide a link to the
Creative Commons license, and indicate if changes were made. The Creative Commons Public Domain Dedication waiver
(http://creativecommons.org/publicdomain/zero/1.0/) applies to the data made available in this article, unless otherwise stated.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-142-2048.jpg)
![Sorokina et al. BMC Bioinformatics (2015) 16:385 Page 2 of 9
hypotheses agree about the importance of enzyme
promiscuity in the evolution of metabolic pathways, i.e.
the capacity of enzymes to catalyze one or several types
of reactions on more or less different substrates. A recent
study in Escherichia coli successfully brings out this
enzyme capacity to adapt themselves to new substrates [7].
In order to explore metabolic pathway conservation
and divergence among organisms, previous studies were
based on pathway alignment to find similar pathways
within or between organisms using the Enzyme Commis-
sion (EC) numbers to define reaction similarities [8–11].
Due to limitations of the EC classification, the notion of
reaction similarity for pathway alignment was improved
using metabolite similarity [12] or substructure changes
[13]. Another approach, that does not require prede-
fined pathways, was based on the detection of motifs
in a reaction network [14]. Few years ago, the concept
of chemical transformation modules, also called reaction
modules, was introduced by Muto et al. [15]. They cor-
respond to sequences of chemical transformations which
are conserved in metabolism. These modules capture the
chemical logic of pathways that may correspond or not to
conserved sets of enzymes. Muto et al. made a systematic
analysis of the conservation of reaction modules by align-
ing metabolic pathways from KEGG [16] and used RClass
(Reaction Class) [17] to group reactions having same pat-
terns of chemical transformations. The same year, Barba
et al. [18] published a study on the modularity of the
purine and pyrimidine metabolism, which presents chem-
ical reaction similarities, and also enriched the reaction
module definition with the notion of enzyme homology.
In the present work, we propose a different formalism
for the detection of reaction modules, although we use the
same definition of modules as Muto et al. [15]. Instead of
using pathway alignment, we adopt an innovative graph
representation of the metabolism where the reaction net-
work is reduced in a Reaction Molecular Signature (RMS)
network. For that, RMS are automatically computed for
all reactions and encode changes in atoms and bonds as
described in [19]. Thereby, reactions sharing a same sig-
nature are grouped together. Paths in the RMS network
are then explored to detect conserved modules. Further-
more, this graph-based formalism allows us to define
several path scores reflecting different biological conser-
vation meanings. These scores are finally analyzed for all
possible paths in the network and for known metabolic
ones and used to build association rules that should pre-
dict metabolic pathway types like metabolite biosynthesis
or degradation.
Methods
Reaction network
Metabolic data was extracted from MetaCyc public
database version 19.0 [20]. MetaCyc contains a large
collection of curated metabolic pathways from all domains
of life. In addition, metabolites, reactions, enzymes and
genes are also listed. Metabolic pathways described in
MetaCyc are generally short (4.3 reactions on average)
and have been experimentally elucidated in at least one
organism. A metabolic network was reconstructed using
MetaCyc reactions as nodes. We linked two reactions by
a directed edge when the product of one reaction is the
substrate of the other one. However, to avoid the high con-
nectivity problems that are common when building such
metabolic networks, we limited shared compounds to
“main compounds”, i.e. metabolites deemed biologically
relevant to both reactions in at least one metabolic path-
way. Only reactions that belong to a metabolic pathway
were taken into account, as only these ones have dis-
tinction between main metabolites and co-substrates sup-
porting the reaction such as water, ATP or NAD. Trans-
port reactions, for which translocated substrate remains
unchanged, were excluded from the network construction
and from further analysis, e.g. ABC transporter ATPase
reactions corresponding to 3.6.3.- EC class.
Reaction molecular signatures
Reaction Molecular Signatures (RMS) were computed for
all MetaCyc reactions, belonging or not to a metabolic
pathway, as described in [19]. These signatures encode
changes in atoms and bonds where the reaction is tak-
ing place. First, structures of all molecules involved in
a reaction were downloaded from MetaCyc website in
MDL Molfile format. Using ChemAxon MolConvert soft-
ware [21], all molecules were standardized by adding
implicit hydrogen atoms and applying aromatization
when needed. Stereo signature molecular descriptors [22]
were then computed for heights 1 and 2 with the MolSig
software (http://molsig.sourceforge.net). These molecu-
lar signatures are encoded using SMILES-like strings [23]
and the height parameter corresponds to a distance for
the inclusion of neighbour atoms and bonds up from
a given atom. Second, corresponding RMS were gener-
ated for each molecular signature height by calculating
the difference between the signatures of the products
and of the substrates. To obtain correct RMS, reaction
equations have to be balanced with explicit compounds
for which Molfile structures are available. It should be
noticed that (i) for a given height, a reaction has only
one RMS signature (ii) reactions sharing a same RMS
have similar chemical transformations (iii) the higher
the height value is more the signature is precise. RMS
of height 1 (RMS-H1) capture the reaction center with
atom and bond changes. To compute RMS of height
2 (RMS-H2), RMS-H1 were partitioned in sub-groups
having similar signatures at height 2. Distances between
signatures were computed using an approximate string](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-143-2048.jpg)
![Sorokina et al. BMC Bioinformatics (2015) 16:385 Page 3 of 9
matching algorithm [24]. Then, a hierarchical clustering
was build on these distances using the Ward algorithm
[25] and the tree was cut at a cophenetic distance thresh-
old of 90. To deal with reaction directionality, RMS hav-
ing strictly opposite signatures were merged in a single
entry. Higher values of the height parameter were not
used because they lead to too precise signatures with
many describing only one reaction. The RMS classifica-
tion of reactions is available in Additional file 1 and the
source code for the RMS computation was deposited in
GitHub (https://github.com/mSorok/createRMS.git). The
RMS method has been chosen in this work as it guarantees
that all reactions described by the same signature per-
form the same chemical transformation, making manual
post-process unnecessary.
RMS networks
The reaction network was reduced in a directed net-
work of chemical transformations represented by RMS.
As shown in Fig. 1, reactions signed by the same RMS
are grouped in a single node. Two RMS are connected
by a directed edge in the RMS network if there is at
least one edge in the original reaction network linking
reactions signed by the corresponding source and tar-
get RMS. For computational complexity reasons and the
lack of explicit representation of repeated reactions in
pathway databases, edges are not created if source and
target RMS are identical (i.e. self-loops are avoided). This
transformation was made for the two RMS heights and
we obtained two networks called RMS-H1 and RMS-
H2 networks. Furthermore, this graph reduction, which
aggregates reaction nodes and edges, allowed us to define
Markov chains transition probabilities of order 1 between
connected RMS. Pr RMSj | RMSi is calculated as the
ratio of the number of outgoing reaction edges linking
RMSi to RMSj among the total number of outgoing edges
from reactions signed by RMSi.
RMS node weighting
Several weights, reflecting different biological conserva-
tion meanings, have been computed on nodes of the
RMS networks. The first weight, wRea, corresponds to the
number of MetaCyc reactions associated to a given RMS,
whether they are present or not in the initial reaction net-
work. It gives a quantitative measure of the diversity of
reactions represented by a RMS.
A second weight, wPageRank, is computed using
PageRank algorithm [26] implemented in the Jung 2.0
Java library [27]. This topological weight is based on a
network architecture exploration in order to locate influ-
ential nodes in the RMS network with the assump-
tion that most important chemical transformations
are likely to have more incoming links from other
transformations.
The last weight, wProt, is an estimation of the num-
ber of proteins associated to a given RMS. Known pro-
tein/reaction associations were extracted directly from
MetaCyc and from Swiss-Prot using EC numbers [28].
These associations were used to compute two ratios cor-
responding to the number of known proteins with the
same Pfam domain composition [29] and associated to
a given RMS Np(p ∈ RMSi p ∈ Domj) divided by
the total number of known proteins having the domains
Np(p ∈ Domj), for d2r ratio, or by the total number of
Fig. 1 Reaction network to Reaction Molecular Signature network. This figure presents a toy example of the reduction of a reaction network in a
RMS network. Reactions sharing a same reaction signature (same node color in the figure) are grouped in a single RMS node. Directed edges of the
reaction network are also merged in the RMS network. Red edges illustrate the computation of Markov transition probabilities Pr(RMS2 | RMS1),
Pr(RMS3 | RMS1) and Pr(RMS5 | RMS1). They correspond to the proportion of reaction edges, among the five outgoing edges of RMS1 reactions
(blue nodes), connecting RMS1 to RMS2, RMS3 and RMS5](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-144-2048.jpg)
![Sorokina et al. BMC Bioinformatics (2015) 16:385 Page 4 of 9
known proteins associated to the RMS Np( p ∈ RMSi), for
r2d ratio.
d2r(RMSi, Domj) =
Np( p ∈ RMSi p ∈ Domj)
Np( p ∈ Domj)
(1)
r2d(RMSi, Domj) =
Np( p ∈ RMSi p ∈ Domj)
Np( p ∈ RMSi)
(2)
Next, the association score, score(Dom, RMS), was com-
puted as the harmonic mean of d2r and r2d values. This
score represents a trade-off between sensitivity and speci-
ficity to associate protein domains to chemical transfor-
mations and tends to be very low when domains or RMS
are very frequent.
score(Domj, RMSi) =
2 × d2ri,j × r2di,j
d2ri,j + r2di,j
(3)
Finally, wProt is, for each protein domain associated to
the given RMS, the geometric mean of the total num-
ber of UniProt proteins associated to a domain multiplied
by the score(Dom, RMS). Only proteins from UniProt
reference proteomes [28] (version 2015_04 with 2,424
reference proteomes) were considered to provide broad
coverage of the tree of life while reducing taxonomic
over-representation.
wProt(RMS) = n
n
j=1
Np( p ∈ Domj) × score(Domj, RMS)
(4)
This weight gives a quantitative measure of the diver-
sity of enzymes associated to a RMS. High value of wProt
may indicate that the chemical transformation is widely
represented among organisms and/or that many enzymes
catalyze this transformation because of many gene dupli-
cations or many enzyme families.
RMS path enumeration and scoring
An enumeration of all paths of length 1 (one edge and
two RMS nodes) to 4 (four edges and five nodes) was
made in both RMS networks using the Grph Java library
[30]. In this path enumeration, loops were not allowed (i.e.
a node cannot be found more than once in a path). To
make them comparable, metabolic pathways from Meta-
Cyc were translated in overlapping RMS paths of the same
length. In addition, a Pathway Conservation Index (PCI)
was computed for each RMS path and represents the
number of distinct corresponding reaction paths that are
present in at least one MetaCyc pathway.
According to previously defined RMS weights, path
conservation scores, named scoreRea, scorePageRank and
scoreProt, were calculated as the geometrical means of
path node weights multiplied by their probability of tran-
sition to the next node of the path. As an illustration, the
formula of scoreRea is given in which RMSi and RMSi+1
are two consecutive nodes and n is the path length.
scoreRea(RMSs → RMSn) (5)
= n−1
n−1
i=s
wRea(RMSi) × Pr (RMSi+1 | RMSi)
ScorePageRank and scoreProt are computed in the same
way using wPageRank and wProt, respectively.
Results and discussion
From reaction to RMS networks
Among the 12,377 MetaCyc reactions, RMS of of height
1 (RMS-H1) and 2 (RMS-H2) have been computed for
9,001 reactions excluding transport reactions and reac-
tions without proper compound structures as described
in the Methods section. As shown in Table 1, RMS-H1
gathers on average about two times more reactions than
RMS-H2. Indeed, RMS-H2 signatures give more precision
about the chemical transformations than RMS-H1 as they
encode additional information about the neighborhood of
the reaction center that may be important for the chemical
reactivity.
This fully automated chemical classification of reac-
tions was compared with the Enzyme Commission (EC)
classification which is a human expertise classification of
enzymatic activities [31]. Even if efforts were made to
automate the classification of new activities [17, 32, 33],
the EC classification covers only half of all known enzy-
matic reactions. Among the 4,574 reactions linked both
to an EC number and to a RMS, a simple similarity mea-
sure (Rand index) was computed between the third level
sub-subclasses of EC numbers (179 classes) and the RMS-
H1 (1,437 classes). We obtained a Rand index value of
97.68 % meaning, even if the RMS classification has a
finer granularity, both classifications are thus similar (see
Additional file 2 for detailed counts). Reactions classified
in a same RMS tends to have the same third level EC
class. Nevertheless, we found cases where the two clas-
sifications differs such as the example depicted in Fig. 2.
From a chemical point of view, the D-glutamate cyclase
and the L-lysine-lactamase reactions correspond to the
formation or the hydrolysis of a lactam involving a pri-
mary amine and the carbon of the keto function of a
Table 1 Reaction molecular signature statistics
Height 1 Height 2
Number of RMS 2477 4775
Number of reactions by RMS
Minimum 1 1
Average 3.63 1.89
Maximum 312 144](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-145-2048.jpg)
![Sorokina et al. BMC Bioinformatics (2015) 16:385 Page 5 of 9
Fig. 2 Example of reactions having a same RMS signature but classified in different EC classes. a D-glutamate cyclase reaction annotated with the
EC 4.2.1.48. b L-lysine lactamase reaction annotated with EC 3.5.2.11. This both reactions make the same the chemical transformation represented
by RMS-H1.1372, which encodes, in SMILES-like strings, the difference between the products and the substrates of atomic signatures of height 1
carboxylic acid. These reactions are encoded by the same
RMS but their EC classes differ: the D-glutamate cyclase is
classified as a carbon-oxygen lyase (EC number 4.2.1.48),
whereas the L-lysine-lactamase is a hydrolase acting on
a carbon-nitrogen bond of a cyclic amide (EC number
3.5.2.11). These differences show that EC numbers are
mainly focused on enzymatic activities and take in consid-
eration the biological context to classify the reactions (e.g.
the in vivo reaction directionality). These ambiguities, that
are quite common between lyases and hydrolases or trans-
ferases, were also previously reported in other chemical
classifications of reactions like MOLMAP [34].
Finally, an initial reaction network was established using
metabolic pathway information from MetaCyc. It is made
of 5,830 reaction nodes and 11,197 directed edges with
an average node degree of 2.6. This graph was reduced
in two RMS networks using RMS-H1 and H2 signatures.
As summarized in Table 2, RMS networks are more com-
pact than the reaction network: RMS-H1 and RMS-H2
networks contain a third and a half of nodes, respectively.
Table 2 Statistics on reaction network and RMS networks
Reaction RMS-H1 RMS-H2
network network network
Number of nodes 5830 1768 3365
Number of edges 11197 6107 8721
Average node degree 5.17 9.10 3.33
Average node out degree 2.60 4.36 2.99
Average node in degree 2.27 3.94 6.84
Node reduction rate 1 0.30 0.57
By aggregating reactions in RMS nodes while preserv-
ing their initial connectivity, RMS graph structure should
efficiently capture conserved paths of chemical reactions
even for reactions not already associated to a metabolic
pathway. Indeed, 2,278 reactions not included in the initial
reaction network are linked to a chemical transformation
context in the RMS networks since they are classified
in the RMS networks with other reactions from known
pathways.
Conserved RMS paths in metabolic pathways
An exploration of the RMS networks was conducted by an
enumeration of all paths of length 1 (one edge, two RMS)
to 4 (four edges, five RMS). To evaluate their conservation
in the light of known metabolic pathways, a Pathway Con-
servation Index (PCI) was computed for each RMS path
and corresponds to the number of distinct reaction paths
present in MetaCyc pathways. The number of RMS paths
with a PCI ≥2 is reported in Table 3 for each path length
and for both signature heights. We found, for RMS-H1,
between 117 and 600 conserved RMS paths depending of
the path length and fewer paths (between 128 and 380)
for RMS-H2 as they encode more precise signatures (see
Additional file 3 for the complete list). They correspond to
Table 3 Number of conserved modules (PCI ≥ 2)
Path length RMS-H1 network RMS-H2 network
1 600 380
2 365 214
3 212 141
4 117 128](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-146-2048.jpg)
![Sorokina et al. BMC Bioinformatics (2015) 16:385 Page 6 of 9
conserved chemical transformation modules, also named
reaction modules in a previous study [15]. Indeed, Muto
et al. obtained similar results but with a higher num-
ber of detected conserved paths (between 338 and 928
for the same path lengths). Although our results are not
directly comparable to those of Muto et al. by the usage of
different primary data sources (i.e. MetaCyc and KEGG,
respectively), the RMS paths detected by our method can
be directly considered as conserved modules whereas the
paths obtained by Muto et al. need a manual examina-
tion to obtain conserved modules from them. In fact, they
adopted a looser definition of chemical conservation with-
out taking into account side compounds and using finger-
print similarities to group reactions without the constraint
that the reactions perform the same chemical transfor-
mation. Only 34 reaction modules were finally confirmed
by the authors [15]. Among the modules detected by our
method, we found, for instance, that the β-oxidation path-
way, that is well-known for fatty acid degradation, is also
conserved for other molecule types (Fig. 3). This module,
also detected by Muto et al. for a subset of compounds
(two among eight), has four reaction variants in its first
step. As another example, we detected a new three-step
module for the biosynthesis of aldoximes from amino
acids, which are notably precursors of several secondary
metabolites produced by plants (Fig. 4). More generally,
nearly half (48 %) of metabolic pathways contains at least
one conserved module in the height 1 RMS network (see
Table 4). Interestingly, pathways involved in the genera-
tion of precursor metabolites and energy (‘Energy’ type in
Table 4) are the most conserved (78 % of them in RMS-H1
network). Besides, the proportion of conserved pathways
involved in biosynthesis and degradation is also important
and comparable for both types, 42 % and 47 % respectively.
RMS path scoring and learning
To go further, our method proposes an evaluation of
chemical module conservation in the metabolism using
three scores corresponding to different biological points
of view. Indeed, scoreRea reflects the diversity of reac-
tions performing the same chemical transformations on
different substrates, scoreProt represents the conservation
of enzymes performing these chemical transformations
across the tree of life and scorePageRank shows the topo-
logical importance of the module in the network by high-
lighting chemical hubs. These scores were computed for
all paths and analyzed more precisely for paths of length
2 in the RMS-H2 network (Table 5). It should be noticed
that the scoreProt cannot be computed for about 20 %
of paths as they contain at least one RMS without any
known protein catalyzing the corresponding reactions, i.e.
30 % of the RMS-H2 correspond to orphan enzyme activ-
ities. As depicted in Fig. 5, paths from known metabolic
pathways present statistically significant higher values for
the three scores than in all possible paths computed from
the RMS network (p-value < 2e−16 using Tukey’s HSD
tests). Similar results were obtained for RMS-H1 net-
work (see Additional file 4). These results confirm that
the defined scores are useful to capture biologically rel-
evant paths in the RMS network and should allow us to
discover new metabolic modules. Furthermore, we found
only a weak correlation between scoreRea and scorePageR-
ank (Spearmans’ correlation coefficient of 0.66) and
no correlation between other pairs of scores. There-
fore, the proposed scores can be considered as rather
independent and then used conjointly to explore the
RMS network.
Next, these scores were analyzed in the light of
MetaCyc pathway classification using five main types
Fig. 3 Conservation of β-oxidation module for non-fatty acid compounds. In addition to fatty acids, the β-oxidation module was found conserved
for the transformation of 8 compounds represented in the figure. For the first step, we found 4 reaction variants encoded in different RMS of height
1: three RMS correspond to a dehydrogenation between the alpha and beta carbons but with different acceptors, another corresponds to a
coenzyme A ligation. A color code indicates the corresponding substrates. Only molecules marked with an asterisk were also detected by Muto et al.
(KEGG Reaction Module RM018)](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-147-2048.jpg)
![Sorokina et al. BMC Bioinformatics (2015) 16:385 Page 7 of 9
Fig. 4 A conserved module for the biosynthesis of aldoximes from amino acids. a This module is made of three chemical transformations encoded
by RMS-H2 signatures. It corresponds to the oxidative decarboxylation of an anmino acid to its aldoxime. b The module is conserved in different
MetaCyc pathways for five distinct proteinogenic amino acids. Produced aldoximes are precursors of nitrogen-containing secondary metabolites in
plants, like cyanogenic glycosides for seed germination and defense, or auxin phytohormones
of biological processes: biosynthesis, degradation/
utilization/assimilation, detoxification, generation of
precursor metabolites and energy, and a last type, called
“others”, that gathers other MetaCyc main pathway
classes. By performing pairwise comparisons of pathway
types (i.e. Kruskal-Wallis rank sum tests completed by
post-hoc Tukey’s HSD tests, see Additional file 5), we
found significant differences (p-values < 0.05) among all
pathway types for at least one of the three conservation
scores. These results presume that pathway types could
Table 4 Number of pathways containing at least one conserved
module (length 2, PCI ≥ 2) classified by their type
Pathway type RMS-H1 network RMS-H2 network
Biosynthesis 263 (42 %) 154 (24 %)
Degradation 172 (47 %) 95 (25 %)
Detox 3 (27 %) 3 (23 %)
Energy 61 (78 %) 51 (65 %)
Other 19 (33 %) 10 (17 %)
All 518 (46 %) 313 (27 %)
be predicted by machine learning using a combination of
the three scores. Thus, pathway assignment rules were
generated with the NNge algorithm [35, 36] implemented
in Weka [37]. As the number of RMS paths per pathway
type is very unbalanced (e.g. the “biosynthesis” class
contains almost twice the number of paths than other
Table 5 Statistics on conservation scores for paths of length 2 in
the RMS-H2 network
ScoreRea ScorePageRank ScoreProt
All enumerated
paths (n = 72173)
Min score 0.04 3.32e−6 4.39e−4
Average score 0.61 7.69e−5 25.17
Max score 17.58 1.20e−3 3913.24
Paths in known
pathways (n = 3001)
Min score 0.04 8.63e−6 7.81e−4
Average score 1.07 1.55e−4 118.57
Max score 17.58 1.20e−3 3913.24](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-148-2048.jpg)
![Sorokina et al. BMC Bioinformatics (2015) 16:385 Page 8 of 9
Fig. 5 Boxplots of conservation scores for enumerated and known metabolic paths. For paths of length 2 (two edges and three nodes) in the
RMS-H2 network, distributions of the three conservation scores (i.e. scoreRea, scoreProt and scorePageRank) are presented in all possible paths from
the RMS network (identified as “All paths” in the figure) versus paths solely included in known metabolic pathways (“Known metabolic pathways”).
The latter present significant higher scores (p-value < 2e−16 using Tukey’s HSD tests)
types), classes were virtually balanced using resampling
function of Weka. We successfully obtained rules that
correctly classify RMS paths in pathway types with an
accuracy greater than 89 % (see Additional file 6).
Conclusions
We present here a novel metabolic network repre-
sentation where nodes are chemical transformations
depicted by reaction molecular signatures. This data
model is particularly useful for finding conserved chemi-
cal transformation modules in metabolic pathways as they
correspond to paths in the RMS network. An impor-
tant number of modules was detected and could be
integrated in metabolic databases, like KEGG [16] or
MetaCyc [20], to help biologists looking for similar path-
ways. Furthermore, new metrics (i.e. scoreRea, scoreProt
and scorePageRank) were introduced to evaluate module
conservation according to different biological meanings.
We show that known metabolic paths present higher score
values than random ones and that the scores, used con-
jointly, may predict module pathway types. In terms of
improvement of the graph reduction method, it may be
of interest to dynamically adapt the precision of the reac-
tion signatures when merging reaction nodes to take into
account the local graph topology. This could be achieved
taking inspiration from the method proposed by Xu
et al. [38] in which the maximum entropy principle and
the Markov chain model-reduction problem were applied.
Finally, it should be highlighted that our method can be
easily adapted to other types of reaction classifications
based on chemical transformations.
Although its construction is based on an initial reac-
tion network, the RMS network offers new insights
into metabolism as it could capture relevant metabolic
contexts even without precise definition of initial reaction
sets or metabolite structures. Indeed, more than two
thousand reactions lacking a metabolic pathway were
integrated in the RMS network and now share com-
mon contexts with reactions from known pathways. Fur-
thermore, considering that many orphan enzymes have
network neighbours that are orphans themselves [2],
computational tools [39, 40] have difficulties to find
candidate genes for these missing enzymes by defining
correct genomic contexts (e.g. chromosomal clusters, co-
occurrence profiles) that include candidate proteins and
known enzymes. As a perspective, one of the possible
improvements of these methods could be the use of a RMS
network instead of a reaction network as it may be easier
to find proper genomic contexts using relaxed notions of
metabolic context. This enhancement may also be applied
in the discovery of gene clusters corresponding to new
metabolic pathways.
Additional files
Additional file 1: Reaction molecular signature classification of
reactions. (XLSX 410 kb)
Additional file 2: Comparison of RMS and enzyme commission
reaction partitions. (PDF 414 kb)
Additional file 3: List of conserved chemical transformation modules.
They correspond to RMS paths present in known metabolic pathways with
a PCI (Pathway Conservation Index) ≥2. (XLSX 76 kb)
Additional file 4: Boxplots of conservation scores for enumerated
and known metabolic paths of length 2 in the RMS-H1 network.
(PDF 306 kb)
Additional file 5: Statistical analysis of path score distributions
according to their pathway type. Kruskal-Wallis and Tukey HSD statistical
test results comparing scoreRea, scoreProt and scorePageRank distributions
for paths in RMS-H1 and H2 networks belonging to at least one known
metabolic pathway and depending on their pathway type. (PDF 317 kb)
Additional file 6: Metabolic pathway type prediction rules generated
by NNge algorithm. NNge model and cross-validation results for pathway
type prediction rules. (PDF 374 kb)](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-149-2048.jpg)

![122
Conclusion du Chapitre II
Une nouvelle représentation du métabolisme a été présentée dans cet article. Ce modèle de
données basé sur un réseau métabolique, où les nœuds sont des types de transformations
chimiques, est particulièrement utile pour retrouver des modules conservés. Ces modules de
transformations chimiques peuvent aider les biologistes dans la recherche de nouvelles voies
métaboliques similaires ou non à des voies métaboliques connues.
En considérant que beaucoup d’activités orphelines de séquences ont leurs voisins métaboliques
qui sont aussi orphelins [8], des outils comme CanOE [225] ont des difficultés pour trouver des
gènes candidats pour ces activités en définissant des contextes génomiques corrects qui incluent
des enzymes connues et des protéine candidates. La suite du travail de cette thèse était donc
l’utilisation du réseau de RMS, au lieu d’un réseau de réactions, pour faciliter la recherche de
contextes génomiques appropriés. Ce type d’approche peut aussi être appliqué pour la découverte
de groupes de gènes correspondants à de nouvelles voies métaboliques. C’est ce type d’approches
qui est présenté dans le chapitre suivant.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-151-2048.jpg)

![124
Chapitre III
Association de contextes
génomiques avec des modules
conservés de transformations
chimiques
Dans un grand nombre de cas, et particulièrement dans les organismes procaryotes, les gènes co-
localisés sur les chromosomes (dans des structures opéroniques notamment) sont souvent
impliqués dans une même fonction cellulaire. Dans un premier temps, une méthode simple de
prédiction de blocs de gènes proches sur les chromosomes (directons) a été développée et utilisée
sur l’ensemble de génomes disponibles au sein de la plateforme MicroScope [169].
Les directons, ainsi prédits, ont ensuite été placés dans un contexte métabolique représenté sous
la forme d’un réseau de signatures moléculaires de réactions (RMS). Pour cela, a été utilisée
l’association Pfam-RMS présentée dans le chapitre II de cette thèse, ce qui a permis d’associer les
gènes des directons contenant au moins un Pfam à des RMS du réseau. Ces associations
représentent des transformations chimiques potentielles que peuvent catalyser les protéines
codées par les gènes de l’opéron. Des sous-graphes formés des RMS ainsi sélectionnées sont
ensuite extraits, et leur nœuds colorés en fonction des gènes associés. Les chemins ayant un
maximum de couleurs (dans lesquels le plus grand nombre des gènes du directon sont impliqués)
et les meilleurs scores de conservation sont sélectionnés comme candidats pour l’annotation du
directon.
La troisième partie de ce chapitre est consacrée à une étude de cas. Il s’agit de replacer dans un
contexte génomique et métabolique une famille d’enzymes, les Baeyer-Villiger monooxygénases.
Ce sont des enzymes capables d’insérer un atome d’oxygène dans une liaison carbone-carbone,
transformation chimique très utile en chimie organique et ayant des applications industrielles](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-153-2048.jpg)

![126
I. Prédiction des directons dans les génomes
bactériens
Un opéron est une unité d’ADN fonctionnelle regroupant des gènes qui opèrent sous le signal
d’un même promoteur. Ces gènes sont co-transcrits et traduits à partir d’un ARN messager
polycistronique et concourent souvent à la réalisation d’une même fonction cellulaire. Les
opérons sont principalement connus chez les bactéries et les archées.
Le terme de directon réfère à un ensemble maximal de gènes adjacents localisés sur le même brin
d’ADN. Les directons sont relativement faciles à calculer et sont souvent de bons candidats pour
la prédiction d’opérons. Nous avons écrit une méthode de prédiction de directons adaptée à
l’analyse de génomes présents dans la plateforme MicroScope. Cette méthode sélectionne des
groupes de CDS (CoDing Sequences) sur le même brin suivant plusieurs critères (Figure 26):
- les CDS sont prédites par deux méthodes différentes (AMIGene [260] et Prodigal [261]) ;
- il y a maximum 100 nucléotides entre deux CDS
- il n’y a aucune CDS prédite simultanément par les deux méthodes sur le brin opposé
Figure 26. Critères de définition d’un directon : le nombre maximal de nucléotides entre deux CDS est de 100 ; les CDS
chevauchants sont pris en compte ; il ne doit pas y avoir de CDS sur le brin opposé de l’ADN.
Les CDS chevauchantes (distance négative entre deux CDS) sont considérées comme faisant
partie d’un seul directon. En effet, dans les organismes ayant une structure chromosomique](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-155-2048.jpg)
![127
compacte (comme les procaryotes et les virus), le chevauchement des gènes est très commun et
n’empêche pas leur transcription en ARNm polycistronique et leur traduction.
Les directons ont été prédits pour tous les génomes microbiens contenus dans la plateforme
MicroScope [169]. Des directons ont été prédits dans 5709 séquences génomiques, avec en
moyennes 644 directons par génome et 3,2 gènes par directon.
Le plus grand directon en nombre de gènes est de 52. Ce directon est retrouvé chez Kineococcus
radiotolerans, une bactérie polyextrémophile. Il pourrait ici s’agir d’un cas d’une surprédiction liée à
la nature de cette bactérie, car celle-ci présente un génome exceptionnellement compact avec des
puissants mécanismes de réparation de l’ADN qui participent à sa résistance à la radioactivité, la
dessiccation et à de nombreuses substances toxiques.
L’organisme qui a les directons les plus longs (8.75 gènes en moyenne) est Borrelia burgdorferi, une
bactérie ayant comme vecteur les tiques et responsable de la maladie de Lyme chez l’homme
[262]. Cette bactérie possède, en effet, beaucoup de grands opérons (allant jusqu’à 25 gènes) qui
sont impliqués, principalement, dans la motilité, la chémotaxie (mouvements en réponse à un
stimulus chimique) et l’infection.
Cette méthode, très simple, a été validée en comparant les directons prédits avec les opérons de la
base de données de RegulonDB qui sert de référence pour Escherichia coli K-12 MG1655 [178].
Dans RegulonDB les gènes sont partitionnés en 811 opérons, alors que notre méthode a détecté
973 directons. Globalement, nos prédictions sont assez cohérentes, notre méthode ayant
tendance à prédire des directons plus longs que les opérons dans RegulonDB. Cette comparaison
a été réalisée en étudiant l’appartenance simultanée ou non à un directon puis à un opéron des
gènes de toutes les paires de gènes possibles du génome. Ceci a permis de calculer trois
métriques :
- l’indice de Rand, qui est le rapport entre toutes les paires en accord (qui sont ensemble
dans un même directon d’une part et dans un même opéron d’autre part ou, qui sont
dans les deux cas dans des groupes différents) et toutes les paires possibles. Il s’agit d’une
mesure de comparaison de partitions, considérant qu’ici les gènes sont partitionnés en
opérons ou en directons. L’indice de Rand est un nombre entre 0 et 1, 0 étant pour deux
partitions complètement différentes, et 1 pour deux partitions identiques.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-156-2048.jpg)
![128
- la sensibilité : le rapport entre le nombre de paires où les deux gènes sont dans le même
opéron et le même directon et le nombre de toutes les paires dans un même opéron
- la spécificité : le rapport entre le nombre de paires où les deux gènes sont dans le même
opéron et le même directon et le nombre de toutes les paires dans un même directon
Dans la comparaison des partitions des gènes en directons par notre méthode et en opérons dans
la base de données RegulonDB, l’indice de Rand est de 0.9988, ce qui signifie que les deux
partitions sont très proches. Il faut cependant nuancer ce chiffre très haut, car le nombre total de
gènes à partitionner est assez élevé, et le nombre de paires en accord négatif (dans des groupes
différents dans les deux partitions) est d’autant plus grand, ce qui biaise ce calcul. Les mesures de
sensibilité et de spécificité permettent de nuancer cet index, car ne tiennent pas compte de toutes
les paires en accord négatif. La sensibilité de similitude entre les directons et les opérons est de
0.86 et la spécificité de 0.73. Ces chiffres, bien qu’assez élevés, ce qui démontre bien la similarité
des prédictions, reflètent aussi la légère différence du nombre et de taille des directons et des
opérons.
Des comparaisons similaires ont été réalisées en comparant les directons prédits chez E. coli K-12
et Acinetobacter baylyi ADP1 avec les prédictions des méthodes DOOR [263] et ProOpDB [264].
Notre méthode permet de détecter des blocs génomiques comparables en taille et en nombre à
ceux des deux autres ressources. De plus, nous pouvons calculer les directons rapidement sur
tous les génomes à notre disposition dans MicroScope. Il a donc été décidé d’utiliser les directons
prédits de cette façon pour les analyses combinant le contexte génomique au contexte
métabolique représenté, pour sa part, par les réseaux de signatures moléculaires de réactions.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-157-2048.jpg)
![129
II. Projection des directons sur le réseau de
signatures moléculaires de réactions
Des métriques d’association entre les familles de protéines Pfam, correspondant à des domaines
de protéines, et les RMS ont été établies selon la méthode décrite dans le chapitre II de cette
thèse. Il s’agit notamment d’un score de sélectivité (équivalent à un F-score) basé sur un calcul de
la sensibilité et de la spécificité d’association, qui représentent la fraction de protéines associées, à
la fois, à un domaine Pfam donné et à une RMS donnée. Le nombre total de protéines associées
constitue également une métrique intéressante pour donner une indication quantitative à ce score.
Ces métriques permettent ainsi d’évaluer la probabilité qu’une protéine soit impliquée dans la
catalyse de tel ou tel type de transformation chimique.
Pour chacun des gènes des directons prédits selon la méthode décrite dans la section précédente,
les domaines Pfam des protéines correspondantes ont été déterminés à l’aide du logiciel
InterproScan [145]. Des RMS ont ensuite été associées à ces gènes via les domaines Pfam
calculés. Une limite de cette méthode est de ne pas pouvoir associer de RMS à des gènes n’ayant
pas de résultat Pfam. De plus, certaines RMS (environ 35%) ne peuvent pas être associées à des
gènes car elles n’ont pas de protéines connues pour catalyser la transformation ou les protéines
connues n’ont pas de domaines Pfam.
Pour chaque directon, les associations gènes-RMS sont ensuite projetées sur le réseau de RMS.
Les nœuds, correspondant aux RMS présentes dans le directon, sont ainsi sélectionnés et
« coloriés » avec une couleur par gène. A partir de ces nœuds et de toutes les arêtes du réseau
initial, un sous-réseau est extrait. Les nœuds isolés sont supprimés et s’il existe plusieurs sous-
graphes connexes, ils sont considérés comme des entités distinctes.
Pour chaque sous-graphe, tous les chemins possibles sont énumérés, et ne sont sélectionnés que
les chemins passant par toutes les couleurs ou un maximum de couleurs – c’est à dire par des
RMS qui sont catalysées par le produits de tous (ou un maximum) de gènes du directon. Ce
processus de projection de directons sur le réseau de RMS est décrit en Figure 27.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-158-2048.jpg)
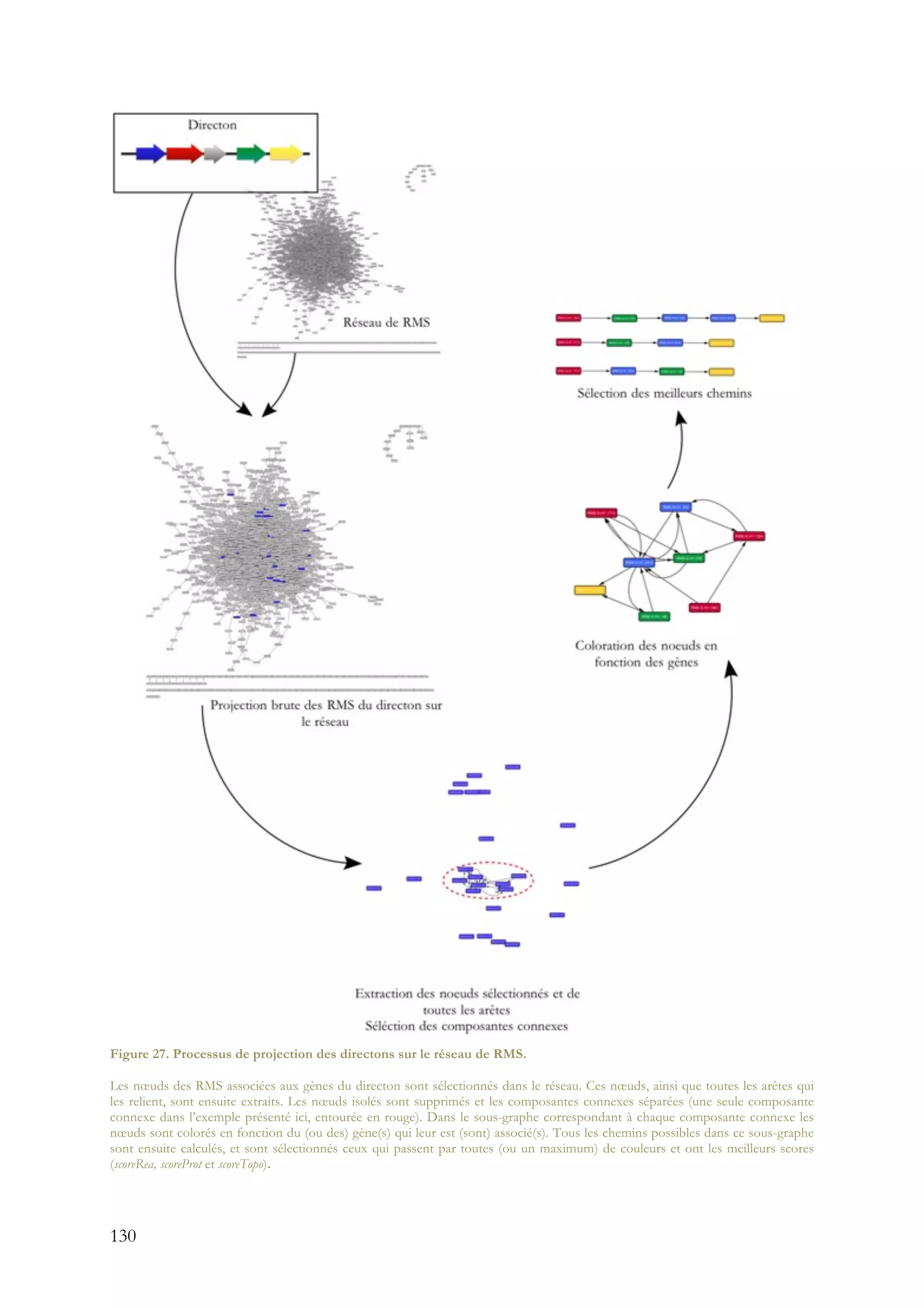
![131
Vu que la taille de ces sous-réseaux est relativement faible (une dizaine de nœuds en général), il
était plus simple, d’un point de vue computationnel, d’énumérer tous les chemins possibles et
ensuite calculer le nombre de couleurs représentées dans les chemins que d’utiliser des
algorithmes complexes de recherche de chemins colorés optimaux (ce qui peut aussi être assimilé
à la recherche de motifs, comme le fait le programme MOTUS [246], par exemple).
Un certain nombre de chemins de transformations chimiques candidats pour les directons est
ainsi obtenu. La sélection des meilleurs chemins repose ensuite sur la comparaison de leurs scores
(scoreRea, scoreProt et scoreTopo (aussi appellé scorePageRank dans l’article [30]), cf. chapitre II). Il n’est
pas forcément nécessaire que tous les scores d’un chemin donné soient plus élevés que ceux des
autres chemins, ainsi, par exemple, un chemin avec un scoreTopo ou un scoreRea particulièrement
élevé sera préféré à un chemin où les trois scores sont plutôt moyens. En effet, on préfèrera un
chemin très conservé selon un seul critère (conservation chimique, enzymatique ou topologique)
à un chemin moyennement conservé pour l’ensemble des score. Il faut aussi remarquer que,
parmi les chemins candidats, le scoreProt sera toujours non nul alors qu’il l’est pour environ 30%
des chemins dans le réseau global de RMS. Ceci vient du fait que les gaps (i.e. RMS non associées
à un gène du directon) ne sont pas autorisés dans l’extraction des sous-graphes lors de la
projection du directon sur le réseau. Ainsi, toutes les RMS des chemins sélectionnés sont
associées à au moins une famille Pfam et à au moins un gène du directon.
Pour la prise en compte des RMS sans famille Pfam associée, ce qui est incontestablement
intéressant pour l’annotation de protéines à fonction inconnue ou non-associées à une famille
Pfam, un paramètre de gap à 1 permettrait d’intégrer les voisins directs des nœuds RMS
sélectionnées lors de la recherche de sous-graphes. Néanmoins, les réseaux de RMS,
indépendamment de la hauteur des signatures de réaction, sont des graphes assez compacts où le
nombre moyen de voisins d’un nœud (i.e. le degré) est de 6,4. L’inclusion de gaps rend donc la
taille des sous-graphes extraits assez importante. La sélection des chemins candidats est alors
beaucoup plus compliquée et requiert, cette fois-ci, des stratégies d’exploration plus performantes
qui n’ont pas été développées au cours de cette thèse mais qu’il serait intéressant d’élaborer par la
suite.
De cette façon, pour chaque directon est obtenu un certain nombre de chemins candidats
associés à des scores. La sélection du chemin le plus plausible, dans le cas où plusieurs chemins
différents ont des scores élevés, nécessite pour l’instant l’intervention d’un expert ayant la](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-160-2048.jpg)

![133
III. Etude de cas : identification de contextes
génomiques et métaboliques pour les enzymes
Baeyer-Villiger Monooxygénases
L’oxydation de type Baeyer-Villiger (BV) est une transformation chimique transformant des
cétones linéaires ou cycliques en esters ou lactones correspondants en introduisant un atome
d’oxygène dans un lien carbone-carbone [265]. Cette réaction peut être réalisée par des enzymes
appelées Bayer-Villiger Monooxygénases (BVMOs). Ce sont des flavoenzymes, c’est à dire des
oxydoréductases qui nécessitent un dinucléotide flavine-adénine (FAD) comme groupement
prosthétique pour fonctionner. Elles sont capables de catalyser des réactions d’oxydation sur des
cétones linéaires, cycliques et aromatiques. Pendant la réaction d’oxydation, un atome d’oxygène
est incorporé entre deux carbones connectés, alors que l’autre atome d’oxygène est capturé dans
une molécule d’eau avec les atomes d’hydrogène provenant du cofacteur NAD(P)H. Les BVMOs
sont des protéines solubles dans un milieu aqueux et ne nécessitent pas d’autres protéines pour
fonctionner. Il existe au moins deux classes de BVMOs : les BVMOs de type I qui sont
constituées d’une seule chaine polypeptidique et sont dépendantes de FAD et de NADPH pour
catalyser leur activité, et les BVMOs de type II, très peu étudiées, composées de deux sous-unités
différentes et utilisant le FMN comme cofacteur flavinique et le NADH comme donneur
d’électron. Dans cette étude de cas, seules les BVMOs de type I sont analysées. Dans la figure
Figure 28 est représentée la structure générale d’une BVMO de type I (code Protein Data Bank
3GWD) avec les deux cofacteurs montrés avec la représentation en bâtons.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-162-2048.jpg)

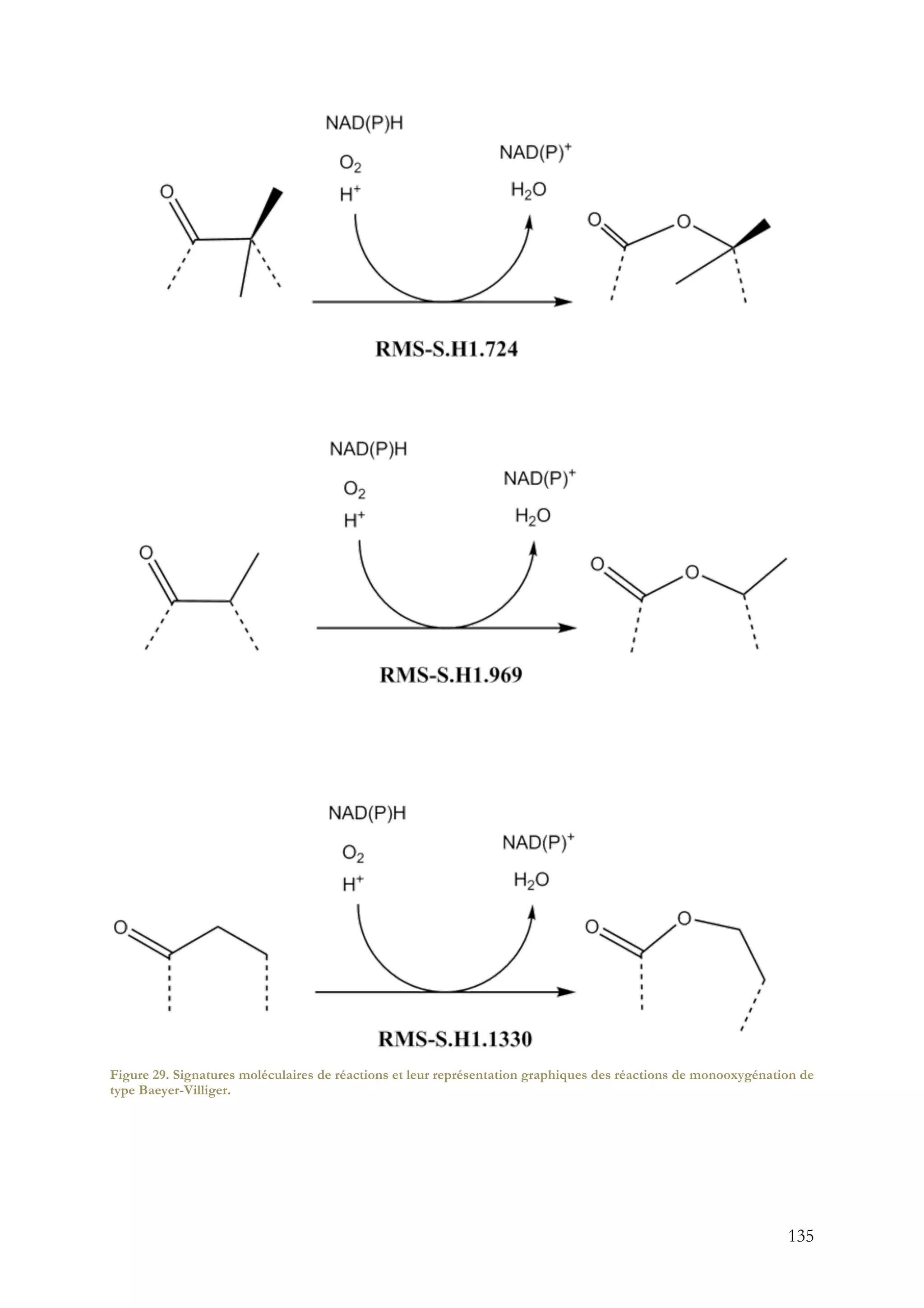
![136
La sous-sous-classe des EC numbers correspondant à ces réactions est EC 1.14.13. Cependant,
deux des réactions n’ont aucun EC number associé et six sont annotées avec un EC number
partiel. Les autres réactions sont associées à sept EC numbers différents, dont dix sont associées
à EC 1.14.13.105. Cependant, pour les réactions annotées avec un EC number complet, cette
annotation diverge à certains moments avec la classification par RMS, basée sur la transformation
chimique opérée par chaque réaction. Ces divergences de classification sont présentées dans la
Table 2. Très peu de protéines sont disponibles dans MetaCyc pour ces réactions.
Table 2. Comparaison de la classification EC et RMS pour les réactions de type Baeyer-Villiger issues de MetaCyc. Les
identifiants UniProt sont indiqués lorsqu’il y a une protéine connue associée à la réaction. Un décalage est observé entre les deux
classifications.
Identifiant de réaction
MetaCyc
EC Number RMS Identifiants UniProt
CYCLOHEXANONE-
MONOOXYGENASE-RXN
1.14.13.22 RMS-S.H1.1330 Q9R2F5
CYCLOPENTANONE-
MONOOXYGENASE-RXN
1.14.13.16 RMS-S.H1.1330
RXN-11537 1.14.13 RMS-S.H1.1330 Q940V4
RXN-11538 1.14.13 RMS-S.H1.1330 Q940V4
RXN-12654 1.14.13.170 RMS-S.H1.1330 E3VWK3
RXN-720 1.14.13 RMS-S.H1.1330 Q50LE0,Q940V4
RXN-9395 1.14.13.105 RMS-S.H1.1330
RXN-9396 1.14.13.105 RMS-S.H1.1330
RXN-9431 1.14.13.105 RMS-S.H1.1330
RXN-9435 1.14.13.105 RMS-S.H1.1330
RXN-9487 NULL RMS-S.H1.1330 Q6UEF3
RXN-9492 NULL RMS-S.H1.1330 Q6UEF3
R543-RXN 1.14.13.162 RMS-S.H1.724
RXN-12713 1.14.13.54 RMS-S.H1.724
RXN-13043 1.14.13 RMS-S.H1.724
1.14.13.54-RXN 1.14.13.54 RMS-S.H1.969
R422-RXN 1.14.13 RMS-S.H1.969
R423-RXN 1.14.13 RMS-S.H1.969
RXN-12661 1.14.13.171 RMS-S.H1.969 Q82IY8
RXN-7817 1.14.13.54 RMS-S.H1.969
RXN-9390 1.14.13.105 RMS-S.H1.969
RXN-9391 1.14.13.105 RMS-S.H1.969
RXN-9420 1.14.13.105 RMS-S.H1.969
RXN-9440 1.14.13.105 RMS-S.H1.969
RXN-9441 1.14.13.105 RMS-S.H1.969
RXN-9442 1.14.13.105 RMS-S.H1.969
III.2 Identification des contextes génomiques des BVMOs
Afin d’identifier le contexte génomique des BVMOs dans les génomes à notre disposition dans la
plateforme MicroScope [169], il faut tout d’abord y repérer les gènes codant ces enzymes. Deux
motifs complémentaires d’acides aminés ont été utilisés pour détecter les BVMOs : le motif](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-165-2048.jpg)
![137
« FxGxxxHxxxW » – spécifique des monooxygénases en général et le motif « GxWxxNxYPG »
– spécifique des BVMOs [265]. Un motif indique la nature et la position relative des acides
aminés importants dans la séquence d’une protéine pour le maintien d’une fonction. Par exemple,
dans le motif spécifique des BVMOs, à un endroit de la séquence, il doit nécessairement y avoir
une glycine, suivie par n’importe quel acide aminé, puis un tryptophane, puis deux acides aminés
quelconques, une asparagine, encore n’importe quel acide aminé, puis une tyrosine suivie d’une
proline et d’une glycine. La présence de ces deux motifs dans une séquence protéique est donc
nécessaire pour considérer la protéine comme étant une BVMO.
Nous avons donc recherché, parmi tous les génomes microbiens disponibles au sein de la
plateforme MicroScope, des CDS qui codent des protéines ayant ces deux motifs à l’aide du
programme ps_scan (PROSITE scanning program). 1234 protéines ont ainsi pu être récupérées,
dans 506 génomes différents. Il y a donc entre deux et trois BVMOs en moyenne dans les
organismes possédant ce type d’activité enzymatique.
Puisque c’est le contexte génomique des BVMOs qui nous intéresse dans cette étude, seules les
BVMOs présentes dans un directon sont gardées. Parmi les 1234 BVMOs prédites, 969 sont dans
un des 814 directons appartenant à 468 génomes. Ces directons permettent ainsi de définir
plusieurs contextes génomiques pour les BVMOs qui serviront à ancrer, par la suite, des
contextes métaboliques.
Figure 30. Dendrogramme présentant le résultat du clustering hiérarchique des directons en fonction de leur contenu
en RMS. Rouge - cluster 1, violet - cluster 2, jaune - cluster 3, vert – cluster 4 et bleu – cluster 5.
En suivant la méthode présentée dans le deuxième chapitre de ce manuscrit et rappelée en début
de ce chapitre, les protéines des directons contenant au moins une BVMO ont été associées à des
RMS en utilisant leur contenu en domaines Pfam [144]. Afin d’identifier les différences et les](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-166-2048.jpg)

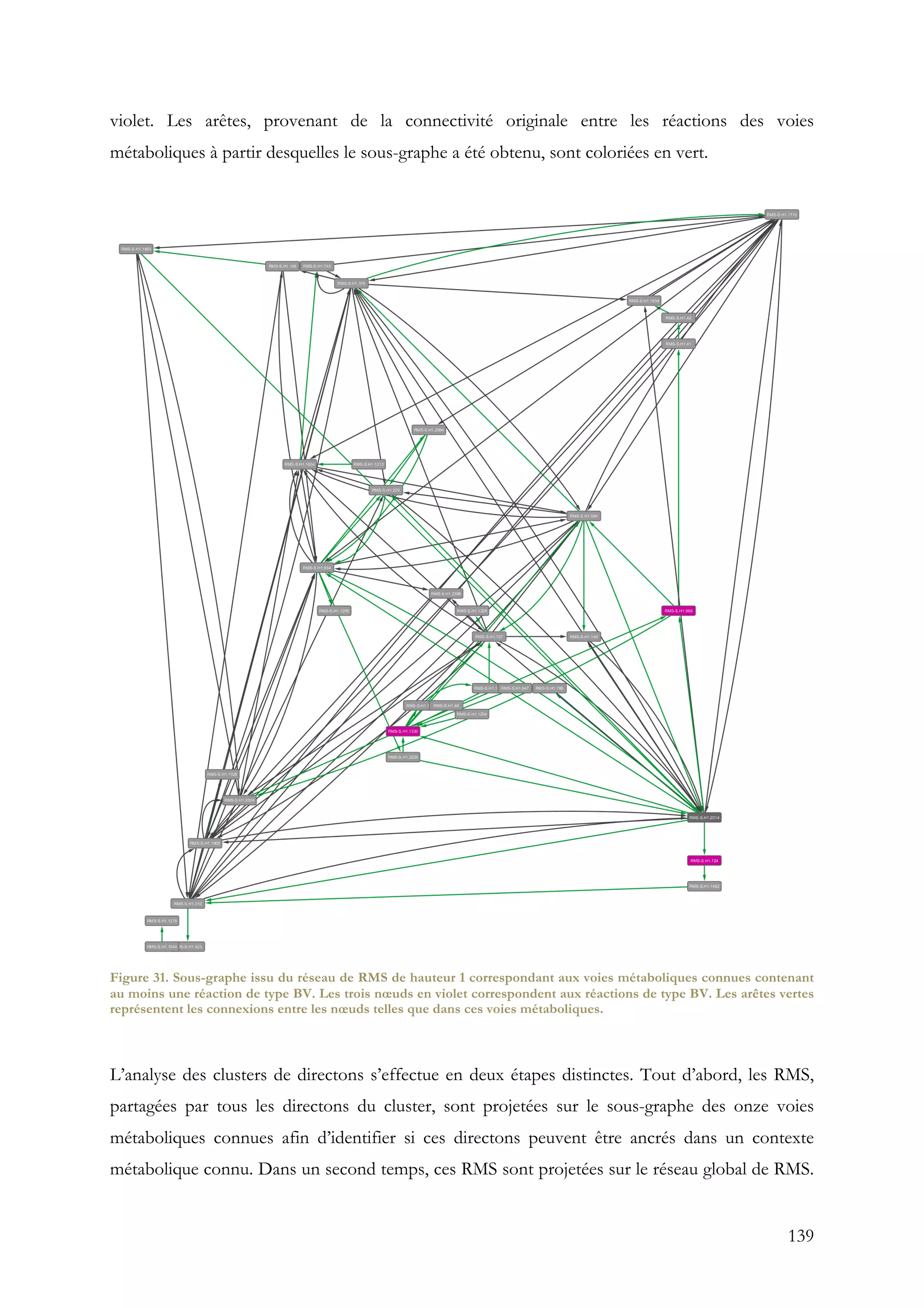

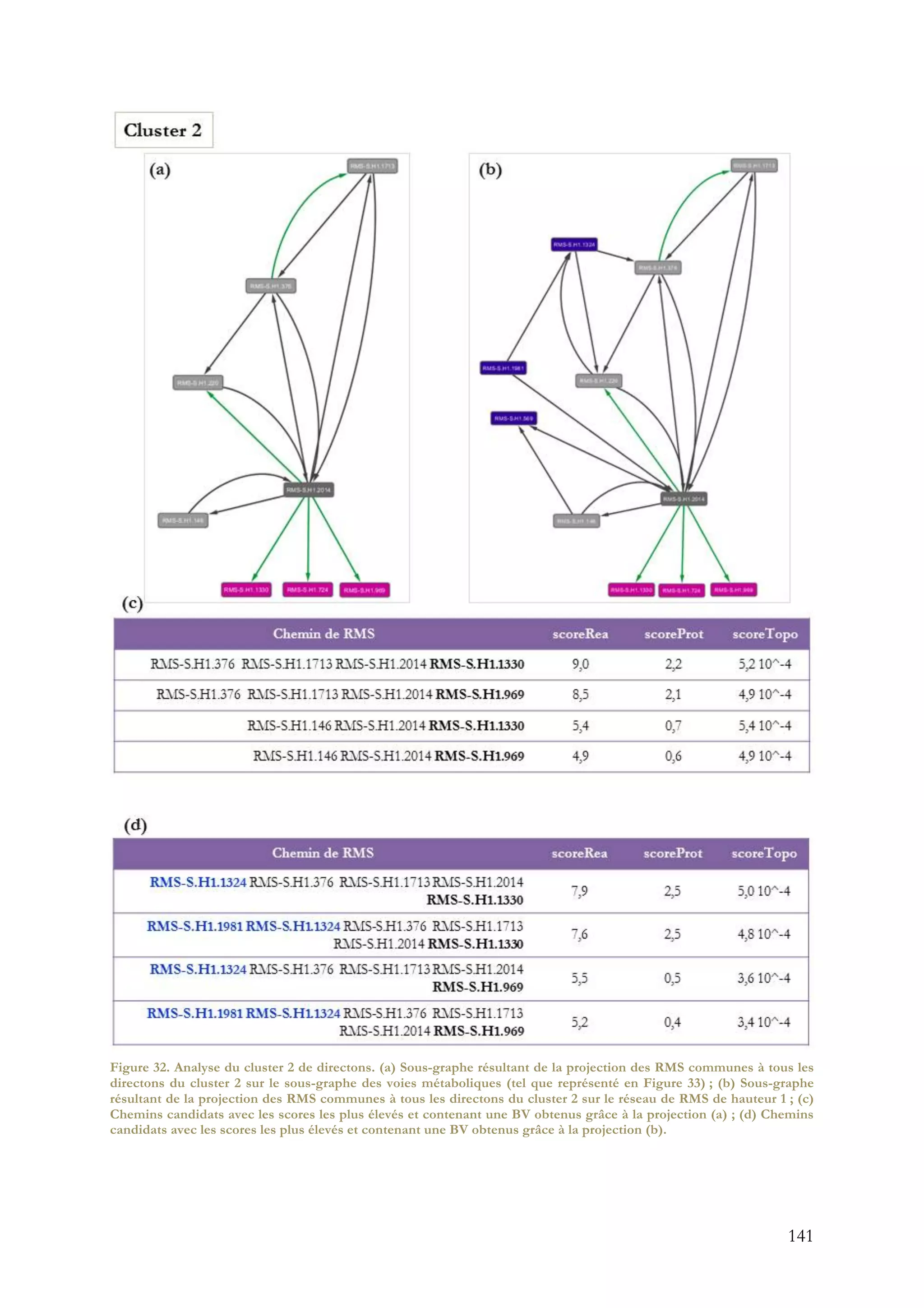
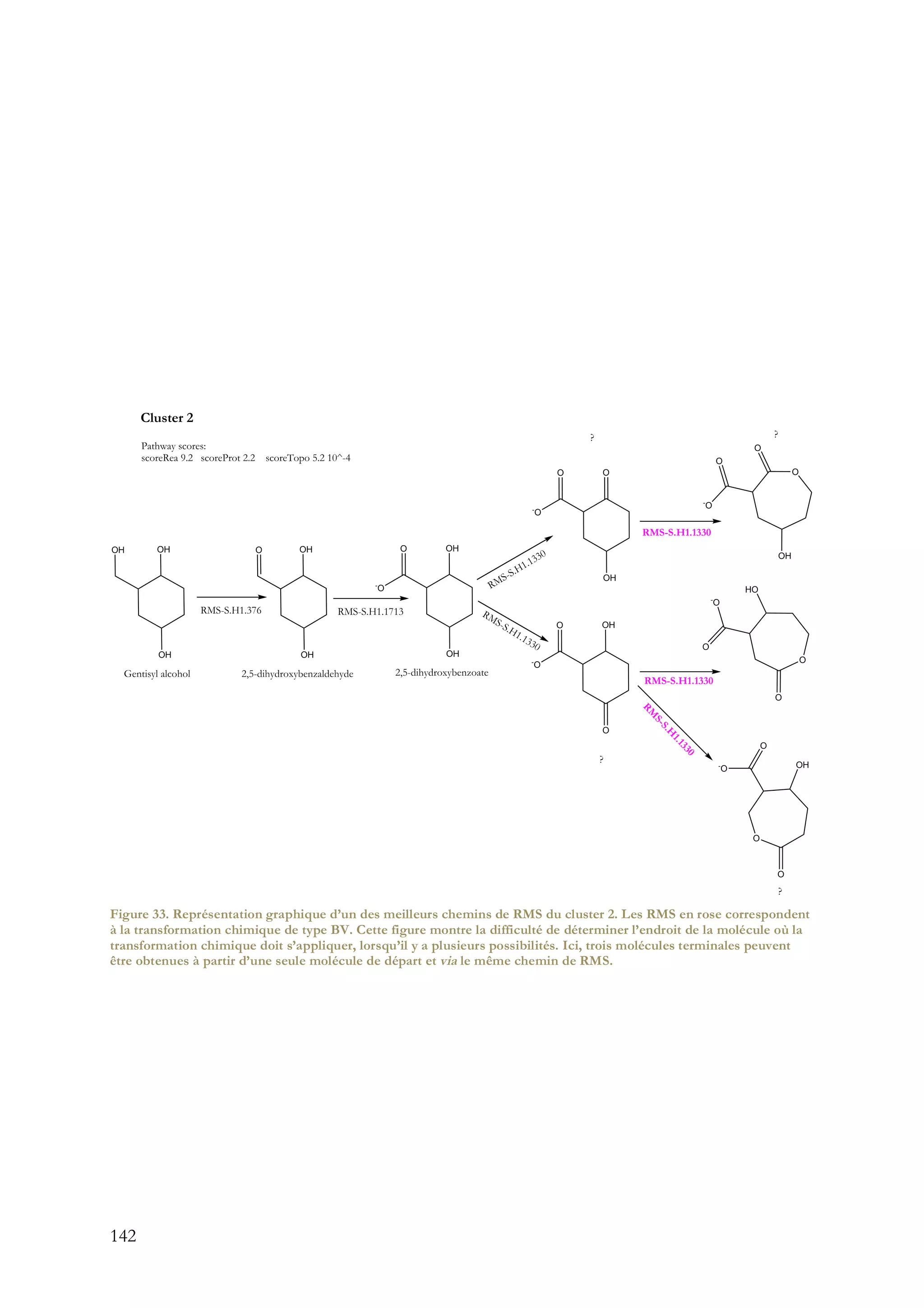

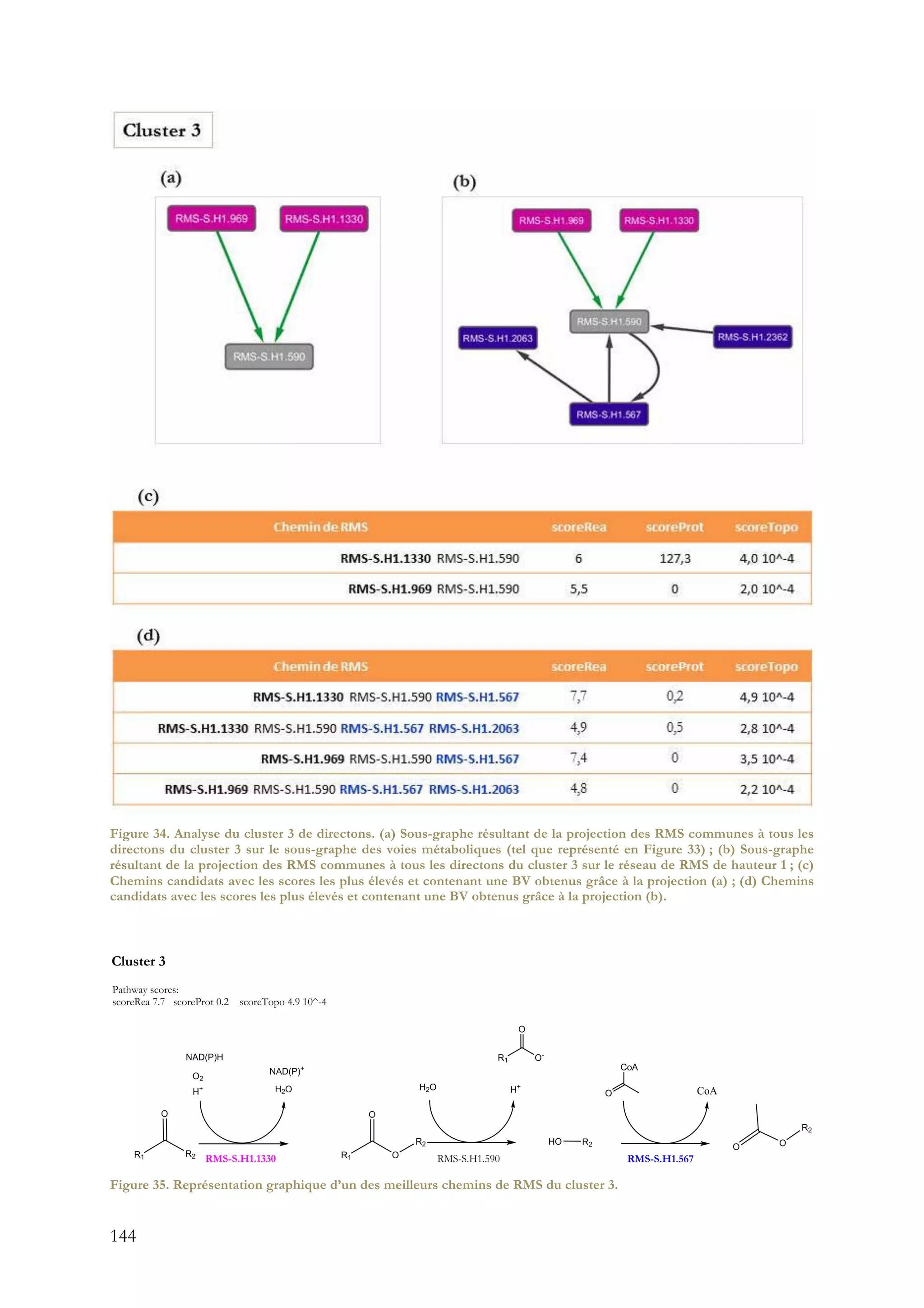

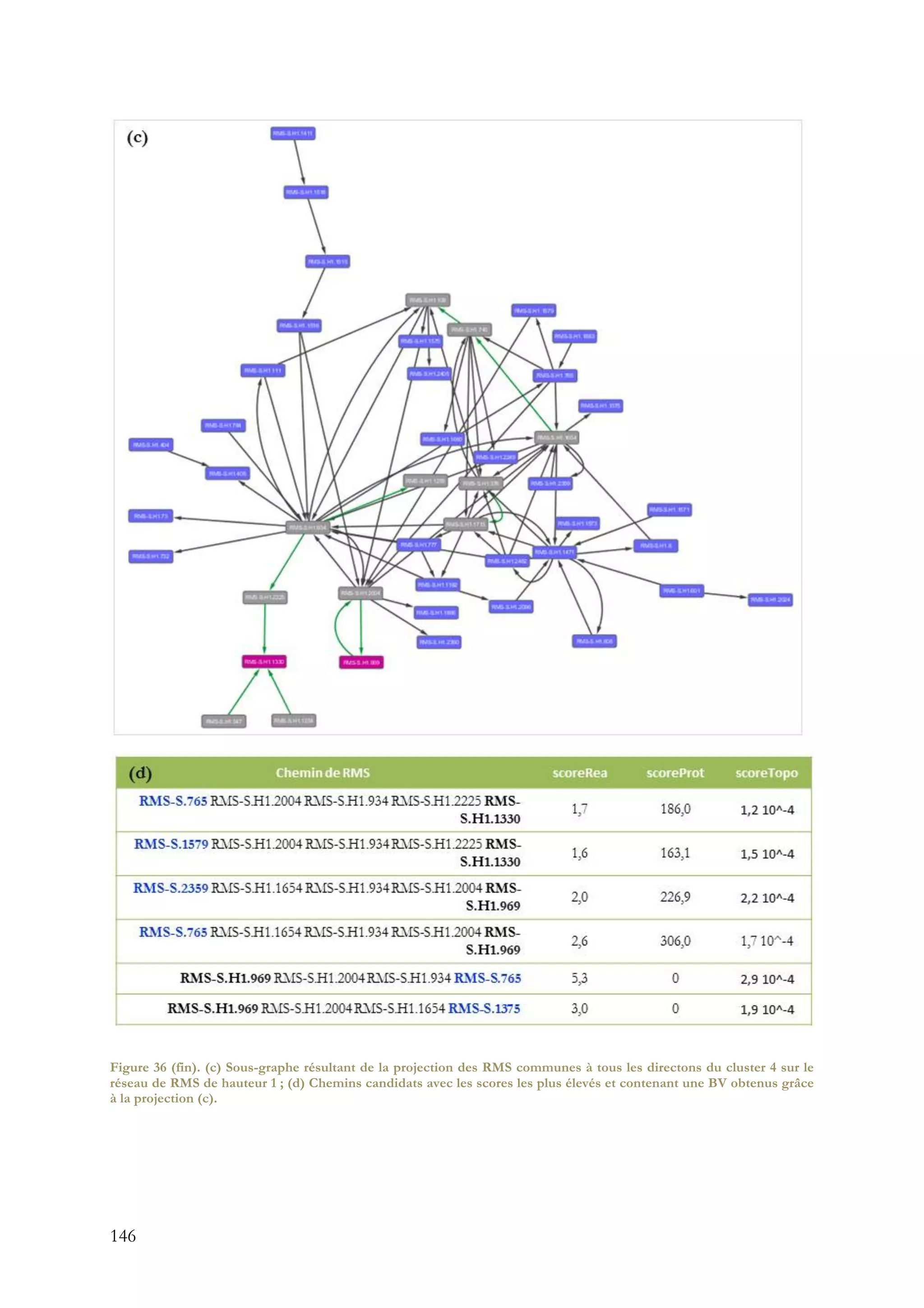
![147
Cluster 5
Dans le cluster 5, le plus petit des clusters avec seulement 59 directons, cinq RMS sont retrouvées
dans chaque directon. Il n’y a cependant aucune intersection entre ces cinq RMS et les RMS
présentes dans les voies métaboliques connues. La projection de ces RMS sur le réseau global de
RMS de hauteur 1 n’a pas non plus permis d’établir de liens avec les éléments conservés de ces
directons et les BVMOs. Les BVMOs putatives des directons de ce cluster n’ont donc pas pu être
mises dans un contexte métabolique.
L’approche présentée ici permet de remettre les BVMOs à la fois dans un contexte génomique et
dans un contexte métabolique. L’association des projections sur le réseau de voies métaboliques
connues puis sur le réseau de RMS global permet dans un premier temps d’ancrer les BVMOs
dans un contexte métabolique connu pour ensuite l’étendre dans un deuxième temps.
On a ainsi pu placer dans un contexte métabolique plus de 60% des BVMOs dont le contexte
génomique avait été précédemment identifié. La poursuite de cette étude nécessite une expertise
humaine et des expérimentations pour valider les chemins métaboliques prédits. Un criblage
enzymatique à haut débit des BVMOs permettrait d’identifier des métabolites candidats et d’aider
à choisir les chemins optimaux de transformations chimiques que les enzymes des directons, dans
lesquelles se trouvent les BVMOs, sont capables de catalyser.
Une des améliorations possibles, pouvant être apportées à cette étude de cas, est d’affiner le
clustering des directons, notamment en découpant le cluster 1 en 2 clusters, afin d’identifier des
RMS communes à tous les directons et identifier un contexte métabolique pour eux aussi.
L’association des protéines aux RMS, qui, pour l’instant, est effectuée au travers de la
composition des protéines en domaines Pfam [144] devra aussi être améliorée. En effet,
l’association Pfam-RMS est dans certains cas peu fiable, car, tout d’abord, certains domaines
Pfam ne sont pas directement liés à la fonction enzymatique, de plus, un type de réactions peut
être lié à beaucoup d’entrées Pfam, ou, inversement, un domaine Pfam peut être associé à
beaucoup de réactions dont les transformations (RMS) sont différentes. Ce double problème
provient principalement de la généricité de certaines familles Pfam. Une méthode alternative de
prédiction de RMS pour les protéines sera proposée dans les perspectives de ce travail.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-176-2048.jpg)



![151
Dans la troisième partie de ce manuscrit, a été présenté un exemple d’utilisation du réseau de
RMS pour la définition d’un contexte métabolique pour une famille d’enzymes.
Dans un premier temps, une méthode simple de prédiction de directons (opérons potentiels) a
été développée et utilisée sur l’ensemble des génomes disponibles au sein de la plateforme
MicroScopee [169] qui est développée au sein du laboratoire où la thèse présentée ici s’est
déroulée. Ensuite, un processus de projection de ces directons sur le réseau de RMS a été établi
afin de placer les gènes qui les constituent dans un contexte métabolique cohérent, et de
déterminer si un module conservé de transformations chimiques peut être réalisé par un directon
donné.
Ces deux méthodes ont ensuite été utilisées pour une étude de cas. Les enzymes de la famille des
Baeyer-Villiger monooxygénases (BVMOs) ont été placées dans un contexte génomique en
repérant tous les directon contenant un gène codant une BVMOs, repéré par la présence de deux
motifs de séquence spécifiques. Ces directons contenant une BVMOs ont été classifiés en cinq
groupes distincts en fonction de leur contenu en RMS. Deux de ces cinq groupes n’ont pas pu
être placés dans le réseau de RMS d’une façon cohérente, mais les trois autres ont été assignées à
un contexte métabolique. Dans les trois cas, le contexte métabolique était différent et un ou
plusieurs chemins de RMS (modules) avec des scores élevés de conservation ont été proposés.
Ces modules candidats devront par la suite être analysés par des experts en biochimie et,
éventuellement, testés en laboratoire.
La combinaison des méthodes de contexte génomique au réseau de RMS développé au cours de
cette thèse peut avoir des applications intéressantes pour l’annotation fonctionnelle des enzymes
ainsi que pour la découverte de nouvelles voies métaboliques. Les perspectives envisagées pour la
suite de ce travail de thèse sont décrites dans la section suivante.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-180-2048.jpg)
![152
Perspectives
La représentation du métabolisme sous la forme d’un réseau de transformations chimiques
encodées en signatures moléculaires de réactions (RMS) ouvre un grand nombre de perspectives
dans l’étude de celui-ci. Un certain nombre d’entre elles sont présentées dans cette partie.
Cette représentation peut être utile pour l’assignation de séquences pour les enzymes orphelines.
En effet, beaucoup d’outils développés pour résoudre ce problème se basent sur le contexte
métabolique et génomique de ces activités [226, 266], or, beaucoup d’entre elles ont leurs voisines
qui sont aussi orphelines de séquences [8]. Le réseau de RMS permet ainsi de définir un contexte
métabolique plus relâché facilitant son ancrage sur des contextes génomiques pouvant contenir
des gènes candidats pour plusieurs réactions orphelines.
Les RMS regroupent souvent plusieurs réactions, dont certaines sont orphelines. En explorant
une famille d’enzymes connues pour catalyser des réactions décrites par une RMS, des protéines
de cette famille peuvent être proposées comme candidates pour les réactions orphelines de la
RMS. Cela suppose que la famille possède une certaine promiscuité de substrats qui peut, par
exemple, être évaluée par une analyse de la structure de ces protéines : comparaison des sites
actifs et des expériences d'amarrage (docking) moléculaire.
Nous avons soulevé le problème de RMS orphelines dans le deuxième chapitre de cette thèse. En
effet, plus de 35% des RMS n’ont aucune séquence protéique qui a pu leur être associée, ce qui
signifie qu’aucune des réactions qu’elles rassemblent n’est catalysée par une enzyme connue. Il est
donc important de prioriser la recherche de candidats pour les transformations chimiques
orphelines, notamment avec des méthodes existant déjà pour les enzymes orphelines [226, 266]
ou en en développant des nouvelles, adaptées à la représentation du métabolisme avec des RMS.
Comme il a été souligné dans l’article de revue sur les enzymes orphelines, une partie d’entre elles
sont considérées comme orphelines à cause du retard entre les bases de données et la littérature.
Afin de limiter ce retard de connaissances, il est nécessaire de mettre en place un standard
international permettant de déposer des enzymes et des activités caractérisées expérimentalement
en même temps que les publications qui y sont liées, comme c’est le cas pour la soumission des
séquences nucléiques dans les bases de données comme GenBank [267] et l’European Nucleotide
Archive [268]) en même temps que leur publication dans les journaux.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-181-2048.jpg)
![153
Il est aussi envisageable d’étendre le concept des activités orphelines aux métabolites orphelins,
qui sont des métabolites identifiés dans un organisme, mais dont on ne connaît pas les enzymes
qui permettent leur synthèse ni leur dégradation. En effet, les avancées en métabolomique, par
spectrométrie de masse ou résonance magnétique nucléaire, permettent de découvrir un grand
nombre de nouveaux métabolites. Dans ce cas, il s’agirait de trouver des chemins de RMS
permettant de relier ces métabolites orphelins d'enzymes à des voies métaboliques nouvelles. Des
méthodes de reconstruction de novo de voies métaboliques et d’identification de nouvelles activités
enzymatiques à partir de données de métabolomique, comme celle de Kotera et al. [269] ou celle
de Prosser et al. [270] pourraient être adaptées à la représentation du métabolisme sous la forme
de chemins et de réseaux de RMS.
Les RMS sont un moyen efficace et automatique de classification des réactions en fonction du
type de transformation chimique qu’elles réalisent. Comme nous l’avons démontré dans le
chapitre II de cette thèse, cette classification est une bonne alternative à la classification EC. Il
serait donc intéressant pour la communauté scientifique de créer une base de données publique
de RMS et des réactions qu’elles décrivent, avec un accès via un serveur web.
La nouvelle façon de représenter et explorer le métabolisme, développée lors de cette thèse, est
une première brique dans l’exploitation de ce type de réseaux métaboliques. Un certain nombre
d’améliorations, notamment méthodologiques, et de perspectives sont envisagées pour la suite.
Tout d’abord, il est envisagé d’adapter dynamiquement la précision de la signature de réaction
lors de la fusion des nœuds de réactions afin de prendre en compte la topologie locale du graphe
et la taille du groupe de réactions. Ceci peut se faire notamment en s’inspirant de la méthode
proposée par Xu et al. [271] dans laquelle ont été appliqués le principe d’entropie maximale et le
problème de réduction de modèles de chaines de Markov.
Les modules conservés de transformations chimiques décrits dans cette thèse sont linéaires, c’est
à dire que chaque RMS du module est précédée et est suivie au maximum par une autre RMS, et
le module a une RMS initiale (qui n’est pas précédée par une autre RMS) et une RMS terminale
(qui n’est pas suivie par une autre RMS). Or, un certain nombre de voies métaboliques décrites
dans les bases de données présentent des structures topologiques plus complexes qu’un chemin.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-182-2048.jpg)

![155
prévu d’implémenter une stratégie permettant de définir des domaines pour les RMS en
s’inspirant de celle utilisée par PRIAM [143] pour les EC numbers qui est basée sur l’algorithme
de MKDOM [142]. Ce type d’approche permet d’identifier des segments communs à toutes les
séquences de protéines dans un groupe, dans le cas présent, toutes les séquences associées à une
même RMS. L’identification d’un (ou des) domaine(s) spécifique(s) à une RMS permettra une
meilleure prédiction de RMS pour les protéines, ce qui améliorera le potentiel de la méthode en
termes d’annotation fonctionnelle des gènes et des groupes de gènes comme les opérons.
La méthode de projection de gènes partageant un contexte génomique sous la forme d’un opéron
ou d’un directon présentée dans le chapitre III de cette thèse prévoit que les produits de ces
gènes catalysent des transformations chimiques directement voisines dans le réseau. Or, certains
gènes sans fonction prédite ou des gènes ne faisant pas parti du contexte génomique analysé
peuvent aussi intervenir dans la voie métabolique et posent donc problème car ils ne sont pas pris
en compte dans la méthode actuelle de projection. Un paramètre de « gap » devrait donc être
introduit dans la projection des groupes de gènes sur le réseau de RMS pour tenir compte de ces
éventualités. Pour faire cela, il faudrait prendre en compte les nœuds voisins des nœuds
sélectionnés par la projection. La taille des sous-graphes ainsi sélectionnés sera plus grande. Il
faudra donc envisager une amélioration méthodologique de recherche de chemins optimaux.
Une autre perspective, qui sera explorée dans le cadre de mon projet postdoctoral, est l’étude de
variations métaboliques interindividuelles grâce aux réseaux de RMS. En effet, les individus d’une
même espèce présentent, généralement, de légères variations au niveau de leur génotype. Ces
différences peuvent concerner des gènes impliqués dans des processus métaboliques. Ainsi,
l’étude de l’impact de variations interindividuelles sur un réseau métaboliques permettra une
meilleure compréhension de phénomènes biologiques comme la prédisposition de certains
individus aux maladies ainsi que leur vieillissement. Même si ces variations sont assez difficiles à
détecter, elles ne sont pas moins importantes à étudier, car elles mènent à la compréhension des
spécificités et des réponses à l’environnement de chaque individu. Dans ce cadre, l’utilisation de
réseaux de RMS peut s’avérer particulièrement utile à plusieurs niveaux. En effet, moins sensibles
aux « trous » dus à une absence d’annotation fonctionnelle de gènes que les réseaux de réactions
ou de métabolites, ils permettent en plus d’établir une tendance générale de présence/absence de
types de transformations chimiques dans l’individu, ainsi que d’étudier les différences de chemins](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-184-2048.jpg)









![165
information system for analyzing bioactivities of small molecules. Nucleic Acids Res
2009, 37(Web Server issue):W623–33.
114. Tipton K, Boyce S: History of the enzyme nomenclature system. Bioinformatics
2000, 16:34–40.
115. Bairoch A: The ENZYME data bank. Nucleic Acids Res 1994, 22:3626–3627.
116. Bastard K, Smith AAT, Vergne-Vaxelaire C, Perret A, Zaparucha A, De Melo-
Minardi R, Mariage A, Boutard M, Debard A, Lechaplais C, Pelle C, Pellouin V,
Perchat N, Petit J-L, Kreimeyer A, Medigue C, Weissenbach J, Artiguenave F, De
Berardinis V, Vallenet D, Salanoubat M: Revealing the hidden functional diversity
of an enzyme family. Nat Chem Biol 2014, 10:42–9.
117. Deville Y: An overview of data models for the analysis of biochemical
pathways. Brief Bioinform 2003, 4:246–259.
118. Orth JD, Thiele I, Palsson BØ: What is flux balance analysis? Nat Biotechnol
2010, 28:245–8.
119. Stelling J: Mathematical models in microbial systems biology. Curr Opin
Microbiol 2004, 7:513–8.
120. Przytycka TM, Andrews J: Systems-biology dissection of eukaryotic cell
growth. BMC Biol 2010, 8:62.
121. Larhlimi A, Blachon S, Selbig J, Nikoloski Z: Robustness of metabolic networks:
a review of existing definitions. Biosystems 2011, 106:1–8.
122. Wagner A, Fell DA: The small world inside large metabolic networks. Proc Biol
Sci 2001, 268:1803–10.
123. Arita M: The metabolic world of Escherichia coli is not small. Proc Natl Acad
Sci U S A 2004, 101:1543–7.
124. Caspi R, Altman T, Dreher K, Fulcher CA, Subhraveti P, Keseler IM, Kothari A,
Krummenacker M, Latendresse M, Mueller LA, Ong Q, Paley S, Pujar A, Shearer AG,
Travers M, Weerasinghe D, Zhang P, Karp PD: The MetaCyc database of metabolic
pathways and enzymes and the BioCyc collection of pathway/genome databases.
Nucleic Acids Res 2012, 40(Database issue):D742–53.
125. Karp P, Paley S: Representations of metabolic knowledge: pathways. Ismb
1994.
126. Karp P, Paley S: Automated drawing of metabolic pathways. Proc 3rd Int Conf
… 1994.
127. Katz L: On the Matric Analysis of Sociometric Data. Sociometry 1947, 10:233–
241.
128. Seeley JR: The net of reciprocal influence. Study II: The balance of power. .
129. Brandes U, Erlebach T (Eds): Network Analysis. Volume 3418. Berlin, Heidelberg:
Springer Berlin Heidelberg; 2005. [Lecture Notes in Computer Science]
130. Katz L: A new status index derived from sociometric analysis. Psychometrika
1953, 18:39–43.](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-194-2048.jpg)
![166
131. Bonacich P: Factoring and weighting approaches to status scores and clique
identification. J Math Sociol 1972, 2:113–120.
132. Hubbell CH: An Input-Output Approach to Clique Identification. Sociometry
1965, 28:377–399.
133. The Anatomy of a Search Engine
[http://infolab.stanford.edu/~backrub/google.html]
134. Lempel R, Moran S: The stochastic approach for link-structure analysis
(SALSA) and the TKC effect. Comput Networks 2000, 33:387–401.
135. Wolf DM, Arkin AP: Motifs, modules and games in bacteria. Curr Opin
Microbiol 2003, 6:125–134.
136. Hartwell LH, Hopfield JJ, Leibler S, Murray AW: From molecular to modular
cell biology. Nature 1999, 402(6761 Suppl):C47–52.
137. Ravasz E, Somera AL, Mongru DA, Oltvai ZN, Barabási AL: Hierarchical
organization of modularity in metabolic networks. Science 2002, 297:1551–5.
138. Thiele I, Palsson BØ: A protocol for generating a high-quality genome-scale
metabolic reconstruction. Nat Protoc 2010, 5:93–121.
139. Bar D: Evidence of massive horizontal gene transfer between humans and
Plasmodium vivax. core.ac.uk .
140. Altschul SF, Koonin E V.: Iterated profile searches with PSI-BLAST—a tool for
discovery in protein databases. Trends Biochem Sci 1998, 23:444–447.
141. Moreno-Hagelsieb G, Hudy-Yuffa B: Estimating overannotation across
prokaryotic genomes using BLAST+, UBLAST, LAST and BLAT. BMC Res Notes
2014, 7:651.
142. Gouzy J, Corpet F, Kahn D: Whole genome protein domain analysis using a
new method for domain clustering. Comput Chem 1999, 23:333–340.
143. Claudel-Renard C, Chevalet C, Faraut T, Kahn D: Enzyme-specific profiles for
genome annotation: PRIAM. Nucleic Acids Res 2003, 31:6633–6639.
144. Finn RD, Bateman A, Clements J, Coggill P, Eberhardt RY, Eddy SR, Heger A,
Hetherington K, Holm L, Mistry J, Sonnhammer ELL, Tate J, Punta M: Pfam: The
protein families database. Nucleic Acids Research 2014.
145. Mulder N, Apweiler R: InterPro and InterProScan: tools for protein sequence
classification and comparison. Methods Mol Biol 2007, 396:59–70.
146. Mitchell A, Chang H-Y, Daugherty L, Fraser M, Hunter S, Lopez R, McAnulla C,
McMenamin C, Nuka G, Pesseat S, Sangrador-Vegas A, Scheremetjew M, Rato C,
Yong S-Y, Bateman A, Punta M, Attwood TK, Sigrist CJA, Redaschi N, Rivoire C,
Xenarios I, Kahn D, Guyot D, Bork P, Letunic I, Gough J, Oates M, Haft D, Huang H,
Natale DA, et al.: The InterPro protein families database: the classification resource
after 15 years. Nucleic Acids Res 2014, 43(Database issue):D213–21.
147. Finn RD, Bateman A, Clements J, Coggill P, Eberhardt RY, Eddy SR, Heger A,
Hetherington K, Holm L, Mistry J, Sonnhammer ELL, Tate J, Punta M: Pfam: the](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-195-2048.jpg)











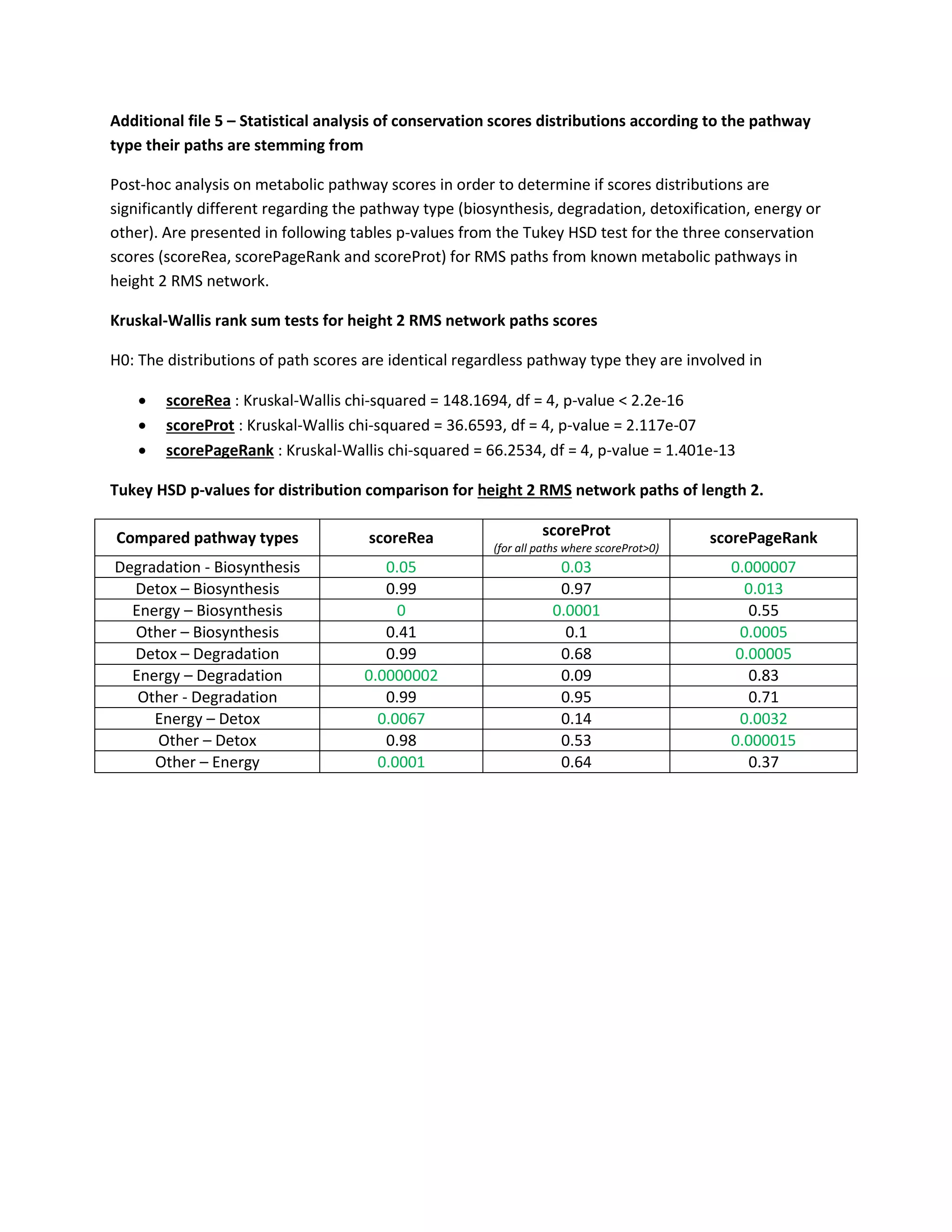





















![class BIOSYNTHESIS IF : scoreRea=0.7191949522280763 ^ scoreProtTaxo=55.61823899667411 ^ scorePageRankTopoDiv=1.3556204474833197E-5 (2)
class BIOSYNTHESIS IF : scoreRea=0.1336306209562122 ^ scoreProtTaxo=113.06193486507043 ^ scorePageRankTopoDiv=8.526134756584441E-5 (3)
class DEGRADATION IF : scoreRea=2.680951323690902 ^ scoreProtTaxo=0.011672391865901513 ^ scorePageRankTopoDiv=2.1883160997492087E-4 (4)
class BIOSYNTHESIS IF : scoreRea=1.0 ^ scoreProtTaxo=0.0 ^ scorePageRankTopoDiv=1.9055649310582596E-4 (2)
class BIOSYNTHESIS IF : scoreRea=1.0 ^ scoreProtTaxo=0.0 ^ scorePageRankTopoDiv=1.8860851388601528E-4 (1)
class DEGRADATION IF : 1.0<=scoreRea<=1.1881770515720091 ^ 4.7879252966052475<=scoreProtTaxo<=5.411851401620706 ^ 1.1442075717380128E-
4<=scorePageRankTopoDiv<=1.2181717835020451E-4 (4)
class ENERGY IF : scoreRea=3.4641016151377544 ^ scoreProtTaxo=16.560296819002364 ^ scorePageRankTopoDiv=2.120396524500385E-4 (3)
class DEGRADATION IF : scoreRea=1.0 ^ scoreProtTaxo=0.0 ^ scorePageRankTopoDiv=1.1328728240243136E-4 (4)
class ENERGY IF : scoreRea=1.0 ^ scoreProtTaxo=21.7568033167796 ^ scorePageRankTopoDiv=7.011720372293434E-4 (4)
class BIOSYNTHESIS IF : scoreRea=1.0 ^ scoreProtTaxo=0.0 ^ scorePageRankTopoDiv=3.3064912264776746E-4 (1)
class BIOSYNTHESIS IF : scoreRea=1.1114378604524227 ^ scoreProtTaxo=19.8045615400731 ^ scorePageRankTopoDiv=5.409945344885199E-5 (1)
class DEGRADATION IF : scoreRea=5.372405894758811 ^ scoreProtTaxo=64.86872458326154 ^ scorePageRankTopoDiv=2.5451465611213983E-4 (1)
class BIOSYNTHESIS IF : scoreRea=0.9274777915203366 ^ scoreProtTaxo=2.284447209188501 ^ scorePageRankTopoDiv=1.341109037928679E-4 (1)
class BIOSYNTHESIS IF : scoreRea=0.28867513459481287 ^ scoreProtTaxo=1.8435289494404254 ^ scorePageRankTopoDiv=5.057229096274693E-5 (1)
class DEGRADATION IF : scoreRea=7.874007874011811 ^ scoreProtTaxo=83.60493888793984 ^ scorePageRankTopoDiv=9.184702952142395E-5 (4)
class BIOSYNTHESIS IF : scoreRea=0.7071067811865476 ^ scoreProtTaxo=488.5222615194924 ^ scorePageRankTopoDiv=2.954382058651906E-4 (1)
class BIOSYNTHESIS IF : scoreRea=1.0 ^ scoreProtTaxo=99.94050773418287 ^ scorePageRankTopoDiv=5.84516351945006E-4 (5)
class OTHER IF : scoreRea=2.0 ^ scoreProtTaxo=0.0 ^ scorePageRankTopoDiv=4.088733957652656E-4 (2)
class OTHER IF : scoreRea=0.20965696734438366 ^ scoreProtTaxo=62.006127251263905 ^ scorePageRankTopoDiv=3.6903530145216174E-5 (1)
class BIOSYNTHESIS IF : scoreRea=1.0 ^ scoreProtTaxo=0.0 ^ scorePageRankTopoDiv=1.9202370889358065E-4 (1)
class BIOSYNTHESIS IF : scoreRea=2.8535691936340255 ^ scoreProtTaxo=0.0 ^ scorePageRankTopoDiv=3.201551503233059E-4 (2)
class BIOSYNTHESIS IF : scoreRea=1.0 ^ scoreProtTaxo=0.0 ^ scorePageRankTopoDiv=1.9202370889358065E-4 (1)
class DEGRADATION IF : scoreRea=1.4142135623730951 ^ scoreProtTaxo=1121.1691346845957 ^ scorePageRankTopoDiv=1.50642091339147E-4 (2)
class BIOSYNTHESIS IF : scoreRea=0.5 ^ scoreProtTaxo=0.0 ^ scorePageRankTopoDiv=4.787860914058206E-5 (3)
class OTHER IF : scoreRea=2.0 ^ scoreProtTaxo=57.65446242084278 ^ scorePageRankTopoDiv=4.287988675749166E-4 (3)
class DEGRADATION IF : scoreRea=0.7071067811865476 ^ scoreProtTaxo=273.33252297887157 ^ scorePageRankTopoDiv=4.474941279021902E-5 (1)
class OTHER IF : scoreRea=0.8498365855987975 ^ scoreProtTaxo=491.14396928120954 ^ scorePageRankTopoDiv=8.933471786716984E-5 (3)
Stat :
class DEGRADATION : 315 exemplar(s) including 288 Hyperrectangle(s) and 27 Single(s).
class BIOSYNTHESIS : 455 exemplar(s) including 385 Hyperrectangle(s) and 70 Single(s).
class OTHER : 92 exemplar(s) including 80 Hyperrectangle(s) and 12 Single(s).
class DETOX : 35 exemplar(s) including 31 Hyperrectangle(s) and 4 Single(s).
class ENERGY : 71 exemplar(s) including 64 Hyperrectangle(s) and 7 Single(s).
Total : 968 exemplars(s) including 848 Hyperrectangle(s) and 120 Single(s).
Feature weights : [0.026621704589354037 0.013098001491379322 0.03430947381803635]
Time taken to build model: 1.72 seconds
=== Stratified cross-validation ===
=== Summary ===
Correctly Classified Instances 7822 94.7432 %
Incorrectly Classified Instances 434 5.2568 %
Kappa statistic 0.9076
Mean absolute error 0.021
Root mean squared error 0.145
Relative absolute error 9.2047 %
Root relative squared error 42.9119 %
Total Number of Instances 8256
=== Detailed Accuracy By Class ===
TP Rate FP Rate Precision Recall F-Measure ROC Area Class
0.922 0.028 0.927 0.922 0.925 0.947 DEGRADATION
0.965 0.06 0.958 0.965 0.961 0.952 BIOSYNTHESIS
0.929 0.003 0.947 0.929 0.938 0.963 OTHER
0.869 0.001 0.926 0.869 0.897 0.934 DETOX
0.935 0.004 0.939 0.935 0.937 0.966 ENERGY
Weighted Avg. 0.947 0.043 0.947 0.947 0.947 0.952
=== Confusion Matrix ===](https://image.slidesharecdn.com/070b335d-896a-491b-bc5d-eba79c5ef7a3-160421120717/75/These_Maria_Sorokina-229-2048.jpg)



