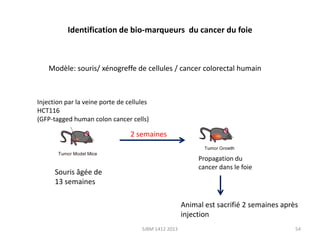
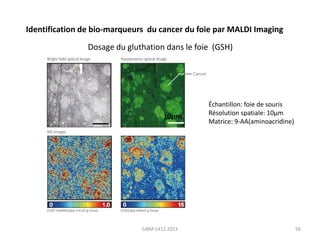
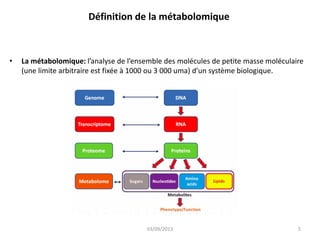
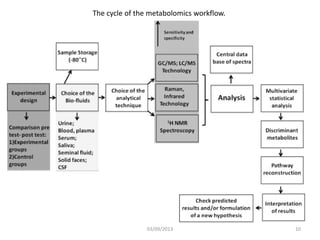
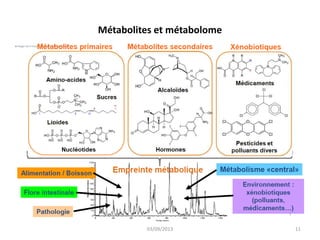
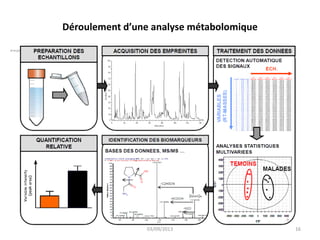
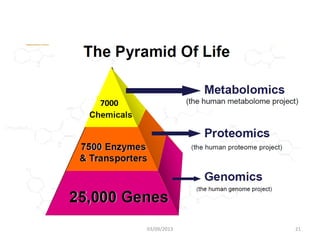
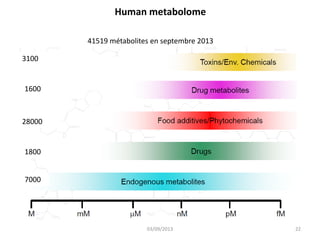
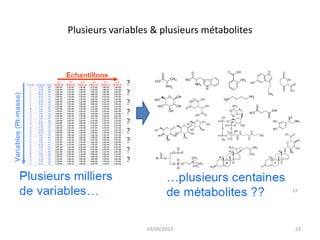
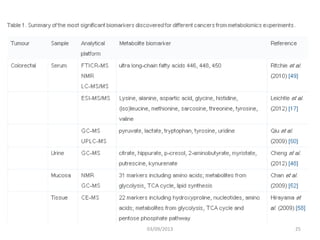
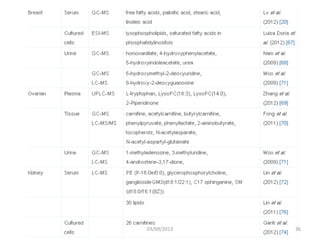
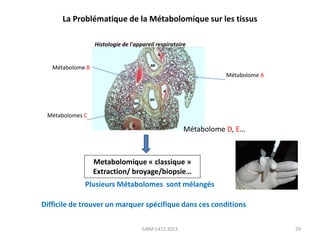
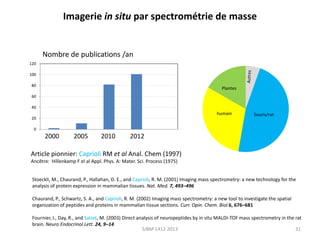
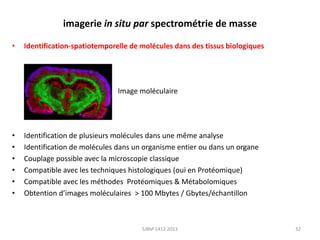
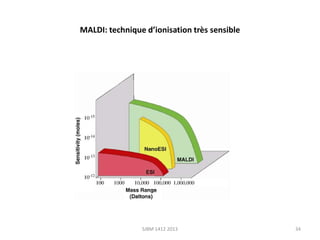
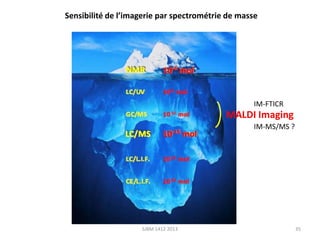
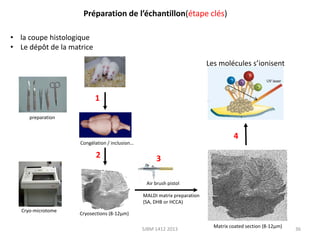
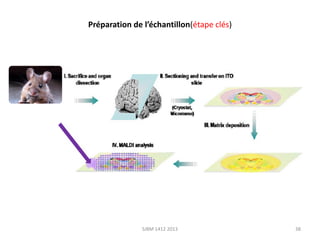
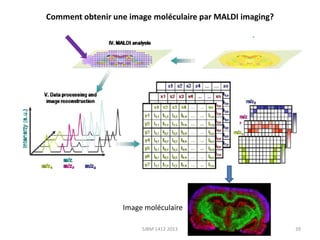
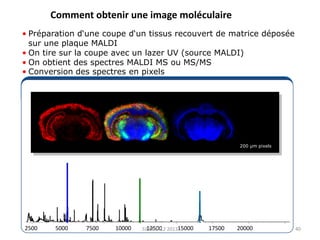
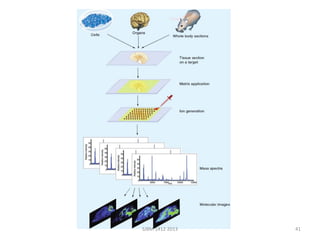
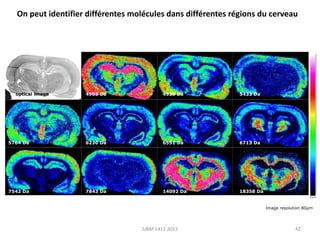
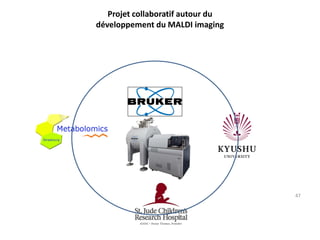
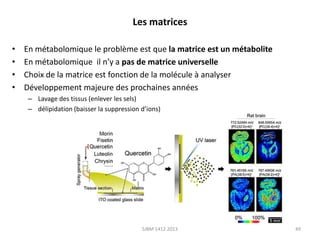
La métabolomique est l'analyse des petites molécules et des voies métaboliques au sein des cellules et des organismes, utilisant des techniques comme la chromatographie et la spectrométrie de masse. Les applications incluent le profilage métabolique en réponse à des pathologies, ainsi que l'imagerie in situ pour identifier des molécules dans des tissus. Le développement de bases de données et l'optimisation des méthodes d'analyse, telles que le MALDI imaging, sont essentiels pour améliorer la sensibilité et la précision dans le domaine.























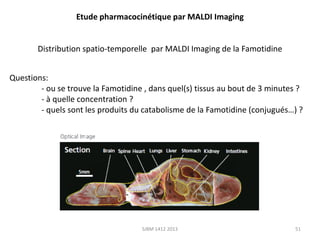
![Distribution spatio-temporelle par MALDI Imaging de la Famotidine chez la
souris
Famotidine
MW= 337.449 g/mol
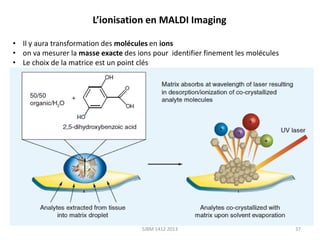
Les molécules s’ionisent
[ M+H+]
La coupe est recouverte de
matrice DHB
Résolution spatiale
basse: 200µm
Accumulation de la Famotidine dans les reins
SJBM 1412 2013
52](https://image.slidesharecdn.com/sjbm14122013dheintz-140214100555-phpapp01/85/Metabolomique-Congres-SJBM-2013-Marseille-52-320.jpg)
![Distribution spatio-temporelle par MALDI Imaging de la Famotidine chez la
souris
Accumulation de produits du catabolisme de la Famotidine dans les reins
[ M+H+]
+/Na
338.05> 259.06
Famotidine
SJBM 1412 2013
+/OH
53](https://image.slidesharecdn.com/sjbm14122013dheintz-140214100555-phpapp01/85/Metabolomique-Congres-SJBM-2013-Marseille-53-320.jpg)