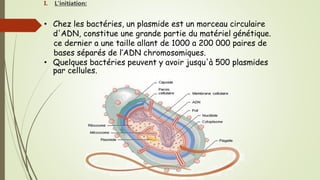
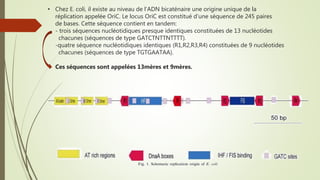
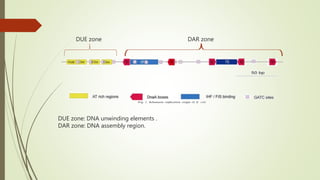
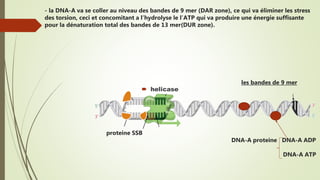
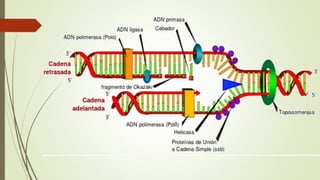
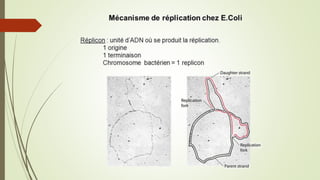
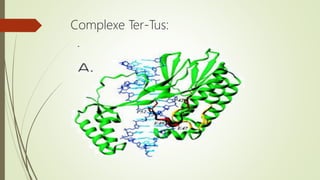
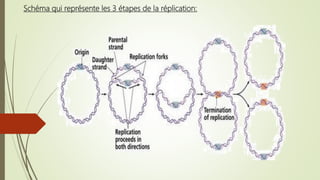
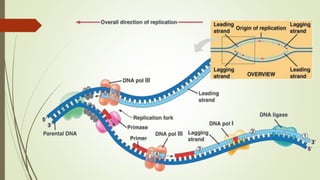
Le document traite des étapes de la réplication de l'ADN chez les bactéries, en se concentrant sur l'initiation, l'élongation et la terminaison. Il décrit le rôle des divers éléments protéiques et séquences nucléotidiques impliqués dans ces processus, y compris le site d'oriC chez E. coli. La terminaison du processus se produit lorsque les fourches de réplication se rencontrent et est régulée par des séquences spécifiques et des protéines comme Tus.